0 - таблица
| PDB код | Тип
(спираль, баррель) |
Какая мембрана (внутренняя или внешняя, организм, органелла) |
Толщина гидрофобной части мембраны в ангстремах | Медиана числа остатков в одном трансмембранном участке |
| 1af6 | баррель | E. coli внешняя мембрана(а какая же еще) | 25.1 ± 0.7 | 188/18 ( 10.(4) ) |
| 2jmm | баррель | designed protein | 23.9 ± 1.3 | 77/8 ( 9.625 ) |
| 2jk4 | баррель | Homo sapiens внешняя митохондриальная мембрана | 23.4 ± 2.3 | 150/19 ( ~7.895 ) |
| 3dzm | баррель | Thermus thermophilus бактериальная мембрана | 28.5 ± 2.6 | 87/8 ( 10.875 ) |
| 4bw5 | спираль | Homo sapiens клеточная мембрана | 31.2 ± 0.9 | 107/4 ( 26.75 ) |
| 4daj | спираль | Rattus norvegicus клеточная мембрана | 32.2 ± 1.2 | 166/7 ( ~23.714 ) |
| 1py6 | спираль | Halobacterium salinarum архебактериальная мембрана | 29.6 ± 2.2 | 146/7 ( ~20.857 ) |
1 - Отбор гомологов
Для того чтобы взять 13 репрезентативных гомологов я провел blastp моего белка(PDB ID: 4HFI) на сайте uniprot(знаю, что на NCBI больше возмоностей, но этот новый дизайн на UniProt моя слабость, плюс, возможность отметить нужные последовательности и сразу же их достать функцией retrieve, очень удобно). Поиск провел по swissprot и так как БД не очень большая, брал гомологи с e-value меньше 10e-15 плюс, используя NCBI taxonomy и свои знания о том, какие организмы относятся к каким таксонам выбрал белки как можно более удаленных друг от друга организмов(что подтвердилось рано ветвящимся деревом, построенным по последовательностям). У выбранных белков большие записи в swissprot со статьями, что говорит о том, что эти белки хорошо изучены.После редактирования названий так, чтобы остались только идентификаторы белков и названия организмов, я получил fasta файл с невыровненными последовательностями Кстати, белки найдены либо у Metazoa либо у Cyanobacteria либо у Proteobacteria, в случае бактерий дальше филума сходство таксономии не идет, у животных - у всех разные классы.
2 - анализ структуры белка
Ни в каких специализированных на трансмембранных белках БД моего белка не оказалось, так что пришлось качать файл с PDB и пользоваться сервисом PPM в OPM.Описание структуры трансмембранного белка Q7NDN8 (идентификатор PDB 4HFI, цепь A)
| PDB ID | Организм | тип мембраны | TC-код | Угол наклона спиралей | Количество трансмембранных спиралей |
| 4HFI | Gloeobacter violaceus | бактериальная мембрана | 1.A.9.8.1(ни один ТС код структуры с белком не связан, этот был найден через swissprot) | 0.0 ± 1.0 Å | 5 |
На сайте tcdb.org можно узнать всю расшифровку TC кода
Расшифровка TC кода
1.A. - каналы α типа1.A.9. - Нейротрансмиттерные рецепторы, семейство LIC (Ligand-gated Ion Channel)
1.A.9.8. - ???
1.A.9.8.1 - Прокариотический ионный канал, закрытый для протонов
3 - анализ множественного выравнивания трансмембранных белков
в JalView программой muscle было построено выравнивание отобранных в 1 задании белковпрограмма TMHMM
Программа TMHMM предсказала трансмембранные участки для человеческого белка P47870-4 (во-первых он таксономически очень далек от изучаемого белка и во-вторых - да, я антропоцентрист), трансмембранные участки подчеркнуты:WEBSEQUENCE TMHMM2.0 outside 1 243
WEBSEQUENCE TMHMM2.0 TMhelix 244 266
WEBSEQUENCE TMHMM2.0 inside 267 272
WEBSEQUENCE TMHMM2.0 TMhelix 273 292
WEBSEQUENCE TMHMM2.0 outside 293 306
WEBSEQUENCE TMHMM2.0 TMhelix 307 329
WEBSEQUENCE TMHMM2.0 inside 330 372
анализ структуры в JalView
Были добавлены 2 строки аннотации, также остатки были раскрашены по гидрофобности (а также by conservation, treshold = 20).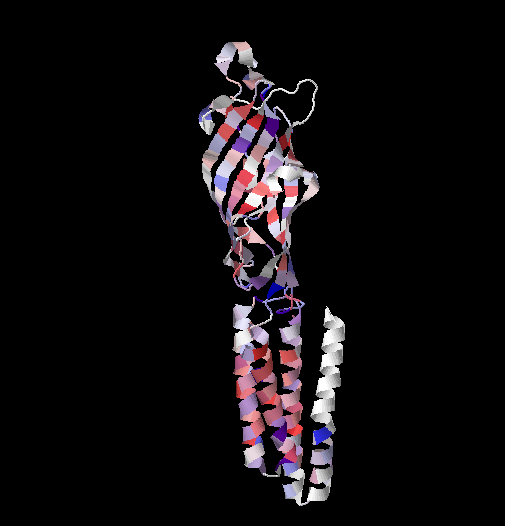
структура 1HFI белка Q7NDN8, раскрашенная по гидрофобности и консервативности
Описание изображения
0 - белок состоит из 5 одинаковых субъединиц, показана одна из них на изображении - n сторона вверху, p сторона внизу, внутренняя часть белка(канал) - слева, внешняя - справа
1 - консервативны 3 из 4 трансмемранных спиралей, но та, что не консервативна, не сообщается с самим каналом. В спиралях довольно много Leu и Ile, причем в самых консервативных позициях
2 - участки между спиралями довольно консервативны почти на всем своем протяжении, хотя и не так, как трансмембранные участки спиралей, но гораздо консервативней, чем немембранные участки спиралей
3 - да, небольшое количество гидрофильных(синий) участков есть в трансмембранных спиралях, причем, некоторые крайне консервативны, что говорит о функциональной значимости, другие не столь консервативны, но в их наличии нет ничего удивительного, если вспомнить, что этот белок - ионный канал.
качество работы TMHMM
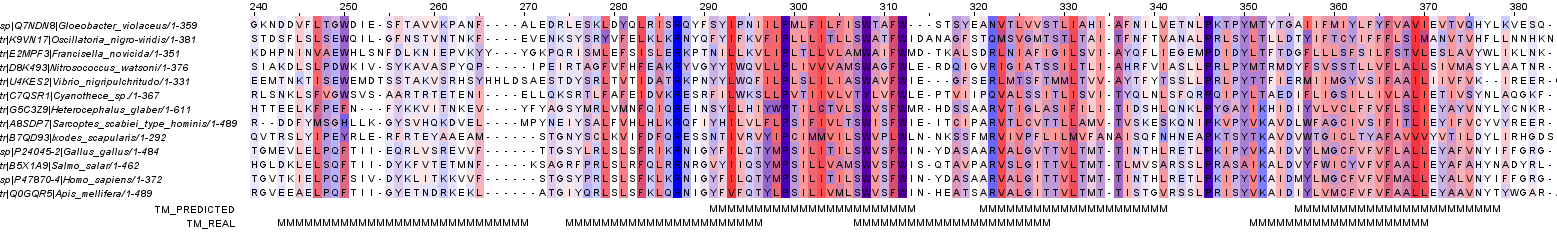
фрагмент выравнивания, содержащий все трансмембранные участки, как описанные в PDB файле, так и предсказанные TMHMM