9 Практикум
1 - Выбор домена и архитектур
Домен
AC - PF02171ID - Piwi
Белки, содержащие домен Piwi (P-element induced wimpy proteins) это регуляторные белки, ответственные за осуществление неполной дифференциации стволовых клеток и стабильности скорости деления клеток зародышевого пути.
ссылка на страницу домена
Архитектуры
В PFam домен содержат 30 архитектур, 2067 последовательностейссылка на страницу со списком архитектур
Часто в архитектурах домен Piwi встречается с доменом PAZ
Выравнивание
В JalView было получено выравнивание всех белков, содержащих домен, также на PDB была найдена структура белка с PDB ID: 1W9H белка PIWI_ARCFU, которую я связал с выравниванием2 - Архитектуры, включающие домен
Я выбрал две простые, но распространенные архитектуры.1 - включающая только домен Piwi, такую архитектуру имеют 543 последовательности

2 - включающая домены Paz и Piwi, такую архитектуру имеют 532 последовательности

Домен PAZ(Piwi Argonaut and Zwille) найден в белках с доменами семейств Piwi и Dicer, функция неизвестна, но, возможно, он помогает гетеродимеризоваться белкам с указанными доменами.
сводная таблица
С помощью скрипта swisspfam-to-xls.py была получена таблица, результаты которой (айдишники белков) были использованы для получения последовательностей для работы скрипта uniprot-to-taxonomy.py В результате я получил сводную таблицу с информацией о белках, содержащих домен Piwi.Выбор таксона
В качестве исследуемого таксона я выбрал всех живых организмов, а субтаксоны - соответственно - Bacteria, Archaea и EucaryotaВыбор белков
Во втором листе таблицы, упоминавшейся выше, я отобрал по 20 представителей 2х выбранных мной доменных архитектур,(позже я удалил "нехорошие" белки в каждом выравнивании, так что представителей оказалось по 19)Выравнивание архитектур
С помощью еще одного скрипта я взял из выравнивания всех доменов Piwi, выбранные мной, удалил пару совсем невыровненных последовательностей, обрезал N и С концы, из которых "торчало" по одной-две последовательности, убрал пустые колонки, сгруппировал и раскрасил оставшиеся белки.проект JalView
10 практикум
Построение филогенетического древа
Названия всех доменов белков были отредактированы и имеют вид X_Y_ZZZZZZ, гдеX это порядковый номер доменной архитектуры, принимает значения 1 -для архитектуры, содержащей только домен Piwi, и 2 для архитектуры, содержащей домены PAZ и Piwi
Y это обозначение таксона, принимает значения E для эукариот, A для архей и B для бактерий
ZZZZZZ это ID белка без указания организма.
В программе MEGA методом minimal-evolution(далее ME) было построено дерево, к которому был применен бутстреп анализ 100 деревьев. Такое же дерево было построено методом maximal likelihood(далее ML).
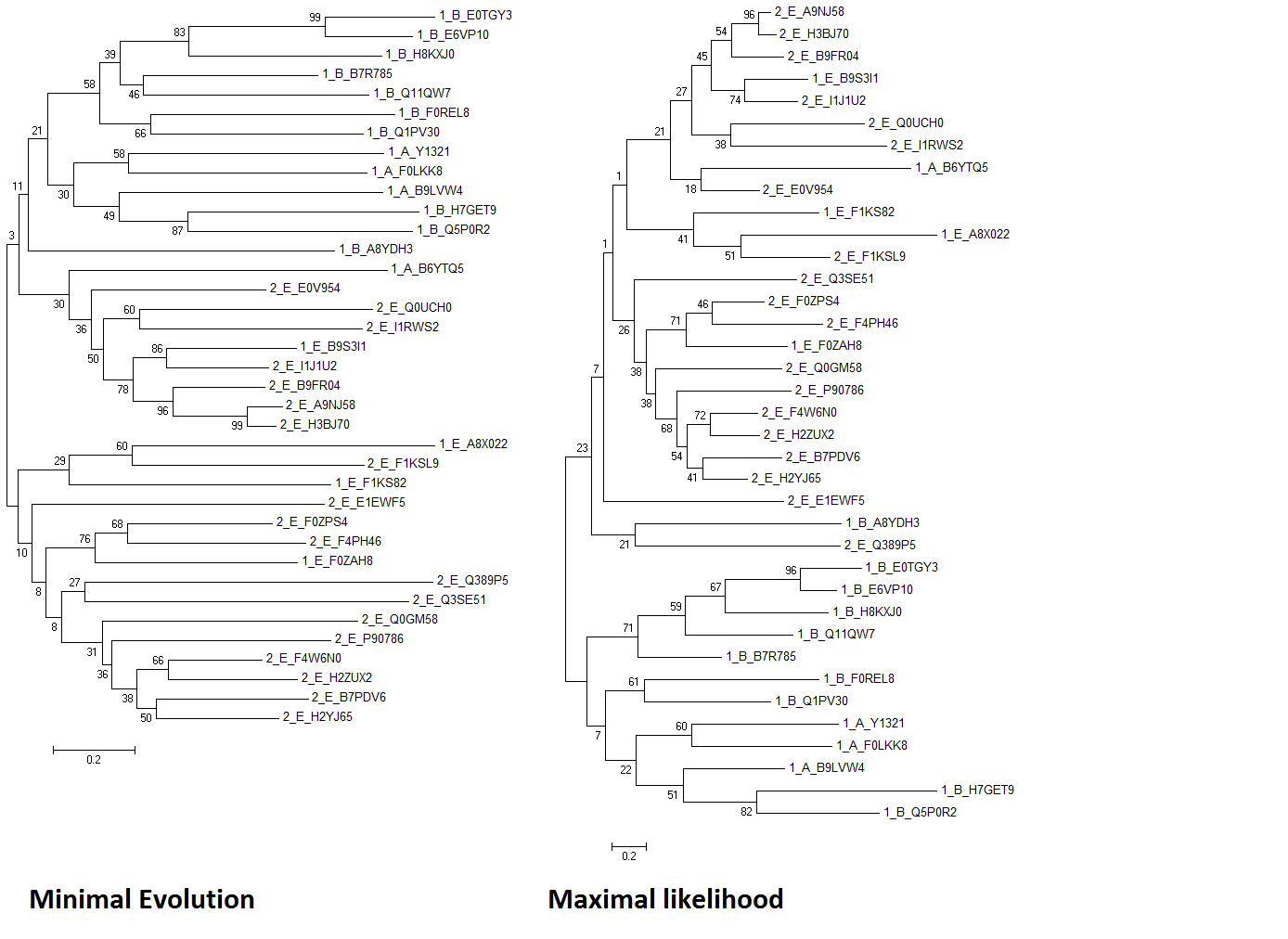
скобочная формула ME скобочная формула ML
Деревья получились откровенно плохими, что видно по результатам бутстрепа. Все клады около корня получили плохую оценку, некоторые не дотягивают даже до 10. в одной и той же кладе встречаются домены из разных архитектур или таксонов, и только небольшие клады (не более 7 листьев) получили оценку выше 50. Метод neighbor-joiningдал такое же дерево, как и ME, так что его для сравнения я использовать не буду. Обращать внимание я буду только на клады, получившие более чем 40% поддержку с помощью бутстрепа, поскольку, как я сказал, прикорневые ноды, имеющие малую поддержку реконструированы из рук вон плохо. Это можно объяснить плохим выравниванием, тем, что я безграмотно "подправил" выравнивания или тем, что уже у луки было несколько белков с доменной архитектурой 1.(архитектура 2 обнаружилась только у эукариот) Я буду исходить из последнего предположения, просто, потому что первые два не могут стать почвой ни для каких гипотез. Итак, посмотрим на хорошие клады. Довольно уверенно найдена клада для 7 бактериальных белков.
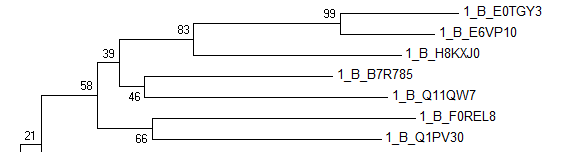
построена методом ME
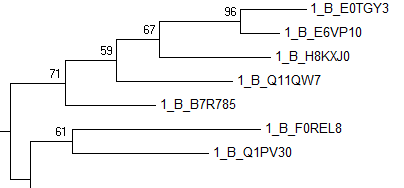
построена методом ML
Можно сказать, что эта бактериальная клада хорошо находится обоими методами с почти идентичной топологией ветвей. Также она довольно хорошо обособлена от других клад в реконструкции ME, но в реконструкции ML от нее отхтодит клада с архейскими и другими бактериальными белками, так что возможно, хотя это и трудно сказать, эти белки имели непосредственного предшественника в геноме луки. Для лучшего понимания, конечно, нужно построить дерево существенно большего размера.
Также хорошо обособлена клада с тремя архейскими и двумя бактериальными белками.

построена методом ME
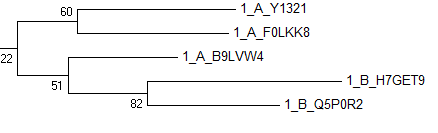
построена методом ML
Возможно, наряду с первой рассмотренной группой эта группа имела своего предшественника у луки, поскольку "отпочковывание" бактериальной клады от архей вполне убедительно.