Таблица 1. Результаты поиска гипотетических гомологов белка PTHP_BACSU
| Поиск по Swiss-Prot | Поиск по PDB | Поиск по "nr" | |
1. Лучшая находка (с последовательностью исходного белка) |
|||
| Accession | P08877.3 | 2FEP_S | NP_389273 |
| E-value | 2e-57 | 1e-57 | 1e-55 |
| Вес (в битах) | 177 | 175 | 177 |
| Процент идентичности | 100% | 99% | 100% |
2. Число находок с E-value < 10–10 |
28 | 29 | 438 |
3. "Худшая из удовлетворительных" находка (последняя в выдаче с E-value < 1) |
|||
| Номер находки в списке описаний | 55 | 33 | 1000 |
| Accession | Q8FFD8.1 | 3CCD_A | ZP_08148112 |
| E-value | 0.027 | 2e-11 | 4e-08 |
| Вес (в битах) | 35.8 | 56.6 | 61.6 |
| % идентичности | 30 | 33 | 39 |
| % сходства | 54 | 59 | 60 |
| Длина выравнивания | 74 | 81 | 77 |
| Координаты выравнивания (от-до, в запросе и в находке) | 12-82; 12-84 | 1-81; 1-81 | 1-77;1-77 |
| Число гэпов | 4 | 0 | 0 |
Исходный белок удалось найти в Swiss-Prot и "nr", а его структуру в PDB
Число явных гомологов (E-value < 1e-10) сильно отличается от остальных в банке
Non-redundant protein sequences(nr), что прежде всего обусловлено тем, что
этот банк - "виртуальный" и включает в себя все белковые последовательности из
всевозможных источников (в первую очередь Swiss-Prot и аннотации кодирующих
участков генов в GenBank), поэтому и подобных находок в нем значительно больше,
чем в банках Swiss-Prot и PDB (Protein Data Bank proteins)
Всего находок 1000. E-value самой последней находки: 4e-08. Число находок
лимитировано значением Max target sequences - предельным размером выдачи
Поиск гипотетических гомологов изучаемого белка с фильтром по таксонам
При исследовании проверялись следующие таксоны:
- Eukaryota (другое царство)
Гипотетического гомолога с критерием: E-value<0,001 не было найдено,
лучшая находка имела E-value=5.8
- Actinobacteria (другой отдел того же царства бактерий);
- Clostridia (другой класс того же отдела Firmicutes)
Гипотетического гомолога с критерием, удовлетворяющим условию не было
найдено, лучшая находка имела E-value=3.5
- Lactobacillales (другой порядок того же класса Bacilli)
- Listeriaceae (другое семейство того же порядка Bacillales)
- Geobacillus (другой род того же семейства Bacillaceae)
- Bacillus anthracis (другой вид того же рода)
Гипотетического гомолога с критерием, удовлетворяющим условию не было
найдено, лучшая находка имела E-value=1.7
Результаты поиска гипотетических гомологов
| Таксон | Номер находки в списке описаний |
Accession | E-value | Вес (в битах) |
% идентичности | % сходства | Длина выравнивания |
Координаты выравнивания (от-до, в запросе и в находке) |
Число гэпов |
| Actinobacteria (taxid:201174) | 1 | O50515 | 8e-07 | 47.4 | 35 | 54 | 91 | 1-88 1-88 |
6 |
| Lactobacillales (taxid:186826) | 1 | O07125 | 2e-34 | 138 | 73 | 89 | 88 | 1-88 1-88 |
0 |
| Listeriaceae (taxid:186820) | 1 | P0A438 | 8e-29 | 117 | 64 | 76 | 88 | 1-88 1-88 |
0 |
| Geobacillus (taxid:129337) | 1 | P42013 | 6e-29 | 117 | 70 | 85 | 88 | 1-88 1-88 |
0 |
При поиске гипотетического гомолога главным критерием было значение E-value<0,001. В результате лучшая находка найдена по таксону Lactobacillales (другой порядок того же класса Bacilli) с E-value=2e-34 в порядке приближения к изучаемому белку Bacillus subtilis(E-value=2e-44).
Использование BLAST для получения карты локального сходства двух последовательностей
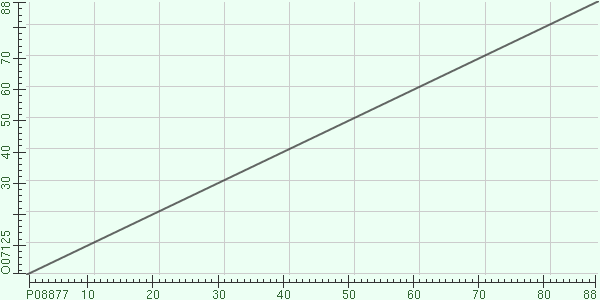
В параметрах BLAST можно подать на ввод более чем одну последовательность ( галочка у Align two or more sequences и появится новое окошко для ввода).Если теперь запустить алгоритм, то мы получим парное выравнивание данных последовательностей и его параметры. Кроме того, BLAST автоматически построит карту локального сходства (рис.1 и рис.2) этих двух последовательностей (Dot Matrix View) - т.е. графическое изображения выравнивания в виде таблицы, на сторонах которой расположены последовательности, и отмечены только те ячейки, которые образованы пересечением строк и столбцов с одинаковыми символами (в нашем случае аминокислотами).
Для BLAST с более чем одной последовательностью также можно варьировать значением e-value: первая карта построена при значении по умолчанию (10), а вторая - при e-value=0.01. Для примера (рис 1., рис 2.) была получена карта локального сходства исходной последовательности (P08877) и последовательности, найденной во втором задании (O07125).
| Рис 1.Карта локального сходства последовательностей с е-value=10 | Рис 2.Карта локального сходства при e-value=0.01 |
 |
 |
Как видно из графиков, значения параметра е-value в данном случае никак не повлияло на карту локального сходства.
© Boskhomdzhieva Baina, 2012