Паттерны и банк ProSite
Задание 1
Теоретически слово awake может встретиться в банке SwissProt 52 раза.
Для вычисления необходимо знать частоты встречаемости аминокислотных остатков соответствующих буквам заданного слова ( A = 8.25,W = 1.08,A = 8.25, K = 5.84 , E = 6.75,). Вероятность нахождения слова awake равна 0.0825*0.0108*0.0825*0.0584*0.0675 = 2.7e-7, умножая на общее количество слов (191670831) получаем искомую величину, равную 51,75.
Вероятность того, что это слово встретится в базе данных равна 2.7e-7.
В ответ на запрос a-w-a-k-e Prosite дал ответ «The search term has not been found in PROSITE entries.»- Создание паттернов по множественному выравниванию
и поиск по паттернам в банке данных Swiss-Prot
Фрагмент множественного выравнивания: 13-25 а.о.
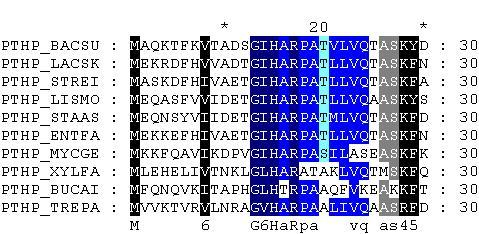
Столбцы, консервативные на 100%, выделены темно-синим цветом, соответственно на 80% - просто синим, а на 70% - бирюзовым
Таблица 1. Результаты исследования
Характеристика паттерна Паттерн В скольких последовательностях банка Swiss-Prot найден мотив, удовлетворяющий паттерну? Все ли последовательности из Вашего выравнивания найдены? Фрагмент последовательности G-I-H-A-R-P-A-T-V-L-V-Q-T в одном только мой белок Сильный G-[ILV]-H-[AT]-R-[PA]-[AT]-[TSA]- [VLMIKQ]-[LFI]-[VA]-[QSK]-x(1) в 36-ти да, все Слабый [AT]-R-[PA]-[AT]-x(0,1)-X(0,1)-x(0,1)-[VA]-[QSK]-X(1) в 1000-ти да, все В результате создания паттернов фрагмента выравнивания моего белка и его гомологов был осуществлен поиск мотивов, удовлетворяющих паттернам, с помощью банка PROSITE и по мере ослабления паттерна число таких мотивов соответственно увеличивалось и находились все последовательности моего выравнивания, а помимо их еще и большое количество последовательностей с названиями, явно отличающимися от названия моего белка. Кроме того при поиске мотива по слабому паттерну не все последовательности были показаны, так как был достигнут лимит =1000, из 1010 найденных.
- Поиск и описание всех мотивов в моем белке P08877(по данным БД PROSITE)
Таблица2: Результаты поиска мотивов
Идентификатор документа PROSITE (AC) Название мотива Краткое описание мотива Тип подписи (паттерн, профиль) Паттерн (регулярное выражение) Специфична ли подпись? Сколько мотивов нашлось в белке? PS00369 PTS_HPR_HIS Сайт домена PTS HPR гистидин-фосфорилирования паттерн G-[LIVM]-H-[STAV]-R-[PAS]-[GSTA]-[STAMVN] специфична 1 PS00589 PTS_HPR_SER Сайт домена PTS HPR серин-фосфорилирования паттерн [GSTADE]-[KREQSTIV]-x-{EPRK}-{VPGL}-x-[KRDN]- S-[LIVMF](2)-{EVPL}-[LIVM]-{EATN}-x-[LIVM]-[GADE] специфична 1 PS00012 PHOSPHOPANTETHEINE Сайт фосфопантетеин-связывания паттерн [DEQGSTALMKRH]-[LIVMFYSTAC]-[GNQ]-[LIVMFYAG]-[DNEKHS]- S-[LIVMST]-{PCFY}-[STAGCPQLIVMF]-[LIVMATN]-[DENQGTAKRHLM]- [LIVMWSTA]-[LIVGSTACR]-{LPIY}-{VY}-[LIVMFA] специфична 1 PS00005 PKC_PHOSPHO_SITE Сайт киназыС-фосфорилирования паттерн [ST]-x-[RK] неспецифична 2 PS00006 CK2_PHOSPHO_SITE Сайт казеин-киназы2-фосфорилирования паттерн [ST]-x(2)-[DE] неспецифична 2 PS00008 MYRISTYL Сайт N-миристоилирования паттерн G-{EDRKHPFYW}-x(2)-[STAGCN]-{P} неспецифична 3
© Boskhomdzhieva Baina, 2012