Cхема из Pfam:
|
||||||
| Пояснения к схеме |
||||||
| № | Pfam AC | Pfam ID | Полное название семейства доменов (с кратким пояснением) |
Положение в последовательности белка PTHP_BACSU | Клан | Interactions(Взаимодействия) |
| 1 | PF00381 | PTS-HPr | Семейство доменов названо по названию цитоплазматического фермента, компонента фосфотрансферазной системы, где источником энергии является фосфоенолпируват, в результате которой бактерия поглощает сахара | 1–84 | Нет описания клана | их 12:Peripla_BP_1(2) Hpr_kinase_C(2) PTS-HPr(2) PEP-utilisers_N(2) PEP-utilizers(2) PTS_EIIA_1(2) |
2. Информация о домене PF00381 моего белка
- Во сколько разных архитектур входит домен?
в 13 разных архитектур - Для какого числа белков, содержащих домен, известна последовательность?
для 3344 белков - Для какого числа разных белков, содержащих домен,
определена пространственная структура (домена или всего белка)?
для 10 белков - Выравнивание seed фрагментов белков
3. Описание доменных архитектур, в которых присутствует два или более разных доменов
|
Таксон
|
Количество белков с доменом PF00359
|
|
| Эукариоты | Зеленые растения | 1 |
| Грибы | 0 | |
| Животные | 13 | |
| Остальные эукариоты | 0 | |
| Археи | 3 | |
| Бактерии | 1476+1(отмечена отдельно) | |
| Вирусы | 0 | |
Домен PF00359 в большей степени распространен в бактериях, в животных он представлен в 13-ти видах, из которых 6 относятся к типу Хордовых(Chordata), а остальные к Членистоногим(Arthropoda), Нематодам(Nematoda) и т.д. К остальным эукариотам принадлежит один вид - Бактерия S5. В вирусах и грибах он не был найден.
|
Таксон
|
Количество белков с доменом PF00381
|
|
| Эукариоты | Зеленые растения | 3 |
| Грибы | 1 | |
| Животные | 0 | |
| Остальные эукариоты | 0 | |
| Археи | 6 | |
| Бактерии | 1590+1(отмечена отдельно) | |
| Вирусы | 0 | |
Домен PF00381 больше всего распространен среди бактерий, но не представлен среди животных и вирусов. Следует отметить, что bacterium S5 (грам-отрицательная бактерия, уменьшающая содержание солей селена) выделяют отдельно, а не в числе других бактерий.
4. Cравнение описания мотивов в разных банках семейств, по данным InterPro
InterPro- это интегрированная база данных, собранная по данным из Pfam, Prosite о семействах белков, доменах, мотивах , сайтах и т.п
Для белка PTHP_BACSU была получена карта всех мотивов, сайтов и доменов, описанных в InterPro (см. рис 1.)
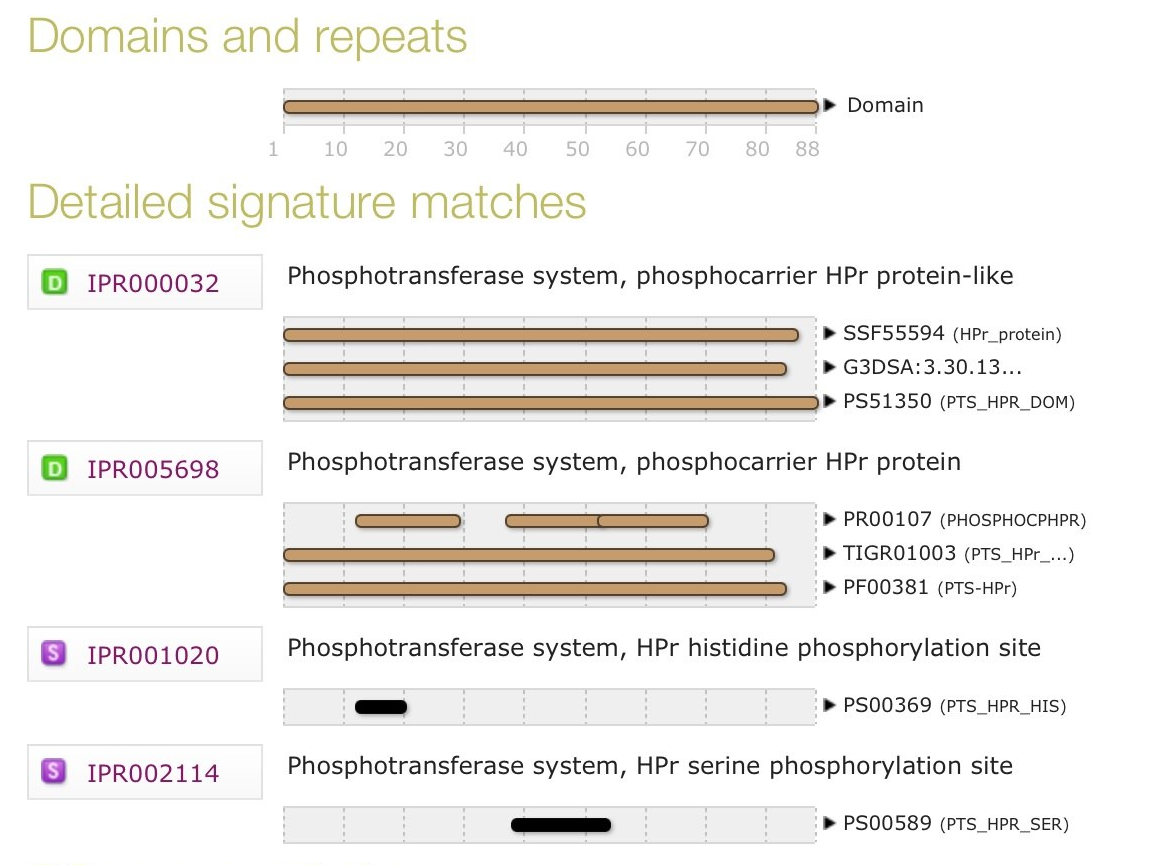
Рис. 1 Все подписи к последовательности PTHP_BACSU, интегрированные в InterPro
Ответы на некоторые вопросы по подписям InterPro:
Наиболее короткий описанный мотив PTHP_BACSU ( на рисунке показан черным) охватывает остатки с 13 по 20 и соответствует активному сайту фермента. Описан в банке PROSITE patterns (PS00369).
Наиболее длинный описанный мотив PTS_HPR_DOM охватывает всю последовательность и описывает ее как единый домен.Описан в банке PROSITE patterns (PS51350).
В InterPro интегрированы известные структуры белков для разных банков. Так, для PTHP_BACSU приведены идентичные структурные черты (Structural features): из SUPERFAMILY, CATH, PROSITE. Они представляют собой 85-ти, 83-х, 88-ми аминокислотные домены соответственно.Однако, PFAM предлагал нам 83 аминокислотный, неполный (обрезанный с краю) домен. Возможно, это связано с тем, что последние аминокислоты наиболее вариабельны для этой группы белков. Поэтому, при составлении множественного выравнивания и выделения эволюционных доменов PFAM искуственно укоротил структурный домен
© Boskhomdzhieva Baina, 2012