Программа getorf пакета EMBOSS
С помощью команды
entret embl:D89965получили файл с записью, содержащей последовательность мРНК Rattus norvegicus
Выполнив команду
getorf d89965.entret -minsize 90 -table 0 -find 1получили набор всех рамок считывания, где:
-minsize 30 -минимальная длину рамки -90 нуклеотидов
-table 0 -стандартный генетический код
-find 1 -нужный тип поиска( начиная со старт- и заканчивая стоп-кодоном)
В итоге получаем 5 рамок различной длины в транслированном виде.Заметим, что 3 последовательность совпадает с приведенной в записи EMBL для белка.
>D89965_3 [163 - 432] Rattus norvegicus mRNA for RSS, complete cds. MALMHFQFTFKQFEQRKSIRSTARKARDDFVVVQTADLFHVAFHYGIAQRGLTITSDDHM AVTAYAYYSCHELTPWLRIQSTNPVQKYGAЗапись EMBL d89965.entret ссылается на запись P0A7B8 Swiss-Prot. Используя команду
FT CDS 163..435 FT /product="RSS" FT /note="Rat Stomach Serotonin receptor-related gene" FT /db_xref="GOA:P0A7B8" FT /db_xref="InterPro:IPR001353" FT /db_xref="InterPro:IPR022281" FT /db_xref="PDB:1E94" FT /db_xref="PDB:1G4A" FT /db_xref="PDB:1G4B" FT /db_xref="PDB:1HQY" FT /db_xref="PDB:1HT1" FT /db_xref="PDB:1HT2" FT /db_xref="PDB:1NED" FT /db_xref="PDB:4G4E" FT /db_xref="UniProtKB/Swiss-Prot:P0A7B8" FT /protein_id="BAA14040.1" FT /translation="MALMHFQFTFKQFEQRKSIRSTARKARDDFVVVQTADLFHVAFHY FT GIAQRGLTITSDDHMAVTAYAYYSCHELTPWLRIQSTNPVQKYGA"
seqret sw:P0A7B8получили файл hslv_ecoli.fasta с последовательностью записи Swiss-Prot.
Определили какой рамке соответствует данная последовательность:
blastp -query hslv_ecoli.fasta -subject d89965.orf -out hslv.blastp
Subject= D89965_5 [294 - 1] (REVERSE SENSE) Rattus norvegicus mRNA for RSS,
complete cds.
Length=98
Score = 200 bits (509), Expect = 4e-71, Method: Compositional matrix adjust.
Identities = 98/98 (100%), Positives = 98/98 (100%), Gaps = 0/98 (0%)
Query 28 MKGNVKKVRRLYNDKVIAGFAGGTADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDR 87
MKGNVKKVRRLYNDKVIAGFAGGTADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDR
Sbjct 1 MKGNVKKVRRLYNDKVIAGFAGGTADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDR 60
Query 88 MLRKLEALLAVADETASLIITGNGDVVQPENDLIAIGS 125
MLRKLEALLAVADETASLIITGNGDVVQPENDLIAIGS
Sbjct 61 MLRKLEALLAVADETASLIITGNGDVVQPENDLIAIGS 98
>D89965_5 [294 - 1] (REVERSE SENSE) Rattus norvegicus mRNA for RSS, complete cds.
MKGNVKKVRRLYNDKVIAGFAGGTADAFTLFELFERKLEMHQGHLVKAAVELAKDWRTDR
MLRKLEALLAVADETASLIITGNGDVVQPENDLIAIGS
>HSLV_ECOLI P0A7B8 ATP-dependent protease subunit HslV (3.4.25.2) (Heat shock protein HslV)
MTTIVSVRRNGHVVIAGDGQATLGNTVMKGNVKKVRRLYNDKVIAGFAGGTADAFTLFEL
FERKLEMHQGHLVKAAVELAKDWRTDRMLRKLEALLAVADETASLIITGNGDVVQPENDL
IAIGSGGPYAQAAARALLENTELSAREIAEKALDIAGDICIYTNHFHTIEELSYKA
Это пятая рамка.
Как можно объяснить наблюдаемую ситуацию?
Возможно, это связано с тем, что исследовали искали белок в желудке крысы с определенными параметрами. После того, как они заметили активность белка со свойствами, похожими на свойства искомого,ученые выделили и секвенировали мРНК.Однако до этого,последовательность подобного белка уже была известна и хранилась в Swiss-Prot(принадлежала бактерии E.coli). Тем самым получается, что белок, полученный из желудка крысы на самом деле принадлежал космополиту E.coli, а значит авторы проаннотировали найденный белок неверно.
Файлы-списки
Командой seqret sw:adh*_*получила все последовательности алкогольдегидрогеназ из Swiss-prot.
Далее командой infoseq adh2_soltu.fasta -only -usa >listfile.txt был получен файл с универсальными адресами USA этих последовательностей.
Из этого списка-файла был получен другой, меньший, с адресами последовательностей, взятых из группы организмов( файл list.txt)
grep -f list.txt listfile.txt >listfile2.txt
На основе нового файла-списка (listfile2.txt) был получен fasta-файл listfile.fasta с последовательностями дегидрогеназ организмов
seqret @listfile2.txt listfile.fasta
EnsEMBL
EnsEMBL- геномный браузер, позволяющий визуализировать информацию о геномах человека и животных.
 |
Рис. 1 Расположение участка с искомым геномом в 5 хромосоме |
 |
Рис. 2 Схема расположения экзонов и интронов в гене по таким данным, как GENCODE, RefSeq/ENA, CCDS set (проект по выявлению кодирующих участков генома) |
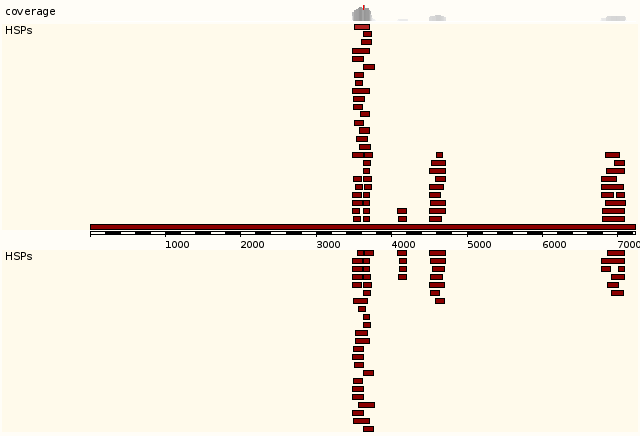 |
Рис. 3 Выравнивание хитов против запроса |
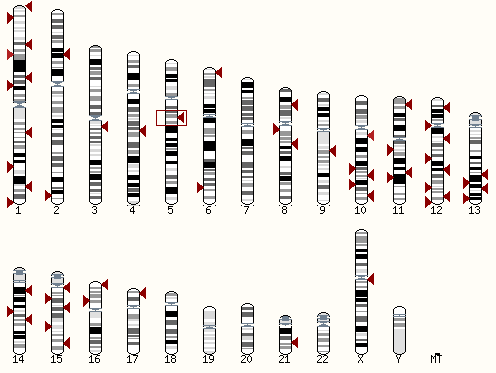 |
Рис. 4Расположение хитов на кариотипе человека |
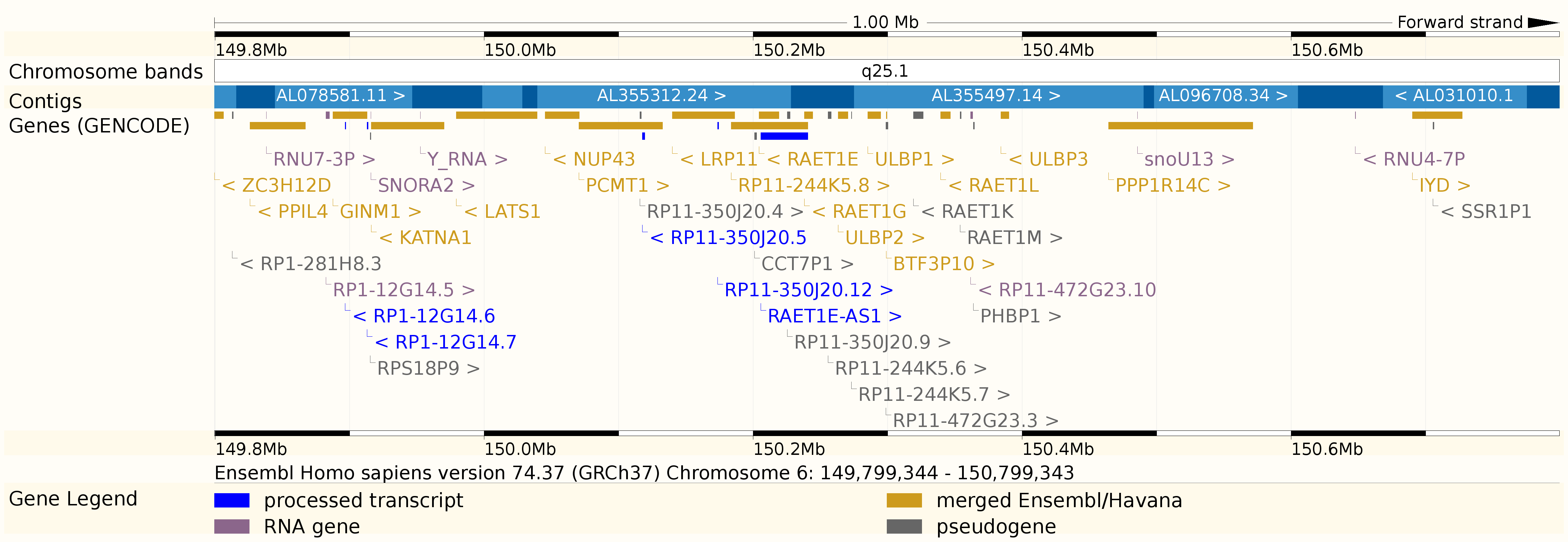 |
Рис. 3 Детальное описание гена |
© Boskhomdzhieva Baina, 2012