UCSC(Human Genome Browser Gateway)
Найдем выбранный в в задании по Online-Blast белок человека в UCSC браузере и укажим:
короткое и полное имя гена: Homo sapiens basic transcription factor 3 (BTF3)
на какой цепи он закодирован: прямая цепь
в какой хромосоме находится: 5
К каким плечу и полосе принадлежит:q 13.2
Координаты гена в последовательности хромосомы:72794250-72801448
Сколько альтернативных продуктов закодировано в гене:2
Число экзонов и длина аминокислотной последовательности: 125 аминокислот, 2 экзона
Для получения изображения были выбраны следующие треки:
Гены USCS и RefSeq
Метилирование ДНК
Консервативность последовательности
Полиморфизмы
Заметим, что гены представителей разных групп позвоночных хорошо выравниваются с геном чедовека, особенно в случае с макакой-резус,
которая выравнивается по всей последовательности, как наиболее эволюционно близкий представитель позвоночных.
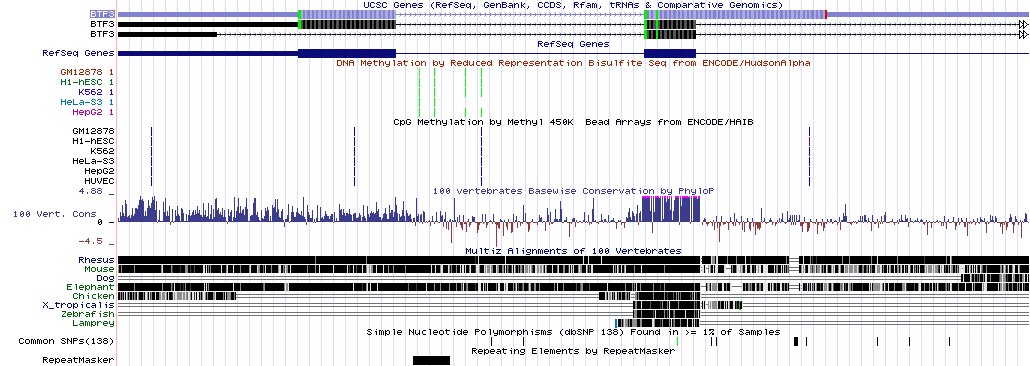 |
Рис. 1 Вариант визуализации гена из Human Genome Browser Gateway с определенными треками |
Ensembl
Задача-построить выравнивание выбранного гена человека с гомологичным геном шимпанзе и определить число различий.Получили выравнивание в формате CLUSTAL.
Далее сравним его с числом часто встречающихся полиморфизмов в этом же гене. На рисунке 2 представлена информация
о том, сколько вaриантов известно по каждому из типов возможного полиморфизма для данного гена.
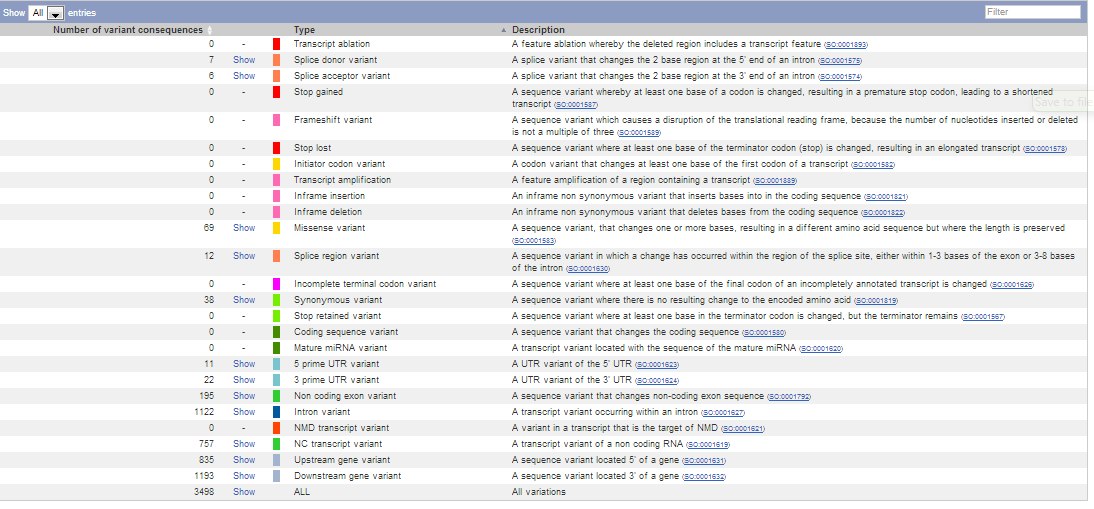 |
Рис. 2 Полиморфизмы для гена человека BTF3 |
Для оценки эволюционного расстояния между последовательностями используем команду dismart. В итоге получили матрицу, где для
каждой пары последовательностей показано "эволюционное расстояние" в количестве замен на 100 пар оснований. В итоге получаем параметр равный 1.04, что свидетельствует о близости видов(последовательности хорошо выравниваются
и сходны между собой).
NCBI Map Viewer
Рассмотрим фрагмент генома в NCBI Map Viewer.Для этого, при поиске в Ensemble необходимо перейти на вкладку location, далее щелкнуть NCBI Map Viewer, где сразу
откроется искомый фрагмент генома (см. рис. 3)
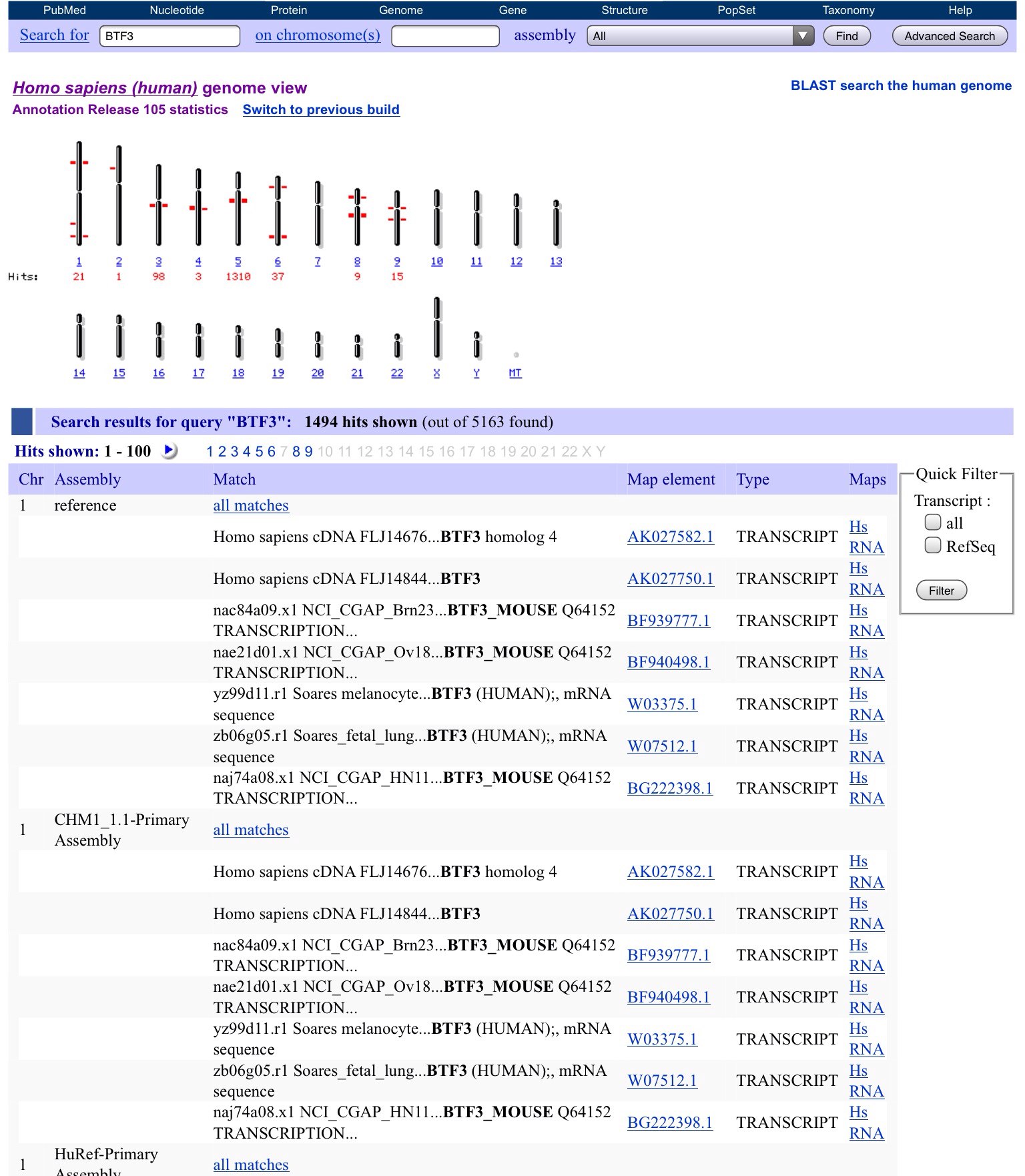 |
Рис. 3 Результат поиска в Map Viewer |
Для того, чтобы посмотреть ген BTF3 в геномном браузере NCBI, можно просто ввести его название в поиске Map Viewer. В результате получим список хитов и
картинку с их расположением на хромосомах(см. рис.4)
 |
Рис. 4 Окресность гена BTF3 и расположение его в хромосоме, полученные через NCBI Map Viewer |
На мой взгляд Map Viewer удобен тем, что предоставляет быстрый доступ ко многим сервисам NCBI, в отличие от других геномных браузеров. Минусами является
вертикальное расположение треков, из-за их невместимости приходится прокручивать слева направо.Поэтому интерфейс мне понравился меньше, чем Ensembl.
© Boskhomdzhieva Baina, 2012



