Построение модели структур A-,-B,и Z- формы ДНК с помощью инструментов пакета 3DNA
С использованием инструментов пакета 3DNA, построим A-,B- и Z-форму дуплекса ДНК, последовательность одной из нитей которого
представляет собой 5 раз повторенную последовательность GATC. Структуры дуплексов сохраним соответственно в файлах gatc-a.pdb, gatc-b.pdb и gatc-z.pdb с помощью
следующих команд:
- fiber -a gatc-a.pdb
- fiber -b gatc-b.pdb
- fiber -z gatc-z.pdb
Работа со структурами нуклеиновых кислот с помощью средств программы Jmol
С помощью программы Jmol выделим в каждой из созданных структур дуплексов ДНК следующие атомы и химические группировки:
- сахарофосфатный остов ДНК;
- все нуклеотиды;
- все аденины;
- атом N7 во всех гуанинах;
- атом N7 в первом по последовательности гуанине;
- акцепторный стебель:остатки 2-7 и соответствующие им 66-71
- Т-стебель: остатки 49-53 и соответствующие им 61-65
- антикодоновый стебель: остатки 38-44 и соответствующие им 26-32
- Д-стебель: остатки 22-25 и соответствующие им 10-13
В результате получили следующие изображения:
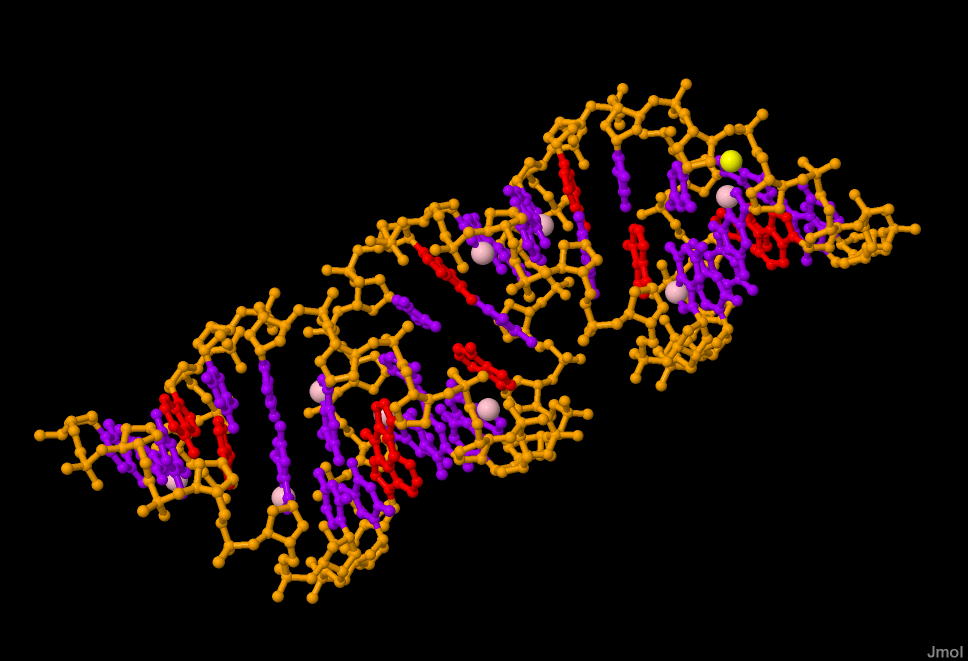 |
Рис 1. Модель A- формы ДНК |
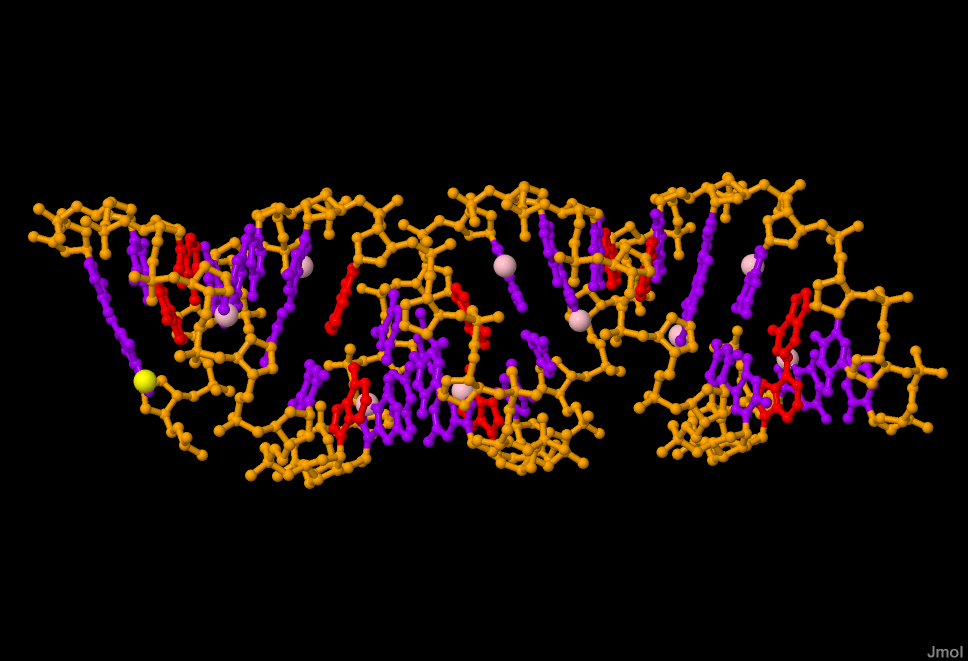 |
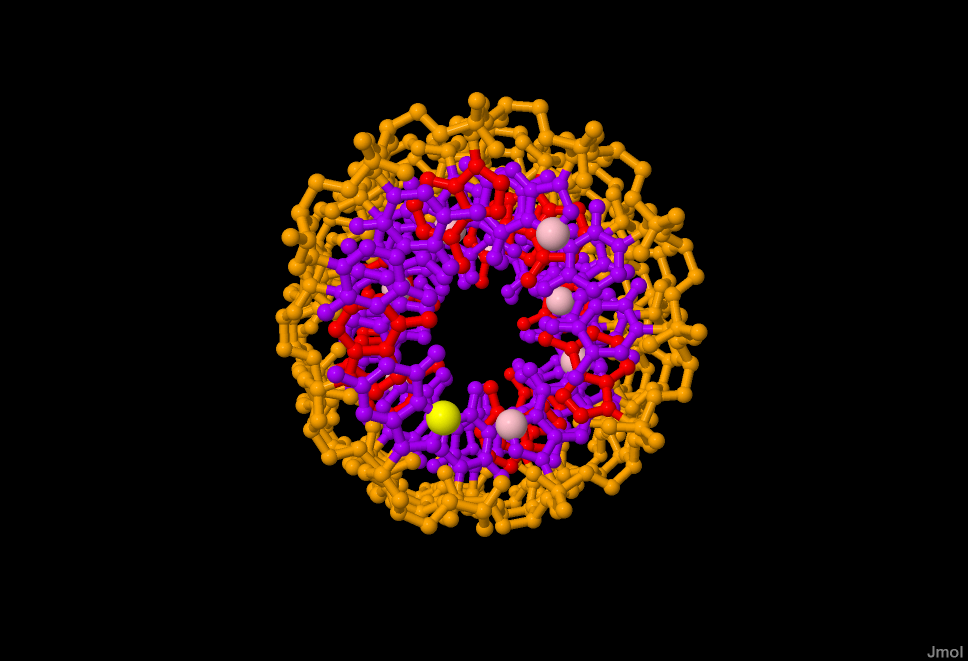 |
Рис 1,2,3 Остов ДНК выделен оранжевым цветом, атомы оснований фиолетовые(кроме аденина-красный),атом N7 во всех гуанинах розовый,а в первом по последовательности гуанине-желтый |
Получение файлов .pdb по идентификаторам 1i3j и 1ml5
На сайте PDB выполним поиск по идентификаторам 1ml5 и 1i3j.
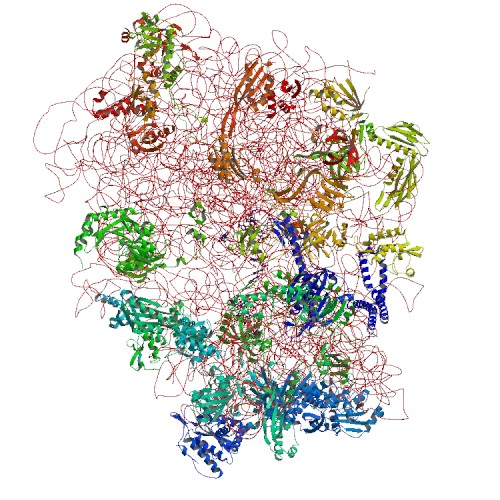 |
Рис 4. Structure of the Escherichia coli ribosomal termination complex with release factor 2.(1ml5) |
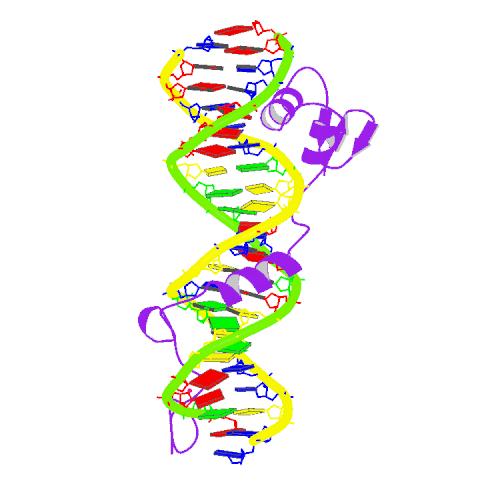 |
Рис 5. Intertwined structure of the DNA-binding domain of intron endonuclease I-TevI with its substrate.(1i3j) |
Проверка заданных структур ДНК и РНК на наличие разрывов
Данные структуры можно рассмотреть в программе Jmol, изучив их на налилие разрывов. В рассматриваемых структурах 1i3j и 1ml5 разрывы отсутствуют.Ниже представлены изображения только нуклеиновой кислоты и проволочной модели для рассматриваемых структур..png) |
Рис 6.Нуклеиновая кислота (1ML5) |
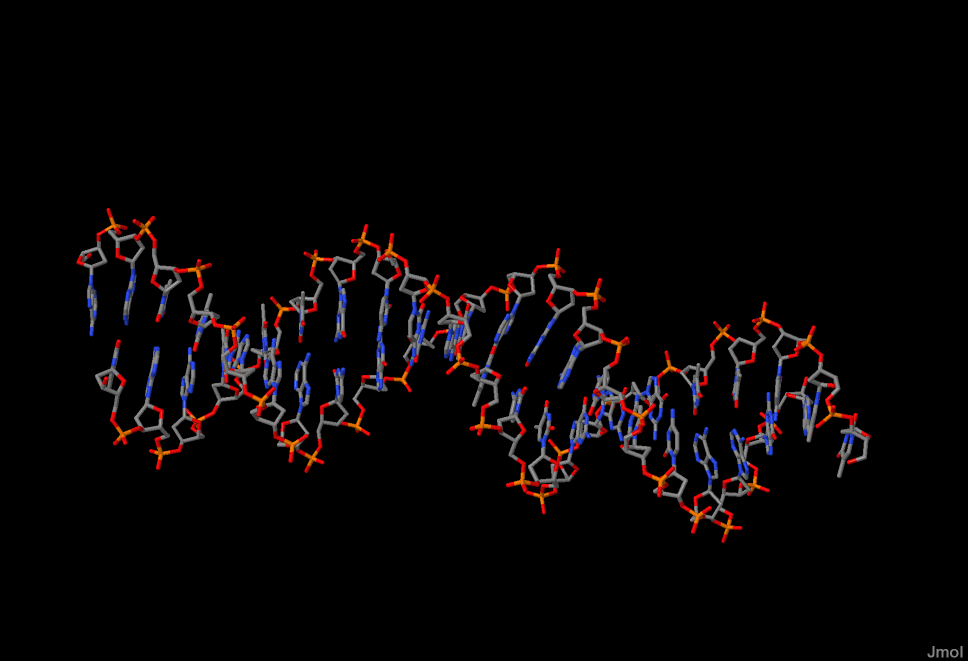 |
Рис 7. Нуклеиновая кислота (1i3j) |
Сравнение структур 3-х форм ДНК с помощью JMol
С помощью программы JMol откроем файл gatc-b.pdb.Рассмотрим структуру и визуально определим большую и малую бороздку.Выберем в структуре гуанин, например G9. Определим,какие атомы оснований явно обращены в сторону большой бороздки, а какие в сторону малой.
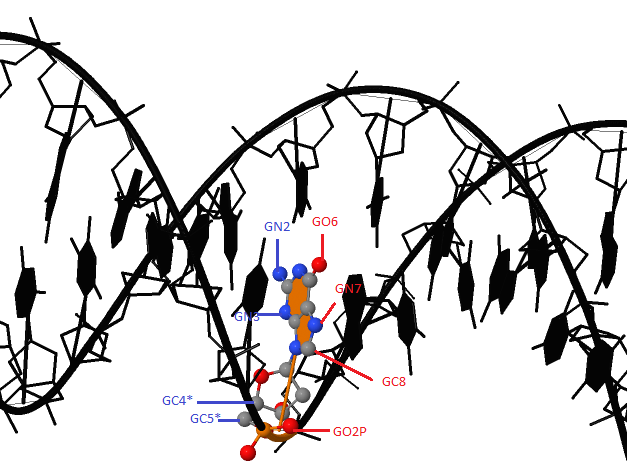 |
Рис 8. Выбранный девятый гуанин.Красным отмечены атомы, обращенные в сторону большой бороздки, синим цветом- в сторону малой |
Молекула гуанина, построенная в программе ChemSketch.Обращенные атомы выделены тем же цветом.
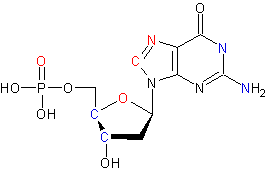 |
В таблице отмечены : (B) -атомы, обращенные в сторону большой бороздки(красный цвет), (b)- атомы, обращенные в сторону малой бороздки-синий цвет.
ATOM 165 P G A 9 -8.877 2.510 -25.152
ATOM 166 O1P G A 9 -10.072 2.945 -24.395
ATOM 167 O2P(B)G A 9 -9.120 1.561 -26.260
ATOM 168 O5' G A 9 -8.094 3.791 -25.705
ATOM 169 C5'(b)G A 9 -7.132 4.443 -24.854
ATOM 170 C4'(b)G A 9 -5.883 4.778 -25.645
ATOM 171 O4' G A 9 -4.919 3.686 -25.648
ATOM 172 C3' G A 9 -6.097 5.085 -27.127
ATOM 173 O3' G A 9 -5.197 6.115 -27.518
ATOM 174 C2' G A 9 -5.704 3.797 -27.850
ATOM 175 C1' G A 9 -4.491 3.471 -26.984
ATOM 176 N9 G A 9 -4.033 2.058 -27.101
ATOM 177 C8 (B)G A 9 -4.777 0.915 -27.294
ATOM 178 N7 (B)G A 9 -4.063 -0.183 -27.354
ATOM 179 C5 G A 9 -2.753 0.265 -27.190
ATOM 180 C6 G A 9 -1.535 -0.463 -27.165
ATOM 181 O6 (B)G A 9 -1.358 -1.672 -27.284
ATOM 182 N1 G A 9 -0.435 0.386 -26.975
ATOM 183 C2 G A 9 -0.504 1.758 -26.828
ATOM 184 N2 (b)G A 9 0.660 2.389 -26.656
ATOM 185 N3 (b)G A 9 -1.648 2.440 -26.851
ATOM 186 C4 G A 9 -2.724 1.631 -27.035
Сравнение основных спиральных параметров разных форм ДНК
Результаты изучения структур из файлов gatc-a.pdb, gatc-b.pdb и gatc-z.pdb с помощью JMol запишем в виде таблицы:
|
A-форма |
B-форма |
*Z-форма |
Тип спирали (правая или левая) |
правая |
правая |
левая |
Шаг спирали (A) |
28,03 |
33,75 |
43,50 |
Число оснований на виток |
11 |
10 |
12 |
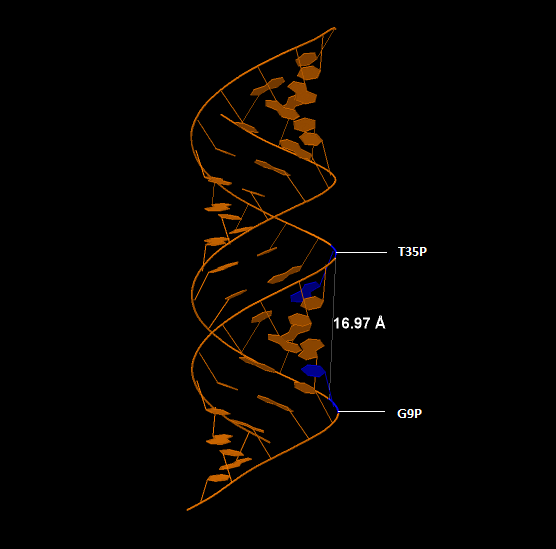
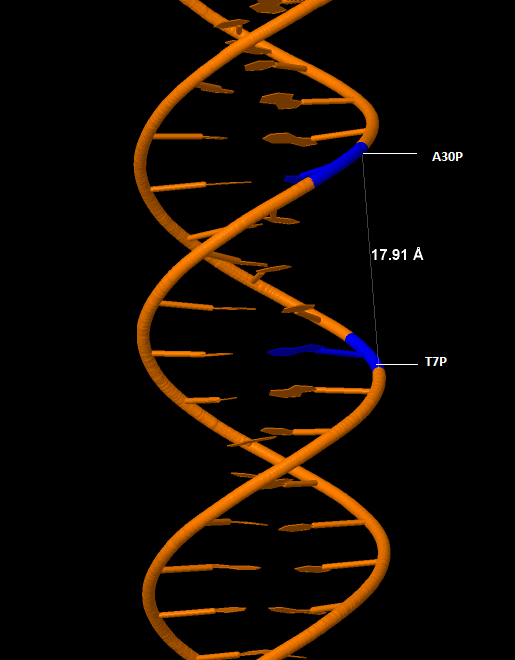
Ширина большой бороздки |
16,97 |
17,91 |
18,30 |
Ширина малой бороздки |
7,98 |
11.69 |
8,68 |
 |
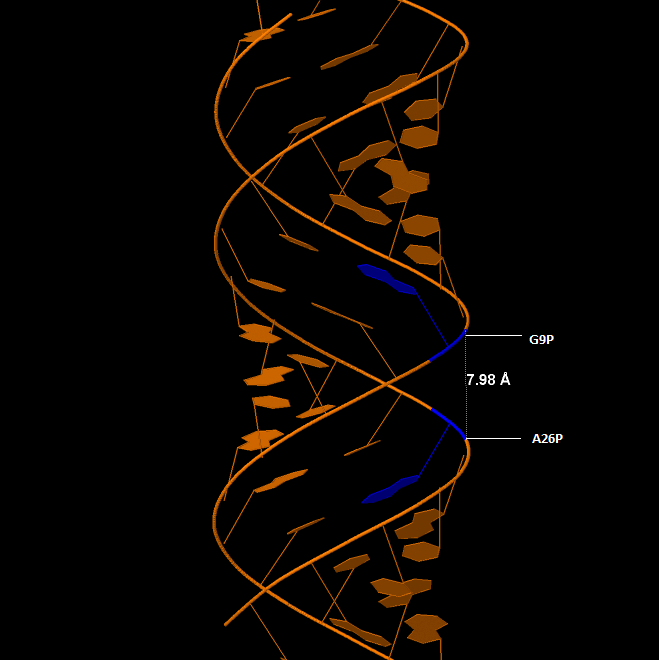 |
Рис 9,10 Большая и малая бороздки A-ДНК |
 |
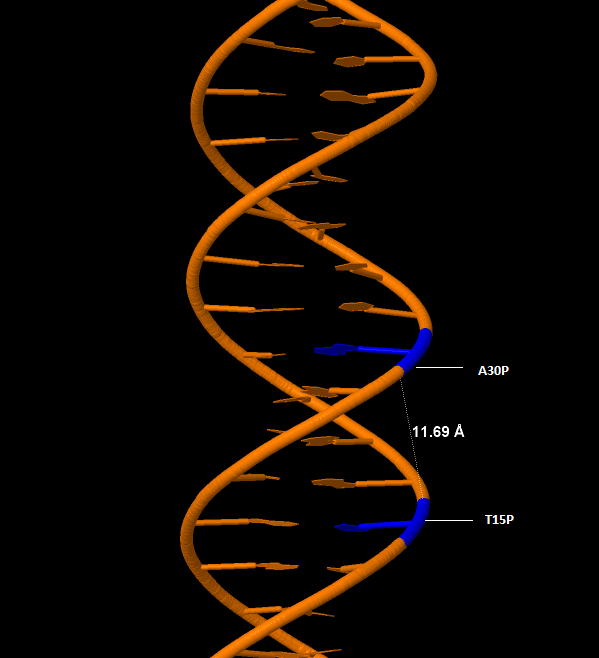 |
Рис 11,12 Большая и малая бороздки B-ДНК |
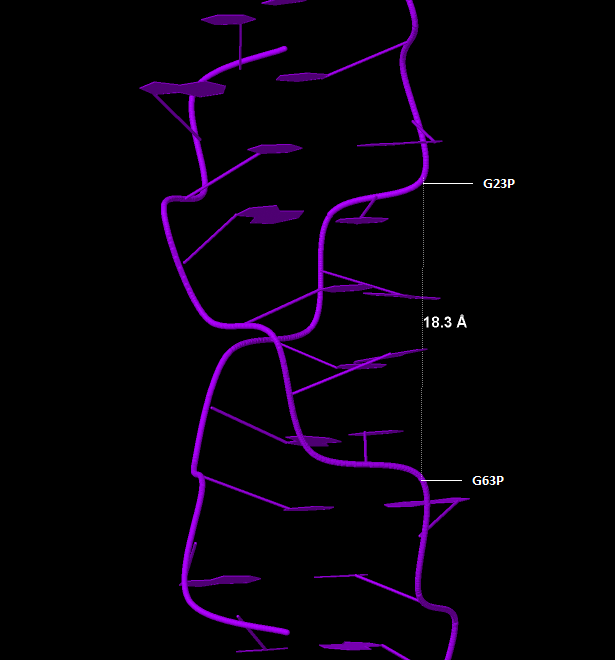 |
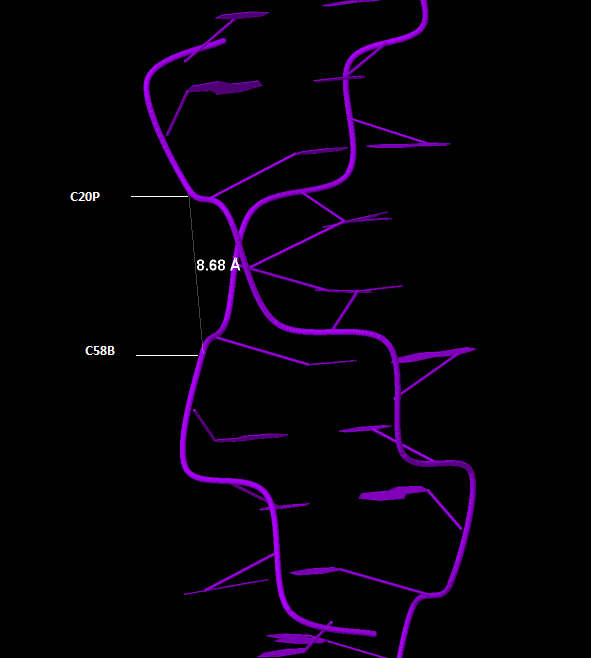 |
Рис 13,14 Большая и малая бороздки Z-ДНК |
Сравнение торсионных углов в структурах A- и B-форм ДНК
С помощью комманды Setting->Torsion в JMol измерим торсионные углы гуанина.В таблице ниже приведены, кроме найденных величин, значения торсионных углов, данные для сравнения(см. в презентации)
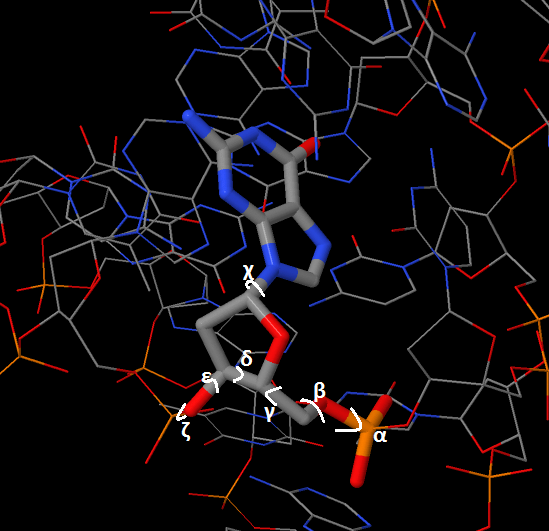 |
| α | β | γ | δ | ε | ξ | χ |
| A-форма (презентация) | ||||||
| -62 | 173 | 52 | 88 или 3 | 178 | -50 | -160 |
| А-форма (JMol) | ||||||
| -51.7 | 174.79 | 41.71 | 79.07 | -147.78 | -75.05 | 22.76 |
| B-форма (презентация) | ||||||
| -63 | 171 | 54 | 123 или 131 | 155 | -90 | -117 |
| В-форма(JMol) | ||||||
| -29.89 | 136.35 | 31.14 | 143.40 | -140.77 | -160.50 | 82.09 |
Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA
Значения торсионных углов в A-, B-, Z-форм, тРНК и ДНК были получены с помощью команд find_pair и analyze пакета 3DNA.
A-форма Strand I base alpha beta gamma delta epsilon zeta chi 1 G --- 174.8 41.7 79.0 -147.8 -75.1 -157.2 2 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 3 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 4 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 5 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 6 A -51.7 174.8 41.7 79.0 -147.8 -75.1 -157.2 7 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 8 C -51.7 174.8 41.7 79.0 -147.8 -75.0 -157.2 9 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 10 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 11 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 12 C -51.7 174.8 41.7 79.1 -147.7 -75.1 -157.2 13 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 14 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 15 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 16 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 17 G -51.7 174.8 41.7 79.0 -147.8 -75.1 -157.2 18 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 19 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 20 C -51.7 174.8 41.7 79.1 --- --- -157.2 Strand II base alpha beta gamma delta epsilon zeta chi 1 C -51.7 174.8 41.7 79.0 --- --- -157.2 2 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 3 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 4 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 5 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 6 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 7 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 8 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 9 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 10 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 11 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 12 G -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 13 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 14 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 15 A -51.7 174.8 41.7 79.0 -147.8 -75.1 -157.2 16 G -51.7 174.8 41.7 79.1 -147.7 -75.1 -157.2 17 C -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 18 T -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 19 A -51.7 174.8 41.7 79.1 -147.8 -75.1 -157.2 20 G --- 174.8 41.7 79.1 -147.8 -75.1 -157.2 B-форма Strand I base alpha beta gamma delta epsilon zeta chi 1 G --- 136.4 31.1 143.4 -140.8 -160.5 -98.0 2 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 3 T -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9 4 C -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 5 G -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 6 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 7 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 8 C -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9 9 G -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 10 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 11 T -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 12 C -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 13 G -29.9 136.3 31.1 143.3 -140.8 -160.5 -98.0 14 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 15 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 16 C -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 17 G -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 18 A -29.9 136.3 31.1 143.3 -140.8 -160.5 -98.0 19 T -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 20 C -29.9 136.3 31.2 143.3 --- --- -98.0 Strand II base alpha beta gamma delta epsilon zeta chi 1 C -29.9 136.4 31.1 143.4 --- --- -98.0 2 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 3 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 4 G -29.9 136.3 31.1 143.3 -140.8 -160.5 -98.0 5 C -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 6 T -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 7 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 8 G -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 9 C -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9 10 T -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 11 A -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 12 G -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 13 C -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 14 T -29.9 136.3 31.1 143.3 -140.8 -160.5 -97.9 15 A -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 16 G -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 17 C -29.9 136.3 31.2 143.3 -140.8 -160.5 -98.0 18 T -29.9 136.4 31.1 143.4 -140.8 -160.5 -98.0 19 A -29.9 136.3 31.1 143.3 -140.8 -160.5 -98.0 20 G --- 136.3 31.2 143.3 -140.8 -160.5 -98.0 Z-форма Strand I base alpha beta gamma delta epsilon zeta chi 1 G --- 179.0 -173.8 94.9 -103.6 -64.8 58.7 2 C -139.5 -136.7 50.9 137.6 -96.5 81.9 -154.3 3 G 52.0 179.0 -173.8 94.9 -103.6 -64.8 58.7 4 C -139.5 -136.8 50.8 137.6 -96.5 82.0 -154.3 5 G 51.9 179.0 -173.8 94.9 -103.6 -64.8 58.7 6 C -139.5 -136.8 50.9 137.6 -96.5 82.0 -154.3 7 G 51.9 179.0 -173.8 94.9 -103.6 -64.8 58.7 8 C -139.5 -136.7 50.9 137.6 -96.5 81.9 -154.3 9 G 52.0 179.0 -173.8 94.9 -103.6 -64.8 58.7 10 C -139.5 -136.8 50.8 137.6 --- --- -154.3 Strand II base alpha beta gamma delta epsilon zeta chi 1 C -139.5 -136.7 50.9 137.6 --- --- -154.3 2 G 51.9 179.0 -173.8 94.9 -103.6 -64.8 58.7 3 C -139.5 -136.8 50.9 137.6 -96.5 82.0 -154.3 4 G 51.9 179.0 -173.8 94.9 -103.6 -64.8 58.7 5 C -139.5 -136.8 50.8 137.6 -96.5 82.0 -154.3 6 G 52.0 179.0 -173.8 94.9 -103.6 -64.8 58.7 7 C -139.5 -136.7 50.9 137.6 -96.5 81.9 -154.3 8 G 51.9 179.0 -173.8 94.9 -103.6 -64.8 58.7 9 C -139.5 -136.8 50.9 137.6 -96.5 82.0 -154.3 10 G --- 179.0 -173.8 94.9 -103.6 -64.8 58.7
Можно заметить, что во всех структурах углы при разных основаниях практически совпадают
| α | β | γ | δ | ε | ξ | χ |
| A-форма | ||||||
| -51.7 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.8 |
| B-форма | ||||||
| -29.9 | 136.4 | 31.1 | 143.4 | -140.8 | -160.5 | -98.0 |
| Z-форма (для гуанинов) | ||||||
| 51.9 | 179 | -173.8 | 94.9 | -103.6 | -64.8 | 58.7 |
| Z-форма (для цитозинов) | ||||||
| -139.5 | -136.7 | 50.9 | 137.6 | -96.5 | 81.9 | -154.3 |
Из таблицы видно, что:
В форме A от предыдущей таблицы сильно отличаются углы ε
В форме В сильно отличаются от предыдущей таблицы углы ε и ξ
Углы ε сходны по значению в формах А и В, но сильно отличаются в форме Z.
Форма Z вообще имеет разные значения углов для разных оснований (для гуанина и цитозина)
Ниже представлена таблица торсионных углов для тРНК 1ML5:
Strand I
base alpha beta gamma delta epsilon zeta chi
1 C --- -120.6 52.7 82.6 -175.2 -59.7 -154.1
2 G -99.8 173.4 83.0 78.4 -144.4 -83.7 -169.0
3 G -69.9 161.6 66.4 81.0 -153.8 -64.7 -175.5
4 A -73.5 -175.0 51.0 84.0 -151.0 -66.3 -160.8
5 U -69.1 165.0 64.2 79.6 -151.4 -75.9 -172.0
6 U -53.6 -180.0 58.0 138.4 --- --- -127.7
7 c --- 172.6 40.7 84.7 -152.7 -77.3 -172.4
8 U -48.2 177.4 36.9 85.1 -149.5 -79.8 -165.1
9 G -41.5 174.4 30.8 87.1 -154.5 -73.0 -154.8
10 U -62.9 167.1 50.5 81.4 -160.1 -66.5 -167.6
11 G -59.5 173.1 53.7 83.4 -167.0 -60.4 -173.4
12 u -85.6 -167.5 58.1 80.1 -136.0 -68.8 -156.2
13 P -53.7 170.2 44.1 79.8 --- --- -153.2
14 A --- -174.6 60.3 75.2 -146.7 -62.0 -173.5
15 P -83.0 179.9 69.4 84.5 -151.5 -69.3 -172.9
16 c -90.9 168.7 74.2 81.4 -141.8 -96.2 -176.2
17 U 41.1 162.3 -47.4 99.2 -152.4 -68.5 -155.8
18 G -57.4 162.1 58.1 83.2 -156.6 -80.8 -170.1
19 G -53.1 168.4 51.5 83.9 -151.2 -77.9 -159.8
20 A -71.7 156.3 70.0 84.2 --- --- -158.4
21 g --- 148.3 57.1 91.2 -134.5 -76.4 173.4
22 C -71.7 179.0 58.3 89.4 -145.9 -71.1 -161.4
23 U -69.4 173.0 49.4 82.9 -161.9 -73.5 -160.1
24 C 138.1 -155.7 -164.0 79.5 -169.9 -79.3 -168.2
25 A 131.5 177.1 -153.2 91.4 -117.4 -65.5 -175.3
26 G -49.9 160.2 41.3 80.4 --- --- -166.9
27 G --- 176.6 59.5 157.3 --- --- -79.5
28 g --- 108.3 47.7 86.7 -116.5 -60.3 174.9
29 A 15.8 -113.2 -116.6 101.1 116.2 41.2 -169.7
30 A 79.6 -173.4 178.6 80.0 --- --- -171.0
Strand II
base alpha beta gamma delta epsilon zeta chi
1 G -78.3 170.0 61.9 81.8 --- --- -163.9
2 C -70.1 168.3 54.9 77.3 -156.1 -69.3 -159.6
3 U -59.3 171.4 47.3 82.2 -154.9 -78.8 -162.3
4 U -60.4 173.3 46.4 82.4 -155.3 -64.5 -161.9
5 A -62.7 171.8 48.6 89.2 -158.2 -77.1 -156.2
6 A -65.7 175.4 58.8 80.3 -148.3 -74.3 -166.1
7 G -41.4 157.7 48.3 79.7 -154.7 -74.5 -172.7
8 A -56.7 176.1 38.0 77.1 -146.2 -75.8 -160.7
9 C -81.6 175.5 62.8 82.3 -150.6 -75.5 -157.3
10 A -73.1 -173.0 51.5 83.0 -157.4 -67.8 -161.9
11 C --- 179.2 33.8 83.2 -157.5 -70.0 -169.9
12 a --- -150.8 68.9 155.7 --- --- -85.3
13 G --- 137.1 -67.9 177.6 --- --- -95.6
14 c -71.3 -179.5 48.3 85.6 --- --- -144.8
15 A -71.7 -175.1 49.1 87.7 -143.5 -62.1 -158.3
16 G -58.6 -176.7 38.8 82.0 -166.5 -56.6 -157.3
17 A -56.4 165.2 52.3 82.8 -159.9 -72.3 -164.7
18 C -72.1 175.0 53.7 81.3 -147.4 -76.3 -162.5
19 C -60.6 171.5 48.8 81.8 -148.1 -73.5 -163.4
20 g -73.1 -177.4 62.7 81.8 -136.5 -71.6 -163.1
21 C -57.8 169.1 47.8 77.6 -148.4 -55.0 -161.7
22 G -51.8 175.1 32.0 88.4 -147.4 -66.7 -158.1
23 A -59.9 179.9 47.3 84.3 -152.1 -73.5 -161.4
24 G --- 161.3 178.2 87.4 -148.7 -78.5 -176.7
25 U --- -140.8 50.3 81.2 --- --- -159.5
26 C --- -175.3 153.2 138.3 --- --- -142.9
27 C --- 175.2 47.2 85.8 --- --- -164.0
28 U -68.2 177.7 51.4 82.3 --- --- -160.5
29 U -68.2 177.7 51.2 82.5 -157.5 -68.9 -160.7
30 U --- 177.7 51.3 82.3 -157.6 -68.8 -160.6
Сравнив примерные значения торсионных углов каждого типа со средними значениями из таблицы в предыдущем задании заметим, что тРНК 1ML5 напоминает A-форму молекулы ДНК.
Торсионные углы DNA-protein complex 1I3JStrand I base alpha beta gamma delta epsilon zeta chi 1 T --- 166.7 60.3 146.6 -171.5 -102.4 -105.7 2 C -50.8 -174.0 31.0 150.4 175.8 -114.2 -85.3 3 T -16.9 -174.6 13.5 150.0 -161.2 -114.5 -99.4 4 T 18.2 -162.5 -58.8 151.2 -159.9 -95.2 -95.8 5 G -60.2 175.4 34.8 153.6 -122.6 -179.0 -65.5 6 G -67.3 137.9 49.4 147.5 -149.2 -150.8 -89.9 7 G -71.5 157.8 56.9 147.8 -174.3 -92.2 -122.1 8 T -55.9 -159.4 25.3 144.7 161.1 -82.0 -95.7 9 C 151.3 150.4 -171.3 135.6 -154.7 -113.1 -129.7 10 T -38.9 -174.8 29.9 144.0 -170.3 -109.5 -105.1 11 A -48.8 -178.3 40.5 147.1 -166.1 -113.6 -93.0 12 C 22.8 -169.9 -55.1 153.1 -178.1 -115.7 -91.8 13 C 27.5 -161.8 -61.8 149.8 -159.9 -93.1 -124.5 14 G -55.8 -169.5 37.6 148.3 170.5 -108.5 -99.3 15 T -43.8 -171.3 39.0 147.8 -168.4 -120.7 -103.4 16 T 26.1 -168.3 -53.3 154.4 179.4 -89.1 -107.1 17 T -50.1 -171.0 29.7 156.8 -166.8 -154.0 -84.6 18 A -44.9 159.2 47.7 146.3 177.3 -90.7 -92.8 19 A -37.0 -155.4 25.0 151.9 -162.2 -112.0 -86.8 20 T 24.7 -126.9 -75.8 161.3 --- --- -119.5 Strand II base alpha beta gamma delta epsilon zeta chi 1 A 16.3 -176.4 -51.6 159.8 --- --- -94.6 2 G -52.4 164.8 49.0 151.0 -161.1 -112.2 -107.6 3 A -38.5 160.2 37.6 152.6 -163.9 -149.8 -85.8 4 A -48.7 157.8 48.3 145.1 -168.7 -123.3 -83.3 5 C -41.4 175.4 31.3 144.7 -156.6 -144.8 -86.6 6 C -27.9 -172.7 17.8 142.9 -164.0 -105.2 -105.6 7 C 41.2 174.7 -57.0 151.5 -176.1 -98.0 -112.2 8 A -35.2 167.6 23.9 147.1 -172.2 -120.7 -111.5 9 G -49.4 -177.4 29.3 152.5 -166.5 -150.0 -85.4 10 A -33.9 175.0 46.1 149.0 -175.2 -99.2 -96.8 11 T -48.9 -167.7 35.9 146.1 -171.5 -123.0 -99.1 12 G 32.9 -177.8 -52.9 151.4 165.5 -100.4 -125.1 13 G -42.4 -161.1 33.1 154.1 177.5 -130.9 -94.6 14 C 32.0 -161.0 -57.9 150.1 -178.5 -93.9 -100.8 15 A -21.7 162.3 31.9 147.7 -170.7 -111.2 -96.5 16 A -35.0 -173.9 24.6 151.0 170.0 -131.3 -84.8 17 A -36.7 174.5 42.1 147.1 175.8 -101.8 -85.5 18 T -31.1 178.3 26.2 144.4 -176.8 -119.8 -93.6 19 T 6.0 -151.1 -80.2 143.3 -167.5 -112.0 -108.4 20 A --- 177.9 47.4 155.3 -132.9 -137.0 -85.2
Значения торсионных углов и средние значения по ним для ДНК 1i3j приведены в файле.Желтым цветом выделены наибольшие значения, красным-наименьшие.Самым "деформированным" нуклеотидом можно считать девятый нуклеотид первой цепи (C)
Определение элементов во вторичной структуре РНК
С помощью команд, используемых в предыдущем упражнении, получим информацию о водородных связях:Strand I Strand II Helix 1 (0.005) B:...2_:[..C]C-----G[..G]:..71_:B (0.006) | 2 (0.008) B:...3_:[..G]G-----C[..C]:..70_:B (0.007) | 3 (0.009) B:...4_:[..G]G-*---U[..U]:..69_:B (0.008) | 4 (0.009) B:...5_:[..A]A-----U[..U]:..68_:B (0.008) | 5 (0.007) B:...6_:[..U]U-----A[..A]:..67_:B (0.016) | 6 (0.007) B:...7_:[..U]Ux----A[..A]:..66_:B (0.012) | 7 (0.016) B:..49_:[5MC]c-----G[..G]:..65_:B (0.017) | 8 (0.015) B:..50_:[..U]U-----A[..A]:..64_:B (0.015) | 9 (0.011) B:..51_:[..G]G-----C[..C]:..63_:B (0.009) | 10 (0.014) B:..52_:[..U]U-----A[..A]:..62_:B (0.023) | 11 (0.015) B:..53_:[..G]G----xC[..C]:..61_:B (0.006) | 12 (0.011) B:..54_:[5MU]u-**-xa[1MA]:..58_:B (0.048) | 13 (0.018) B:..55_:[PSU]Px**+xG[..G]:..18_:B (0.026) x 14 (0.007) B:..38_:[..A]A-*---c[OMC]:..32_:B (0.008) | 15 (0.021) B:..39_:[PSU]P-*---A[..A]:..31_:B (0.006) | 16 (0.010) B:..40_:[5MC]c-----G[..G]:..30_:B (0.010) | 17 (0.007) B:..41_:[..U]U-----A[..A]:..29_:B (0.006) | 18 (0.013) B:..42_:[..G]G-----C[..C]:..28_:B (0.005) | 19 (0.011) B:..43_:[..G]G-----C[..C]:..27_:B (0.006) | 20 (0.007) B:..44_:[..A]Ax*---g[M2G]:..26_:B (0.010) | 21 (0.017) B:..10_:[2MG]g-----C[..C]:..25_:B (0.006) | 22 (0.006) B:..11_:[..C]C-----G[..G]:..24_:B (0.017) | 23 (0.009) B:..12_:[..U]U-----A[..A]:..23_:B (0.013) | 24 (0.011) B:..13_:[..C]C----xG[..G]:..22_:B (0.011) | 25 (0.017) B:..14_:[..A]A-**-xU[..U]:...8_:B (0.008) | 26 (0.018) B:..15_:[..G]Gx**+xC[..C]:..48_:B (0.013) x 27 (0.018) B:..19_:[..G]Gx---xC[..C]:..56_:B (0.012) + 28 (0.012) B:..34_:[OMG]g-*---U[..U]:..13_:C (0.012) | 29 (0.005) B:..35_:[..A]A-----U[..U]:..12_:C (0.011) | 30 (0.008) B:..36_:[..A]A-----U[..U]:..11_:C (0.011) |В соответствии с полученными данными определяем:
- Стебли во вторичной структуре тРНК:
G4-U69 U7-A66 G53-C61 [OMC]-34 c U13 [PSU]-39 c A-31 [5MС]-40 c G-30 [OMC]-32 c A-38 [5MС]-49 c G-65Дополнительные взаимодействия в структуре Т-РНК:
[5MU]-54 c [1MA]-58
[PSU]-55 c G-18
[M2G]-26 c A-44
C-1 c G-24
U-12 c A-23
C13 c G-22
A-14 c U-8
G-15 c C-48
G-19 c C-56
Стэкинг-взаимодействия в РНК
Overlap area in Angstrom^2 between polygons defined by atoms on successive
bases. Polygons projected in the mean plane of the designed base-pair step.
Values in parentheses measure the overlap of base ring atoms only. Those
outside parentheses include exocyclic atoms on the ring. Intra- and
inter-strand overlap is designated according to the following diagram:
i2 3' 5' j2
/|\ |
| |
Strand I | | II
| |
| |
| \|/
i1 5' 3' j1
step i1-i2 i1-j2 j1-i2 j1-j2 sum
1 CG/CG 0.76( 0.01) 0.00( 0.00) 3.20( 0.72) 0.46( 0.00) 4.42( 0.73)
2 GG/UC 2.38( 0.91) 0.00( 0.00) 0.08( 0.00) 0.33( 0.00) 2.79( 0.91)
3 GA/UU 3.72( 2.19) 0.00( 0.00) 0.00( 0.00) 2.58( 0.89) 6.30( 3.08)
4 AU/AU 4.79( 3.43) 0.00( 0.00) 0.00( 0.00) 3.04( 1.74) 7.83( 5.18)
5 UU/AA 0.79( 0.01) 0.00( 0.00) 0.00( 0.00) 3.15( 2.66) 3.93( 2.66)
6 Uc/GA 0.82( 0.00) 0.00( 0.00) 0.00( 0.00) 3.37( 1.85) 4.20( 1.85)
7 cU/AG 0.00( 0.00) 0.00( 0.00) 0.43( 0.00) 3.87( 2.83) 4.30( 2.83)
8 UG/CA 0.17( 0.00) 0.00( 0.00) 2.60( 1.28) 0.13( 0.00) 2.90( 1.28)
9 GU/AC 6.87( 3.89) 0.00( 0.00) 0.00( 0.00) 3.54( 1.98) 10.41( 5.87)
10 UG/CA 0.00( 0.00) 0.00( 0.00) 4.46( 2.20) 0.00( 0.00) 4.46( 2.20)
11 Gu/aC 8.81( 4.20) 0.00( 0.00) 0.00( 0.00) 4.17( 0.85) 12.98( 5.05)
12 uP/Ga 5.72( 1.89) 0.00( 0.00) 0.00( 0.00) 8.48( 3.73) 14.20( 5.62)
13 PA/cG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
14 AP/Ac 4.50( 1.95) 0.00( 0.00) 0.00( 0.00) 6.77( 3.74) 11.27( 5.69)
15 Pc/GA 1.75( 0.00) 0.00( 0.00) 0.00( 0.00) 5.14( 3.31) 6.88( 3.31)
16 cU/AG 0.07( 0.00) 0.00( 0.00) 0.71( 0.00) 0.72( 0.72) 1.49( 0.72)
17 UG/CA 0.07( 0.00) 0.00( 0.00) 3.07( 1.75) 0.04( 0.00) 3.18( 1.75)
18 GG/CC 2.97( 1.47) 0.00( 0.00) 0.36( 0.00) 0.20( 0.00) 3.53( 1.47)
19 GA/gC 3.66( 1.01) 0.00( 0.00) 0.00( 0.00) 5.48( 2.64) 9.14( 3.65)
20 Ag/Cg 0.00( 0.00) 0.00( 0.00) 0.35( 0.00) 1.83( 0.78) 2.18( 0.78)
21 gC/GC 3.38( 0.62) 0.00( 0.00) 0.00( 0.00) 7.14( 3.85) 10.52( 4.46)
22 CU/AG 1.05( 0.02) 0.00( 0.00) 0.03( 0.00) 2.47( 2.16) 3.55( 2.18)
23 UC/GA 1.26( 0.30) 0.00( 0.00) 0.00( 0.00) 1.00( 0.06) 2.26( 0.36)
24 CA/UG 0.00( 0.00) 1.98( 0.00) 2.24( 0.18) 0.00( 0.00) 4.22( 0.18)
25 AG/CU 2.43( 0.70) 0.00( 0.00) 0.16( 0.00) 0.00( 0.00) 2.58( 0.70)
26 GG/CC 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
27 Gg/UC 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
28 gA/UU 3.62( 2.17) 0.00( 0.00) 0.00( 0.00) 0.25( 0.00) 3.87( 2.17)
29 AA/UU 2.74( 2.59) 0.00( 0.00) 0.00( 0.00) 0.26( 0.00) 3.00( 2.59)
© Boskhomdzhieva Baina, 2012