Описание трансмембранных белков с известной 3D структурой
Для трёх трансмембранных бета-баррелей и трех трансмембранных альфа-спиральных белков с помощью базы данных OPM был определён ряд параметров, приведённых таблице ниже.
| PDB код | Тип | Мембрана | Толщина гидрофобной части мембраны, Å | Медиана числа остатков в одном трансмембранном участке |
| 2GFP | Спираль | Внутренняя мембрана Escherichia coli | 31.6 ± 1.5 | 21 |
| 2UUH | Спираль | Мембрана эндоплазматического ретикулума Homo sapiens | 29.4 ± 1.1 | 18.5 |
| 4B4A | Спираль | Внутренняя мембрана Aquifex aeolicus | 30.4 ± 1.2 | 22 |
| 1QJP | Баррель | Внешняя мембрана Escherichia coli | 25.4 ± 1.5 | 9 |
| 3PIK | Баррель | Внешняя мембрана Escherichia coli | 24.3 ± 1.2 | 10 |
| 2JK4 | Баррель | Внешняя мембрана митохондрий Homo sapiens | 23.4 ± 2.3 | 7 |
Отбор гомологов
Для белка HPPA_THEMA произвели поиск гомологов с помощью PSI-BLAST. Данный белок принадлежит к бактерии Thermotoga maritima, поэтому при поиске гомологов были исключены белки филлума Thermotogae. Отобранные последовательности гомологов с порогом на E-value 1e-5 и максимальным количеством хитов 1000 представлены в файле
Анализ структуры выданного белка
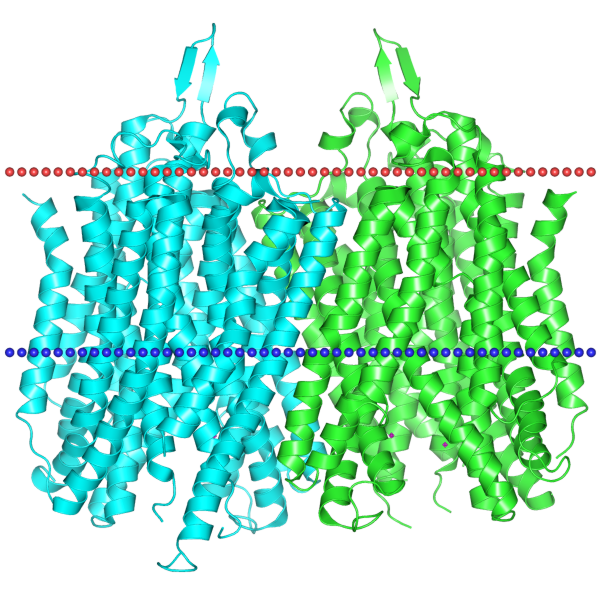 |
Рис.1Структура белка HPPA_THEMA |
Информация о белке HPPA_THEMA, полученная с помощью баз данных OPM и TCDB , представлена в таблице.
| PDB ID | Организм | Тип мембраны | TC-код | Наклон спиралей к нормали | Количество трансмембранных спиралей | Название белка |
| HPPA_THEMA | Thermotoga maritima | Внутренняя мембрана | 3.A.10.1.4 | 0 ± 0°
Отдельно для двух субъединиц: 9°, 9° |
32(2 субъединицы) | Неорганическая пирофосфатаза |
Описание каждого из полей TC-кода для белка HPPA_THEMA:
3.*- первичные активные транспортеры
3.A.*- транспортеры, гидролизующие дифосфатную связь
3.A.10.*- H+ и Na+ транслокация семейства пирофосфатазы
3.A.10.1.4- 2 компонента системы Na+ транслоцирующих пирофосфатаз
Анализ множественного выравнивания
Множественное выравнивание отобранных гомологов было построено с помощью программы Muscle, цветовая схема, позволяющая различать гидрофобные(красный цвет)
и гидрофильные остатки - Hydrophobicity, порог консервативности- 20%.Также к выравниванию добавили аннотацию TM_REAL (участки выбранного белка, являющиеся
альфа-спиралями), привязав к последовательности структуру и отметив трансмембранные спирали буквой "M". Далее добавили к выравниванию предсказание трансмембранных спиралей, выдаваемых программой TMHMM для гомолога, принадлежащего к Spirochaeta bajacaliforniensis.
Для этого создали новую строку аннотации и назвали ее TM_PREDICTED. Проект с выравниванием в файле .
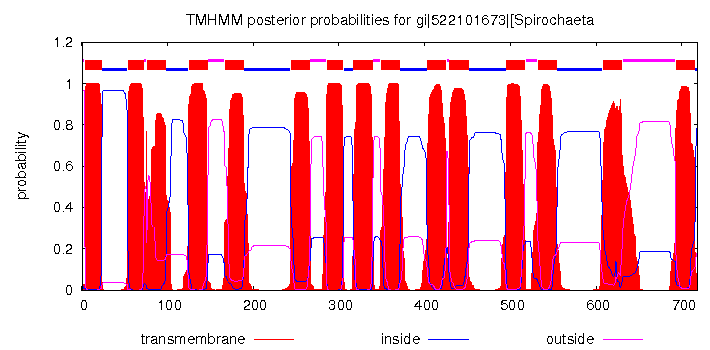 |
Рис.2 Предсказание из TMHMM для гомолога, принадлежащего к Spirochaeta bajacaliforniensis |
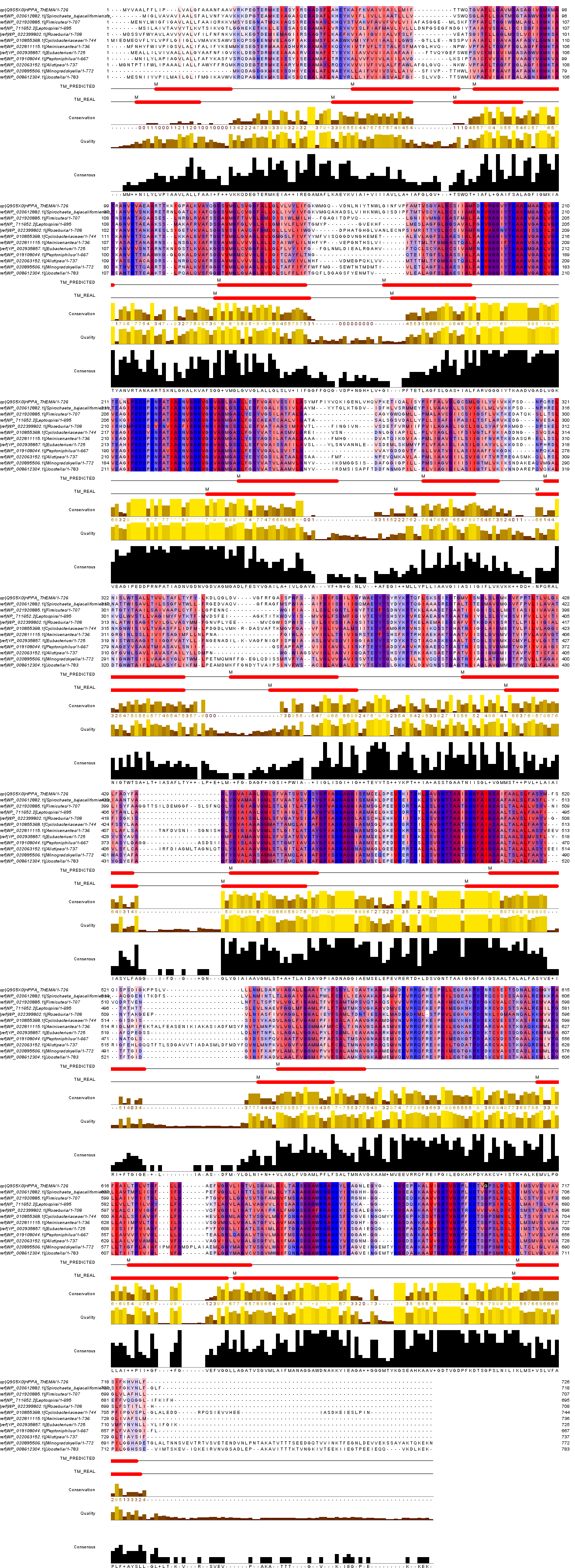 |
Рис.3 Множественное выравнивание HPPA_THEMA и его гомологов |
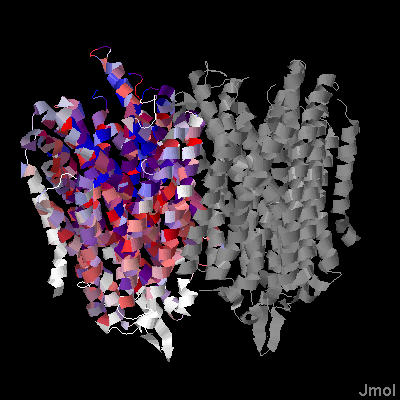 |
Рис.4 Вторичная структура белка HPPA_THEMA |
Часть белка, ориентированная в n-сторону мембраны(внутреннюю)- расположена сверху и содержит положительно заряженные остатки (Arg, Lys), а p-сторона(внешняя) расположена снизу. Участки, относящиеся к трансмембранным спиралям, консервативны(хотя встречаются неконсервативные фрагменты).В трансмемранных спиралях нередко встречаются остатки лейцина, аланина, фенилаланина,валина,метионина, изолейцина, глицина (как видно, трансмембранные участки действительно состоят в основном их гидрофобных остатков). Стоит отметить, что в трансмембранных спиралях также нередко втречаются остатки серина, аспарагина, тирозина(полярны), а также аспарагиновой кислоты и глутаминовой кислоты (полярные и заряжены отрицательно). Как правило эти остатки консервативны, поэтому можно предположить, что их присутствие связано с функцией белка. Если сравнивать результаты, полученные с помощью программы TMHMM и структурную информацию по совпадению на выравнивании, то можно сделать вывод, что трансмембранные спирали в целом были предсказаны верно.Структурная информация больше на несколько остатков, возможно это связано с тем, что программа TMHMM отбирает только те аминокислоты, которые не выходят за пределы мембраны и не являются полярными.
© Boskhomdzhieva Baina, 2014