Пользуясь таксономическим сервисом NCBI определим, к каким таксонам относятся отобранные бактерии.
Получим таблицу:
| Название | Мнемоника | Тип | Класс | Отряд | Семейство |
| Ваcillus subtilis | BACSU | Firmicutes | Bacilli | Bacillales | Bacillaceae |
| Clostridium botulinum | CLOB1 | Firmicutes | Clostridia | Clostridiales | Clostridiaceae |
| Enterococcus faecalis | ENTFA | Firmicutes | Bacilli | Lactobacillales | Enterococcaceae |
| Geobacillus kaustophilus | GEOKA | Firmicutes | Bacilli | Bacillales | Bacillaceae |
| Lactobacillus delbrueckii | LACDA | Firmicutes | Bacilli | Lactobacillales | Lactobacillaceae |
| Listeria monocytogenes | LISMO | Firmicutes | Bacilli | Bacillales | Listeriaceae |
| Streptococcus pyogenes | STRP1 | Firmicutes | Bacilli | Lactobacillales | Streptococcaceae |
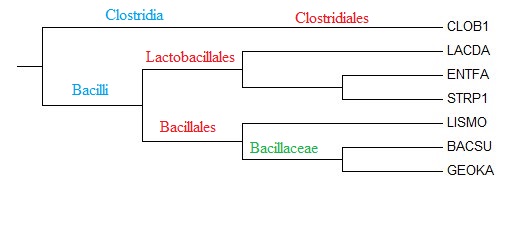 |
Рис.1 Графическое изображение построенного дерева |
Выберем функцию белка-пептил-тРНК гидролаза(PTH) и реконструируем филогенетическое дерево.Далее получим последовательности белков, выполняющих указанную функцию, среди выбранных бактерий fasta-файл.Также провели выравнивание программой muscle(оно представлено на рис 2,в проекте JalView, и в fasta-файле.
 |
Рис.2 Выравнивание белков семейства PTH, взятых из бактерий, представленных в таблице |
Диагностические позиции
Опираясь на полученные данные отметим следующие диагностические позиции:
 |
Рис.3 Выравнивание белков семейства PTH, взятых из бактерий, представленных в таблице, красным показаны диагностические позиции |
В JalView доступны 4 метода для реконструкции филогенетического дерева, которые доступны из меню Calculate > Calculate Tree. Ниже приведены изображения, полученные с помощью программы MEGA.
Average Distant Using % Identity
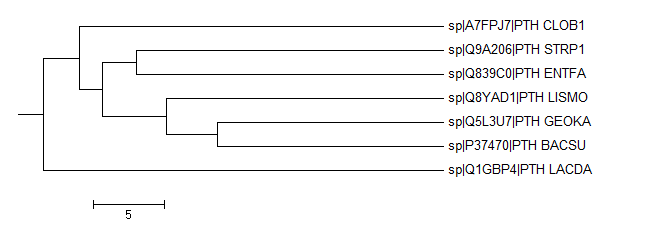 |
Отличие от правильного дерева {LACDA,CLOB1}vs{BACSU, GEOKA, LISMO, ENTFA, STRP1}
Для этого дерева можно отметить большое сходство белка бактерии Clostridium botulinum и белков из класса Bacilli, кроме Lactobacillus delbrueski
Общее:
1){BACSU,GEOKA}vs{LISMO,SPTR1,ENTFA,LACDA,CLOB1}
2){BACSU,GEOKA,LISMO}vs{STRP1,ENTFA,LACDA,CLOB1}
3){BACSU,GEOKA,LISMO,LACDA,CLOB1}vs{ENTFA,STRP1}
Average Distance Using BLOSUM62
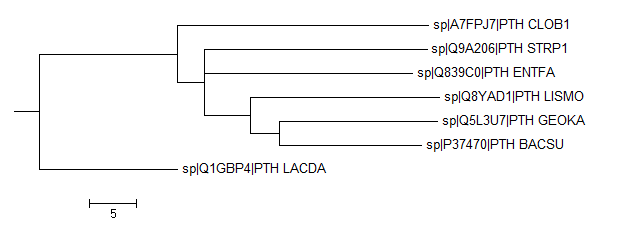 |
Это дерево похоже на правильное, также можно отметить большое сходство белка бактерии Clostridium botulinum и белков из класса Bacilli, кроме Lactobacillus delbrueski
Общее:
1){BACSU,GEOKA}vs{LISMO,SPTR1,ENTFA,LACDA,CLOB1}
2){BACSU,GEOKA,LISMO}vs{STRP1,ENTFA,LACDA,CLOB1}
1){ENTFA,STRP1}vs{BACSU,GEOKA,LISMO,LACDA,CLOB1}
Ветви, присущие только правильному дереву и отсутствующие у реконструированного:
2){ENTFA,STRP1,LACDA}vs{BACSU,GEOKA,LISMO,CLOB1}
Отличающаяся ветвь: {LACDA,CLOB1}vs{BACSU, GEOKA, LISMO, ENTFA, STRP1}
Neighbour Joining Using % Identity
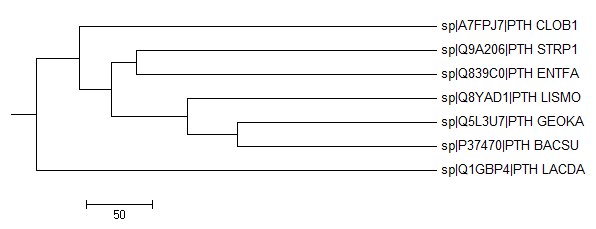 |
Это дерево абсолютно идентично дереву, полученному с помощью метода Average Distant Using % Identity
Neighbour Joining Using BLOSUM62
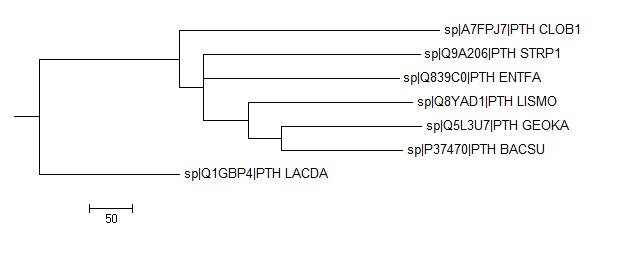 |
Это дерево схоже с деревом, полученным с помощью метода Average Distance Using BLOSUM62
А теперь реконструируем дерево еще одним способом. Для этого, импортируем выравнивание (fasta-файл) в программу MEGA( при импорте выбираем "Analyze"), реконструируем методом "Maximum Parsimony", далее укореняем его.
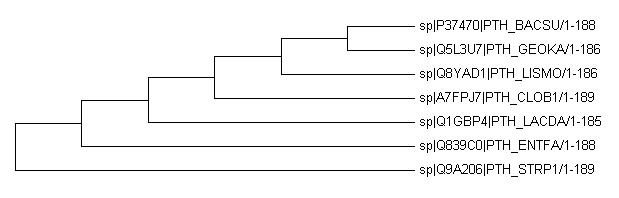 |
Общее:
1){ENTFA,STRP1}vs{BACSU,GEOKA,LISMO,LACDA,CLOB1}
2){ENTFA,STRP1,LACDA}vs{BACSU,GEOKA,LISMO,CLOB1}
3){BACSU,GEOKA}vs{LISMO,SPTR1,ENTFA,LACDA,CLOB1}
4){BACSU,GEOKA,LISMO}vs{STRP1,ENTFA,LACDA,CLOB1}
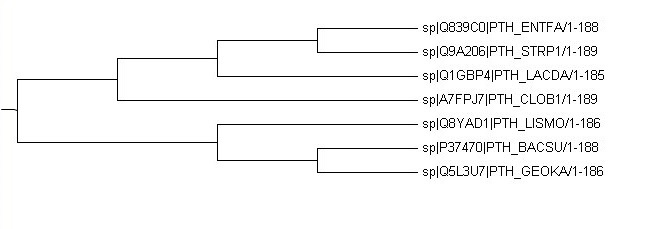 |
Общее:
1){ENTFA,STRP1}vs{BACSU,GEOKA,LISMO,LACDA,CLOB1}
2){ENTFA,STRP1,LACDA}vs{BACSU,GEOKA,LISMO,CLOB1}
3){BACSU,GEOKA}vs{LISMO,SPTR1,ENTFA,LACDA,CLOB1}
4){BACSU,GEOKA,LISMO}vs{STRP1,ENTFA,LACDA,CLOB1}
Заметим, что оба дерева похожи на правильное.
© Boskhomdzhieva Baina, 2014