Практикум 7
Описание записи из UniProt
Задания 1–4
Вариант 1
Чтобы выбрать белок, сначала я нашел все записи, относящиеся к моей бактерии (Photobacterium gaetbulicola Gung47): (taxonomy_id:658445). Нашлось 5 004 записи. В задании сказано, что белок должен быть достаточно полно аннотирован, поэтому я отсортировал выдачу по annotation score (Customize columns → Annotation → отсортировать). Нашлось 20 белков с annotation score 4/5. Из них я выбрал белок, функция которого мне показалась самой любопытной. Это тРНК-серотрансфераза (tRNA sulfurtransferase) (ThiI), AC: A0A0C5WD75. Белок из TrEMBL; класс существования — inferred from homology; функция белка аннотирована автоматически.
Работает он так: сначала другой белок, Iscs, переносит SH-группу со свободного цистеина на цистеин в составе ThiI; получается сульфанилцистеин (рис. 1). Сам ThiI катализирует две реакции. Во-первых, он переносит серу cо своего сульфанилцистеина на урацил в 8-й позиции тРНК (рис. 2), при этом гидролизуется АТФ и окисляется ферредоксин. В аннотации написано, что такая тРНК работает как «фотосенсор» в около-ультрафиолетовом спектре. Во-вторых, он переносит SH-группу на активированный АМФ C-конец белка ThiS (рис. 3).
Вариант 2
Известно, что некоторые Photobacterium светятся, а некоторые — нет; Photobacterium gaetbulicola не светится. Мне стало интересно, что у с нее с генами/белками, которые обеспечивают свечение у Photobacterium.
У светящихся Photobacterium есть оперон luxCDABEG-ribEBHA, в котором расположены гены, обеспечивающие свечение (Urbanczyk и др., 2010). luxA и luxB кодируют две цепи люциферазы, luxC, luxD и luxE кодируют цепи комплекса, синтезирующего субстрат люциферазы (редуктаза жирных кислот), rib кодируют ферменты синтеза флавинмононуклеотида (ФМН), который является вторым субстратом люциферазы. luxG кодирует флавинредуктазу. У некоторых светящихся Photobacterium появляется luxF (неизвестной функции) или отсутствует ribE. lux-оперон был у общего предка Photobacterium и Aliivibrio, а, возможно, и у общего предка Vibrionaceae.
В UniProtKB по поисковому запросу (taxonomy_id:658445) AND ((gene:luxA) OR (gene:luxB) OR (gene:luxC) OR (gene:luxD) OR (gene:luxE) OR (gene:luxF)) ничего не находится; по запросу (taxonomy_id:658445) AND ((gene:ribA) OR (gene:ribB) OR (gene:ribE) OR (gene:ribH)) находится пять записей.
Посмотрим, как расположены гены этих белков в геноме:
wget -O ribs.txt 'https://rest.uniprot.org/uniprotkb/search?query=(taxonomy_id:658445)+AND+((gene:ribA)+OR+(gene:ribB)+OR+(gene:ribE)+OR+(gene:ribH))&format=txt'
# Скачиваем все нашедшиеся записи в файл "ribs.txt".
list="$(grep '^GN ORF' ribs.txt | cut -c 15-25)"
# Вырезаем из них поле "GN ORF_names": оно совпадает с атрибутом "old_locus_tag" в feature table.
grep -E "$(echo $list | tr ' ' '|')" ../../term1/bacterium/genome/*txt | cut -f 6,8,10,15
# Вырезаем из feature table нужные записи и поля.
| Хромосома | Начало гена | Цепь | Название (feature table) | Название (UniProt) | Длина продукта |
|---|---|---|---|---|---|
| 1 | 1 065 395 | + | ribB | ribB | 225 |
| 1 | 1 394 640 | − | ribA | ribA | 350 |
| 2 | 2 507 169 | − | ribA | 199 | |
| 2 | 3 405 255 | − | ribE | ribH | 156 |
| 2 | 3 405 900 | − | ribB | 367 |
Видно, что автоматическая аннотация генома и автоматическая аннотация в UniProt справилась немного по-разному.
Это, конечно, хорошо, но это просто гены синтеза рибофлавина/ФМН, они будут у любого организма и ничего интересного о его отношении к свечению не говорят. Два из них, видимо, расположены в опероне.
Посмотрим, что случилось с lux-генами у этой бактерии. Сначала проверим, не попал ли какой-нибудь белок Photobacterium gaetbulicola в один кластер UniRef с белками, кодируемыми lux-опероном других Photobacterium.
wget -O luxs.txt 'https://rest.uniprot.org/uniprotkb/search?query=(taxonomy_id:657)+AND+((gene:luxA)+OR+(gene:luxB)+OR+(gene:luxC)+OR+(gene:luxD)+OR+(gene:luxE)+OR+(gene:luxF))&format=tsv&fields=accession&size=500'
# Скачиваем в файл "luxs.txt" AC белков, кодируемых lux-опероном, для всего рода Photobacterium.
list="$(tail -n +2 luxs.txt)"
for AC in $list
do
echo 'https://rest.uniprot.org/uniref/search?query=uniprot_id:'${AC}'&format=tsv&fields=id,organism' >> list_UniRef_URL.txt
done
wget -q -O UniRef_lux.txt -i list_UniRef_URL.txt
# Ищем в UniRef записи по UniProt AC (поле почему-то называется "uniprot_id") из нашего файла и скачиваем из них UniRef ID и организмы.
grep 'gaetbulicola' UniRef_lux.txt
Нет, таких белков не нашлось. Тогда сделаем иначе: возьмем референсный белок из каждого кластера UniRef50, в котором есть белки, кодируемые lux-опероном Photobacterium, и выровняем по геному Ph. gaetbulicola с помошью BLAST.
list="$(grep 'UniRef50' UniRef_lux.txt | cut -f 1 | sort -u)"
# Оставляем только записи из UniRef50, убираем повторы. Таких записей оказалось 20.
for ID in $list
do
echo 'https://rest.uniprot.org/uniref/search?query=id:'${ID}'&format=fasta' >> list_UniRef50_URL.txt
done
wget -q -O lux.fna -i list_UniRef50_URL.txt
# Скачиваем референсные последовательности в формате FASTA в файл "lux.fna".
BLAST → load from file → lux.fna; Target databse → UniProtKB; Restrict by taxonomy → Photobacterium gaetbulicola Gung47 [658445].щ
Дефолтные настройки алгоритма не трогал. Нашлось довольно много белков (95). Давайте посмотрим, не находится ли гены каких-нибудь из них близко друг к другу или к генам синтеза рибофлавина.
Наполовину вручную сделал файл list_BLAST_URL.txt с URL страничек с AC нашедшихся записей. (Как написано на странице с результатами работы BLAST, эти результаты удалятся через две недели, поэтому воспроизвести это с этим же файлом скоро не получится, надо будет заново бластовать.)
wget -q -O list_BLAST_AC.txt -i list_BLAST_URL.txt
list="$(cut -c 4- list_BLAST_AC.txt | sort -u)"
for AC in $list
do
echo 'https://rest.uniprot.org/uniprotkb/search?query=accession:'${AC}'&format=txt' >> BLAST.txt
done
wget -q -O BLAST_plain.txt -i BLAST.txt
# Скачиваем записи для всех белков, нашедшихся с помощью BLAST.
grep '^GN ORF' BLAST_plain.txt | cut -c 15-25 > BLAST_locus_tag.txt
# Вырезаем из записей ORF_names, чтобы по ним найти гены этих белков в feature table.
Дальше на питоне:
import pandas as pd
# Записываем ORF_names (locus_tags) в список.
locus = []
file_locus = open('BLAST_locus_tag.txt')
for i in file_locus:
locus.append(i.strip())
file_locus.close()
del file_locus
# Дописываем для рибофлавиновых генов, которые мы нашли отдельно.
locus.extend(['H744_2c2261', 'H744_1c1221', 'H744_1c0908', 'H744_2c3032', 'H744_2c3031'])
# Непростым способом создаем таблицу pandas с нужными генами (теперь я понимаю, что можно было сделать простым).
ft = open('../../term1/bacterium/genome/GCF_000940995.1_ASM94099v1_feature_table.txt')
header = ft.readline()[0:-1].split('\t')
list_BLAST = []
for i in ft:
a = i[0:-1].split("\t")
if a[19][14:] in locus:
d = dict()
for j in range(len(header)):
d[header[j]] = a[j]
list_BLAST.append(d)
ft.close()
del ft
blast = pd.DataFrame(list_BLAST)
blast['start'] = blast['start'].astype('int')
blast['end'] = blast['end'].astype('int')
# Выписываем в отдельные таблицы гены, расположенные на одной цепи одной хромосомы.
plus1 = blast[(blast['chromosome'] == '1') & (blast['strand'] == '+')]
plus2 = blast[(blast['chromosome'] == '2') & (blast['strand'] == '+')]
minus1 = blast[(blast['chromosome'] == '1') & (blast['strand'] == '-')]
minus2 = blast[(blast['chromosome'] == '2') & (blast['strand'] == '-')]
# Смотрим, нет ли среди них таких, между которыми меньше 300 нуклеотидов.
# В feature table гены уже отсортированы по координате, поэтому эту таблицу можно не сортировать.
for i in range(len(plus1) - 1):
if plus1.iloc[i + 1]['start'] - plus1.iloc[i]['end'] <= 301:
print(f"+1 {i}")
for i in range(len(plus2) - 1):
if plus2.iloc[i + 1]['start'] - plus2.iloc[i]['end'] <= 301:
print(f"+2 {i}")
for i in range(len(minus1) - 1):
if minus1.iloc[i + 1]['start'] - minus1.iloc[i]['end'] <= 301:
print(f"-1 {i}")
for i in range(len(minus2) - 1):
if minus2.iloc[i + 1]['start'] - minus2.iloc[i]['end'] <= 301:
print(f"-2 {i}")
Нашлось две таких пары: два рибофлавиновых гена (про них уже написано выше) и вот эти два гена: ptsN (ORF_names: H744_2c0661, UniProt AC: A0A0C5WM50) и hpf (ORF_names: H744_2c0662, UniProt AC: A0A0C5WJR3). ptsN нашелся при сравнении с UniRef50_A0A1L7H6W2 (luxA), hpf — UniRef50_A7KQQ2 (luxC). Посмотрим на них внимательнее (рис. 4, рис. 5).
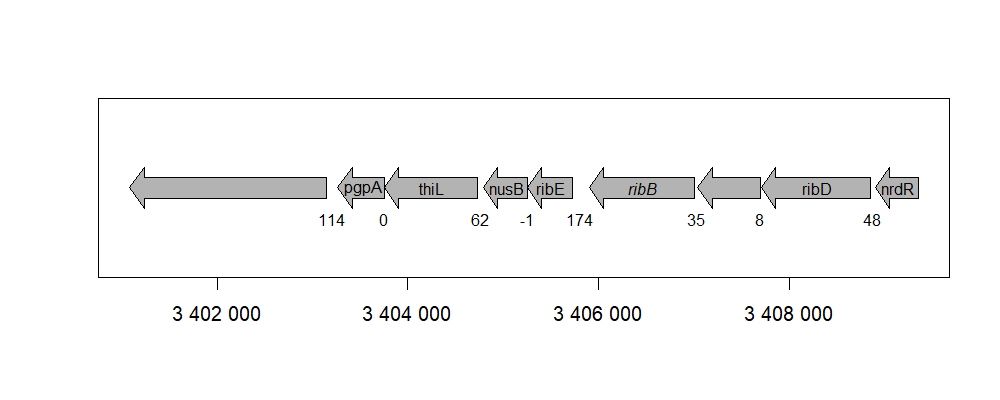
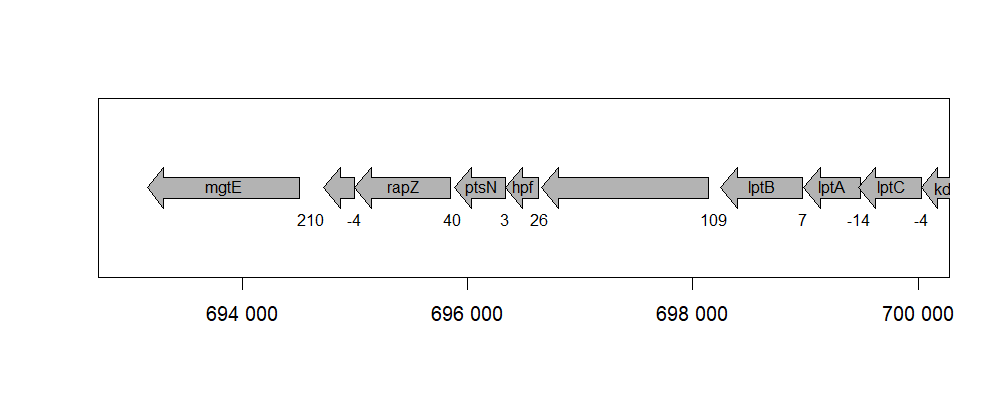
Мое предположение было в том, что гены lux-оперона у Ph. gaetbulicola могли не потеряться, а приобрести какую-то другую функцию. Тогда они были бы насколько-то похожи на своих «предков» из lux-оперона, и они (или хотя бы некоторые из них) могли бы остаться так же расположены в геноме. Я понимаю, что вероятность этого мала, но если бы это было так, я ожидал бы увидеть примерно то, что видно на рис. 5: два гена, белки которых по последовательности напоминают белки, кодируемые lux-опероном, причем расположены они в том же порядке: сначала похожий на luxC, потом — на luxA.
Но это объяснение, конечно, не выдерживает проверки. Белок, соответсвующий hpf, в UniProt называется «Putative sigma-54 modulation protein»; здесь, видимо, автоматические аннотации снова расходятся во мнениях, потому что это, кажется, разные вещи: по запросу (protein_name:"sigma-54 modulation protein") находится 3 809 записей, (gene:"hpf") — 22 259, (protein_name:"sigma-54 modulation protein") AND (gene:"hpf") — 44. Допустим, я поверю аннотации UniProt. Скачаем TSV с taxonomic lineages для выдачи по запросу (protein_name:"sigma-54 modulation protein") и посмотрим, в каких таксонах встречается этот белок.
cut -f 2 *tsv | tail -n +2 | tr ',' '\n' | grep '(class)' | sort | uniq -c | sort -g -r -k 1 | head
cut -f 2 *tsv | tail -n +2 | grep 'Gammaproteobacteria' | tr ',' '\n' | grep 'order' | sort | uniq -c | sort -g -r -k 1
1492 Gammaproteobacteria (class)
526 Flavobacteriia (class)
513 Betaproteobacteria (class)
122 Cytophagia (class)
122 Alphaproteobacteria (class)
121 Actinomycetia (class)
98 Bacteroidia (class)
83 Sphingobacteriia (class)
78 Planctomycetia (class)
61 Clostridia (class)
365 Enterobacterales (order)
327 Pseudomonadales (order)
140 Alteromonadales (order)
112 Xanthomonadales (order)
108 Oceanospirillales (order)
86 Vibrionales (order)
84 Legionellales (order)
64 Moraxellales (order)
42 Chromatiales (order)
25 Pasteurellales (order)
24 Cellvibrionales (order)
20 Methylococcales (order)
16 Thiotrichales (order)
16 Aeromonadales (order)
9 Nevskiales (order)
3 Kangiellales (order)
2 Salinisphaerales (order)
1 Orbales (order)
1 Cardiobacteriales (order)
1 Arenicellales (order)
Большая часть записей относится к гамма-протеобактериям, хотя этот белок есть и у других классов бактерий (еще там есть приколы в виде одной записи в желто-зеленых водорослях и насекомых); внутри гамма-протеобактерий он встречается у разнообразных отрядов. Очевидно, что белок с таким распространением по таксонам никак не может появляться на уровне общего предка нескольких видов одного рода. Если бы мое предположение было верно, для таких белков, наверное, не удалось бы ничего аннотировать, и там было бы написано что-то вроде «uncharacterized protein». Чтобы посмотреть на функцию этого белка, составил такой запрос: (protein_name:"sigma-54 modulation protein") AND (cc_function:*). По нему нашлась одна (!) запись, да и то в ней написано вот это:
DE RecName: Full=Ribosome hibernation promotion factor {ECO:0000255|HAMAP-Rule:MF_00839};
DE Short=HPF {ECO:0000255|HAMAP-Rule:MF_00839};
DE AltName: Full=Hst23 {ECO:0000303|PubMed:9852014};
DE AltName: Full=Putative sigma-54 modulation protein;
DE AltName: Full=SigL modulation protein;
GN Name=yvyD; Synonyms=hpf {ECO:0000255|HAMAP-Rule:MF_00839}, yviI;
GN OrderedLocusNames=BSU35310; ORFNames=orf189;
, то есть это HPF, у которого в альтернативных названиях написано «putative sigma-54 modulation protein». Запрос (gene:hpf) AND (cc_function:*) находит 17 545 результатов. Что-то тут явно нечисто. По-хорошему, надо было бы разобраться во взаимоотношениях сущностей «sigma-54 modulation protein» и «hpf», но это уже, наверное, выходит за рамки этого практикума. Если выбрать только те записи, в которых функция получена экспериментально — (gene:hpf) AND (cc_function_exp:*) — то их останется семь штук, в т.ч. для E. coli K12. Там написано, что он стабилизирует димеризацию рибосом в неактивные 100S-рибосомы в стационарной фазе. Остановимся на этом.
Для второго гена, ptsN, с функцией не сильно лучше. (gene:ptsN) AND (cc_function:*) находит семь записей; в шести из них, включая E. coli K12, написано одно и то же: «seems to have a role in regulating nitrogen assimilation», и приведен код ECO, который на страничке превращается во флажок «by similarity». В седьмом написано «not known; lacks the phosphorylation site found in other PtsN proteins».
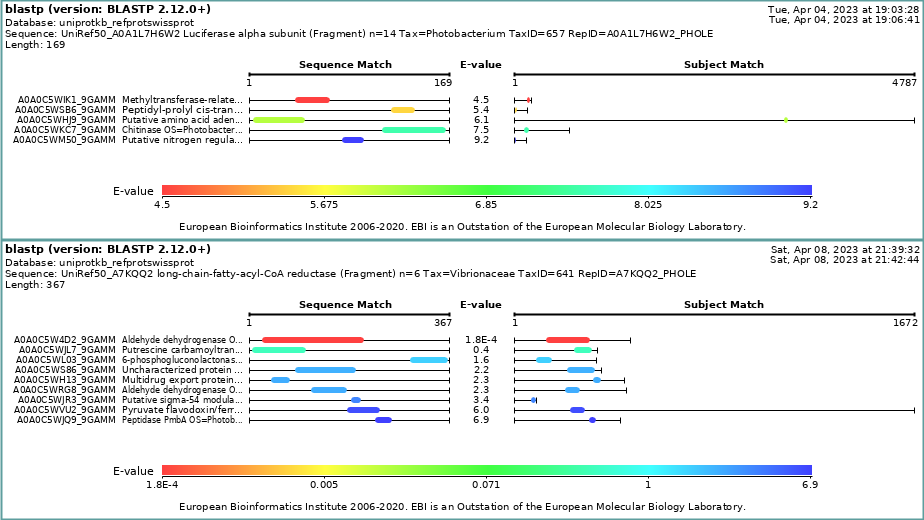
Кстати, нахождение в одном опероне штуки, участвующей в регуляции обмена азота, и штуки, деактивирующей рибосомы, имеет смысл. И абсолютно очевидно, что это не имеет никакого отношения к lux-оперону. Если посмотреть на выравнивание этих генов с luxA и luxC, то это видно и так: они выровнялись только совсем небольшими кусочками (надо было понизить порог E-value в BLAST). Это уже отвлечение, но, на самом деле, из всех этих белков прям хорошо выровнялись только альдегиддегидрогеназа и luxC, что логично: они катализируют, по сути, одну и ту же реакцию (рис. 6).
Если посмотреть на рис. 4, то видно, что это тоже не тот оперон. Рибофлавиновые гены расположены не в том порядке, upstream от них (там, где должны быть lux) находится какой-то регулятор транскрипции с цинковыми пальцами, да и вообще.
Я остановился на выборе из первого варианта. Файлики на kodomo лежат для него.
Задание 5
Выше несколько раз упомянут конфликт аннотации генома (feature table) и аннотации из UniProt. Посмотрим на источник аннотации UniProt для одного из таких случаев. Для нашего hpf-не hpf напротив SubName написано «ECO:0000313|EMBL:AJR07393.1». ECO 0000313 — это информация, автоматически взятая из другой базы данных, в данном случае из INSDC. Если пойти с этим AC в NCBI, то окажется, что это запись в базе данных NCBI Protein. Там в поле DEFINITION правда написано «Putative sigma-54 modulation protein [Photobacterium gaetbulicola Gung47]», но ссылок там нет. В поле DBSOURCE стоит AC записи в NCBI Nucleotide co cборкой второй хромосомы Photobacterium gaetbulicola Gung47. Если для этой записи скачать feature table и найти в ней нужный ген по locus_tag, там все еще будет написано «putative sigma-54 modulation protein». Еще там написано, что таблица трансляции — 11.
Кроме того, в записи в NCBI Protein есть ссылка на BioProject, там есть ссылка на Assembly, там — на RefSeq. И вот если в RefSeq скачать feature table и найти ген по locus_tag, то там уже будет написано «gene: hpf» и «product: ribosome hibernation promoting factor». Я сначала думал, что просто нуклеотидные и транслированные белковые последовательности аннотируют независимо, но в feature table из RefSeq тоже указана таблица трансляции. Я не знаю, как устроен миллион энсибиаевских баз данных, и не могу сказать, где и почему произошла подмена.
Для PtsN из E. coli K12 после CC FUNCTION (с формулировкой «seems to have a role in») написано «ECO:0000250». ECO 0000250 — это информация, вручную добавленная на основе сходства. По-хорошему, потом должна быть указана ссылка, но, как написано на странице с помощью, есть исключения в виде «historic annotations». Эта запись, очевидно, относится к таким. Остальные шесть записей для PtsN с непустыми функциями — тоже.
Давайте посмотрим, как аннотирована функция серотрансферазы из первого варианта. Там написано «ECO:0000256|HAMAP-Rule:MF_00021». Поиск в UniRule по запросу MF_00021 находит запись с UniRule ID UR000075655. На странице с этой записью написано, что для того, чтобы в FUNCTION белку написали то, что там написано, белок должен относиться к бактериям или археям, не быть фрагментированным и удовлетворять сигнатуре HAMAP MF_00021 (https://hamap.expasy.org/rule/MF_00021). Это значит, что он должен насколько-то хорошо выравниваться с матричной последовательностью, полученной на основе трех белков, для которых такая функция подтверждена экспериментально (UniProt AC: P77718, P55913, Q81KU0). Ну, тут вроде понятно.
Список литературы
- Henryk Urbanczyk, Jennifer C. Ast, V. Dunlap. Phylogeny, genomics, and symbiosis of Photobacterium. FEMS Microbiol. Rev. № 35 (2011), pp. 324–342.


