Практикум 8
UniProt Proteomes
Задание 1
Протеом моей бактерии (Photobacterium gaetbulicola Gung 47) — референсный, поэтому я буду использовать его (UP000032303, 4974 белка). В том же семействе, что и моя бактрия, находится холерный вибрион, Vibrio cholerae — очень известная патогенная бактерия. В качестве контроля я буду использовать ее референсный протеом (UP000000584, 3782 белка).
Протеом холерного вибриона может оказаться не очень удачным выбором, если считать необходимым свойством контрольного протеома не только изученность, но и типичность. Vibrionaceae — преимущественно морские бактерии. Большую часть семейства составляют роды Vibrio и Photobacterium. Из примерно 150 видов Vibrio большинство — свободноживущие, многие также бывают ассоциированы с разнообразными морскими организмами, от микроводорослей до ракообразных и рыб. Три вида Vibrio — V. cholerae, V. parahaemolyticus и V. vulnificus — вызывают тяжелые заболевания человека (патогенными могут быть и несколько других видов, но они имеют меньшее значение). Интересно, что большинство штаммов V. cholerae встречается в толще воды, формирует биопленки на поверхности водорослей, панцирей ракообразных (преимущественно копепод) и т. п., и только два серотипа из примерно двухсот патогенны для человека и вызывают холеру. В качестве референсного протеома, конечно, выбран протеом патогенного штамма. Photobacterium также обнаруживаются как симбионты (в широком смысле) морских организмов или свободноживущими (Reen et al., 2006; Machado, Gram, 2017; Sampaio et al., 2022). Штамм Photobacterium gaetbulicola Gung 47 был выделен на литорали в Корее (Kim et al., 2010).
Таким образом, кажется, будто именно V. cholerae должен быть апоморфен и сильно отличаться от типичного представителя семейста; Ph. gaetbulicola, напротив, на первый взгляд не имеет необычных признаков. То есть при сравнении протеомов этих двух бактерий кажется логичным воспринимать именно Ph. gaetbulicola как близкий к предковому, «базовый» вариант, и отличия между ними будут скорее характеризовать особенности эволюционной истории холерного вибриона, а не Ph. gaetbulicola. Но, даже если эти соображения верны, для целей этого практикума это не должно оказаться проблемой.
Скачивание протеомов:
wget -O 'UP000032303.swiss.gz' 'https://rest.uniprot.org/uniprotkb/stream?compressed=true&format=txt&query=proteome:UP000032303'
wget -O 'UP000000584.swiss.gz' 'https://rest.uniprot.org/uniprotkb/stream?compressed=true&format=txt&query=proteome:UP000000584'
Протеом холерного вибриона имеет CPD Standard и не вызывающий опасений BUSCO: C:98.4% (S:98.4% D:0%) F:0.3% M:1.3%. Протеом Ph. gaetbulicola тоже имеет хорошее значение BUSCO: C:99.4% (S:98.9% D:0.5%) F:0.1% M:0.6%, но CPD у него — Outlier (high value). На странице с помощью написано, что для присвоения CPD используется самая маленькая таксономическая группа, включающая этот организм, в которой есть не меньше 30 протеомов, если она не выше класса по рангу. Статус «Outlier (high value)» присваивается, если белков в протеоме не меньше, чем сумма первого и третьего квартилей в этой группе, или количесвто белков в нем отстоит от третьего квартиля не меньше, чем на полтора межквартильных размаха. Я не смог найти, чтобы где-то было указано, по какой группе присвоен CPD данному протеому, но в этом случае это должен быть род Photobacterium: для него находится 210 протеомов, а для меньшей таксоносмической группы, вида Ph. gaetbulicola — толко 3. Действительно, если вычислить пороговое количество белков для присвоения «Outlier (high value)» по всем протеомам Photobacterium, оно окажется равно 4647, а в протеоме Photobacterium gaetbulicola Gung 47 — 4974 белка; он лежит на 91-м процентиле.
Тем не менее, мне кажется, что с этим протеомом все в порядке и его можно использовать.
1) В базе данных есть еще два протеома Photobacterium gaetbulicola из других публикаций других авторов, и у них близкие количества белков: 4850 и 5054. Кстати, у того протеома, у которого 4850 белков, статус CPD — «Close to standard (high value)». Это значит, что я неправильно вычислил пороговое значение. Повторюсь, не смог найти, чтобы где-то было написано, как именно оно присвоено.
2) Если убрать удаленные, избыточные протеомы и протеомы, представляющие не весь геном, то наш протеом переедет на 80-й процентиль, а вычисленное по определению пороговое количество белков для присвоения статуса «Outlier» окажется сильно выше — 5480.
3*) Стоит сказать, что в NCBI Nucleotide я обнаружил 36 kb плазмиду моей бактерии со ссылкой на ту же работу, на котороую ссылается запись с геномом в Assembly, но не добавленную в сборку генома (KC687076.1). Не совсем понимаю, что это значит, но если это значит, что у нее есть такая плазмида, то у нее еще на 31 CDS больше.
В общем, хорошее значение BUSCO и первый пункт кажутся мне достаточно убедительными, чтобы я продолжил с ним работать. Если это необычно большое количество белков в нем вызвано не тем, что их у бактреии на самом деле столько, а какой-то проблемой, то я, если честно, плохо представляю, что именно это могла быть за проблема, а потому не знаю, как ее искать. Правда, меня насторожила отсутствующая в сборке плазмида.
Оценка изученности
Из протеома холерного вибриона 992 белка находятся в Swiss-Prot (26 %), из протеома Ph. gaetbulicola — ни одного.
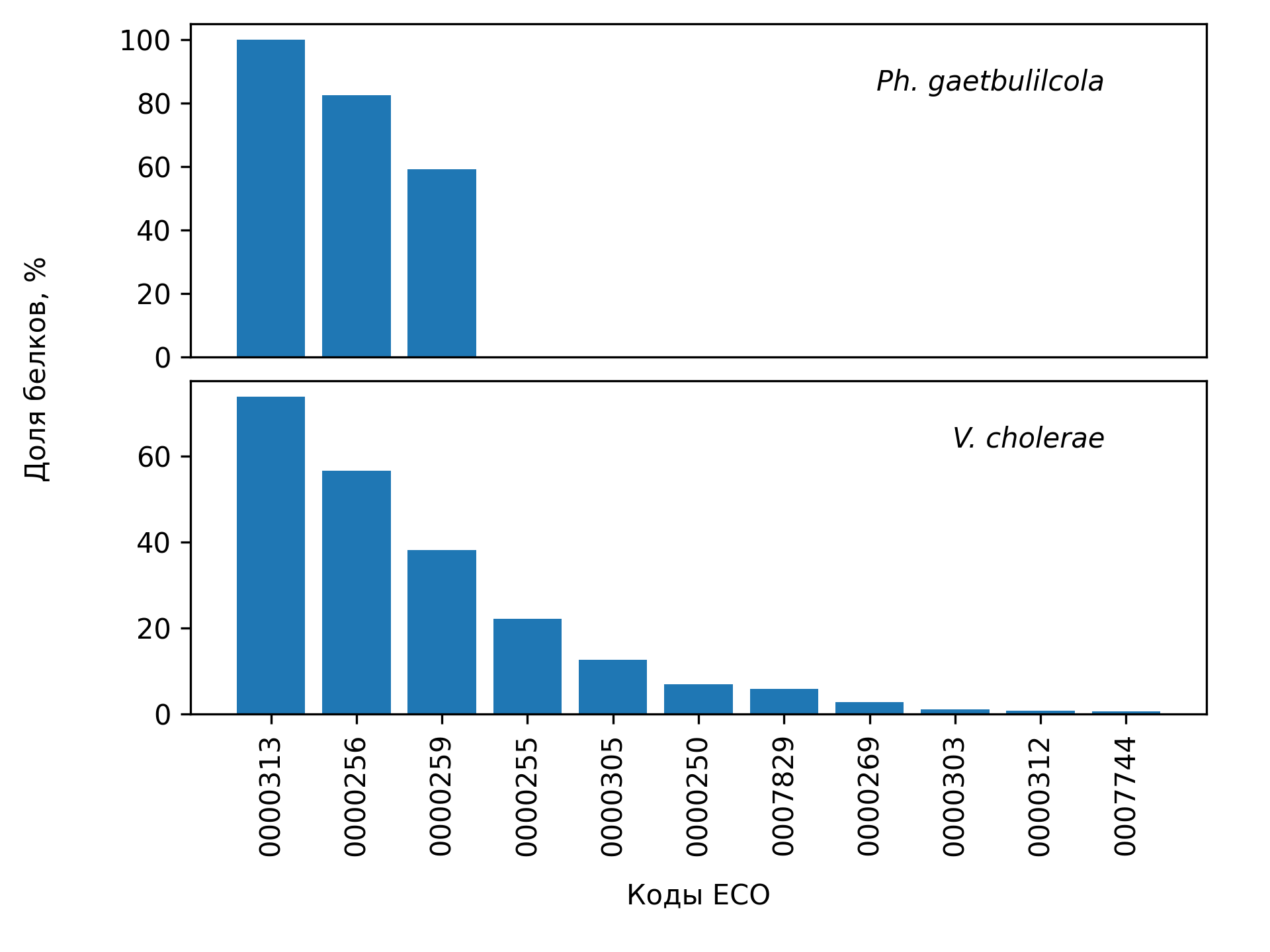
Чтобы лучше охарактеризовать изученность этих протеомов, можно посчитать, какие коды ECO встречаются в их записях (рис. 1, табл. 1).
| Код | Значение | Белков в протеоме V. cholerae | Белков в протеоме Ph. gaetbulicola |
|---|---|---|---|
| 0000313 | In automatic assertions for information which has been imported from another database | 2790 | 4974 |
| 0000256 | In automatic assertions for information which has been generated by the UniProtKB automatic annotation system | 2138 | 4099 |
| 0000259 | Descendant of 0000256 | 1441 | 2942 |
| 0000255 | In manual assertion for information which has been generated by the UniProtKB automatic annotation system; for information which has been generated by various sequence analysis programs that are used during the manual curation process and which has been verified by a curator | 677 | 0 |
| 0000305 | For manually curated information which has been inferred by a curator based on his/her scientific knowledge or on the scientific content of an article | 476 | 0 |
| 0000250 | For manually curated information which has been propagated from a related experimentally characterized protein | 230 | 0 |
| 0007829 | In automatic assertions for information inferred from a combination of experimental and computational evidence | 219 | 0 |
| 0000269 | For manually curated information for which there is published experimental evidence | 103 | 0 |
| 0000303 | For manually curated information that is based on statements in scientific articles for which there is no experimental support | 43 | 0 |
| 0000312 | In manual assertions for information which has been imported from another database | 28 | 0 |
| 0007744 | In manual assertions for information inferred from a combination of experimental and computational evidence | 21 | 0 |
В записях с белками Ph. gaetbulicola есть только ECO 0000313, 0000256 и 0000259, т.е. это только информация из других баз данных или полученная автоматической аннотацией UniProt. Информация, полученная из другой базы данных, упоминается в каждой записи; полученная автоматической аннотацией — в большинстве (хотя бы один из кодов 0000256 и 0000259 встречается в 96 % белковых записей).
В протеоме холерного вибриона мы видим большее разнообразие источников аннотаций. Наверное, самая надежная информация — полученная экспериментально. Информация, непосредственно полученная в эксперименте (0000269), есть для 103 белков (2,7 % протеома). Записей, в которых упоминаются коды 0000269, 0007744 или 0000250 (экспериментальное свидетельство, комбинация экспериментального и вычислительного свидетельства или экспериментальное свидетельство для родственного белка) — 324 (8,6 % протеома). Кажется, не так мало.
Кроме этого, можно просто посчитать, сколько белков с каким классом существования находится в каждом протеоме (табл. 2).
| Бактерия | Evidence at protein level | Evidence at transcript level | Inferred from homology | Predicted |
|---|---|---|---|---|
| V. cholerae | 270 | 9 | 1596 | 1907 |
| Ph. gaetbulicola | 0 | 2 | 2114 | 2858 |
Эти числа дают какое-то представление об изученности протеома холерного вибриона: мы можем сказать, что экспериментально охарактеризованы первые сотни белков этой бактреии.
Для двух белков Ph. gaetbulicola указано свидетельство на уровне транскрипта. На странице с описание UniProtKB flat file написано следующее:
We add the 'Evidence at transcript level' qualifier to all entries with at
least one of the annotations listed below:
1. RP lines containing:
The [MRNA] 'molecule type' [*]
DEVELOPMENTAL STAGE
INDUCTION
RNA EDIT - for non-viral entries only
TISSUE SPECIFICITY
2. CC topics:
RNA EDITING - only for non-viral entries without non-experimental evidence
3. DR lines:
EMBL - with molecule type "mRNA" [*]
[*] These two criteria are only applied to proteins at least 120 residues
long, since small CDS may be regulatory RNAs that are not translated,
and to entries of less than 120 residues which are fragments.
The 'PE 2' assignment overrides assignement to PE category 3.
Найдем эти белки ((proteome:UP000032303) AND (existence:2)) и посмотрм на них внимательнее. Это Lon-протеаза (A0A0C5WWM8) и шаперон DnaK (A0A0C5WTS8). Длина и того и другого больше 120 аминокислотных остатков, поэтому к ним могут быть применимы все три критерия. Только, если просто посмотреть на эти записи, легко убедиться, что ни один из них не выполняется. Я не понял, почему этим белкам присвоен такой статус существования.
Таким образом, по рассмотренным критериям мы можем охарактеризовать протеом Ph. gaetbulicola как максимально неизученный. В качестве характеристики, которая была бы содержательной для протеома, с белками которого совсем не взаимодействовали люди, можем использовать полноту комментариев (поле CC): посмотрим какие «темы» (topics) заполнены в поле CC у белков каждого протеома (рис. 2) (дальше я буду называть их не темами, а полями).
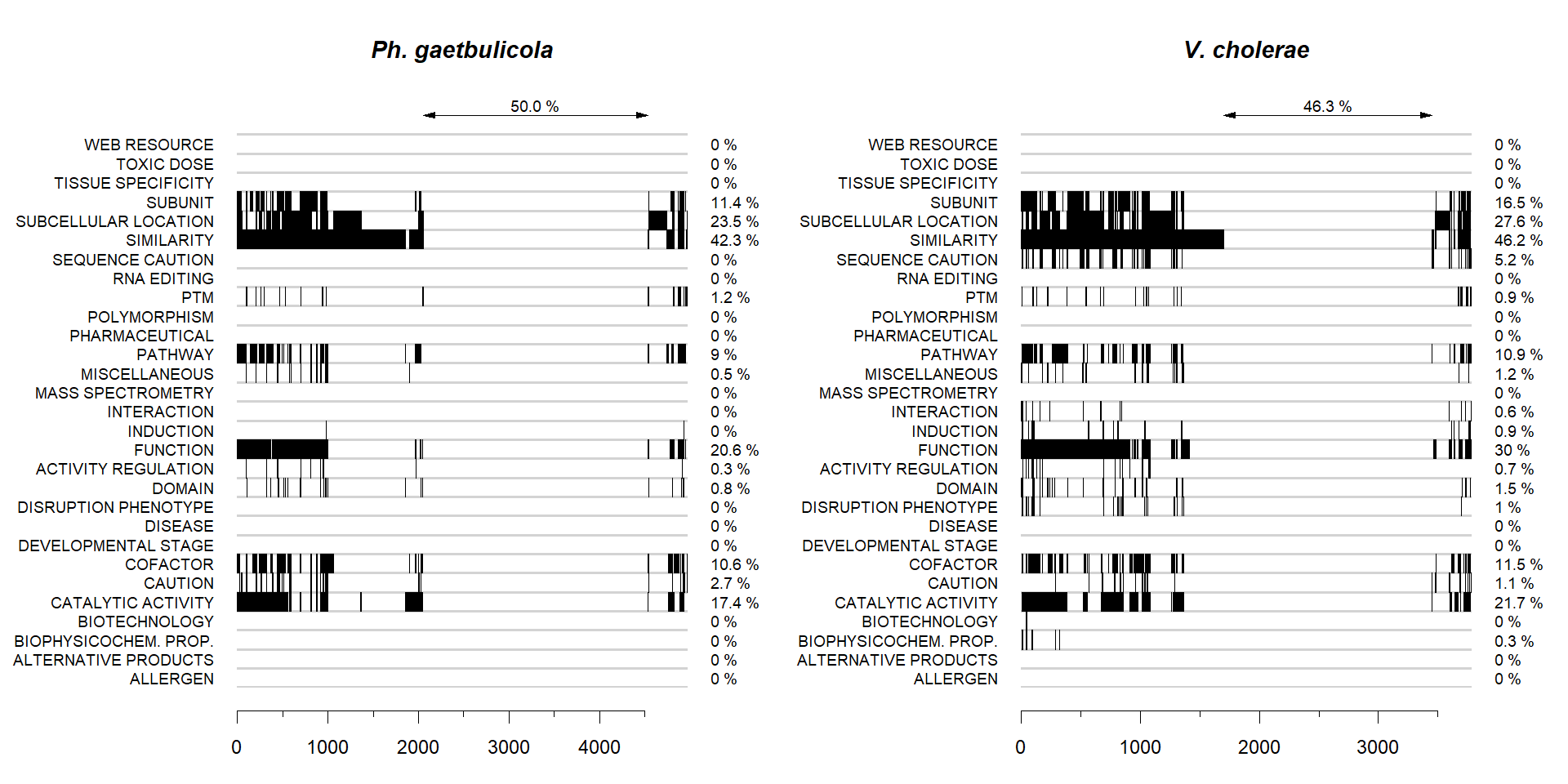
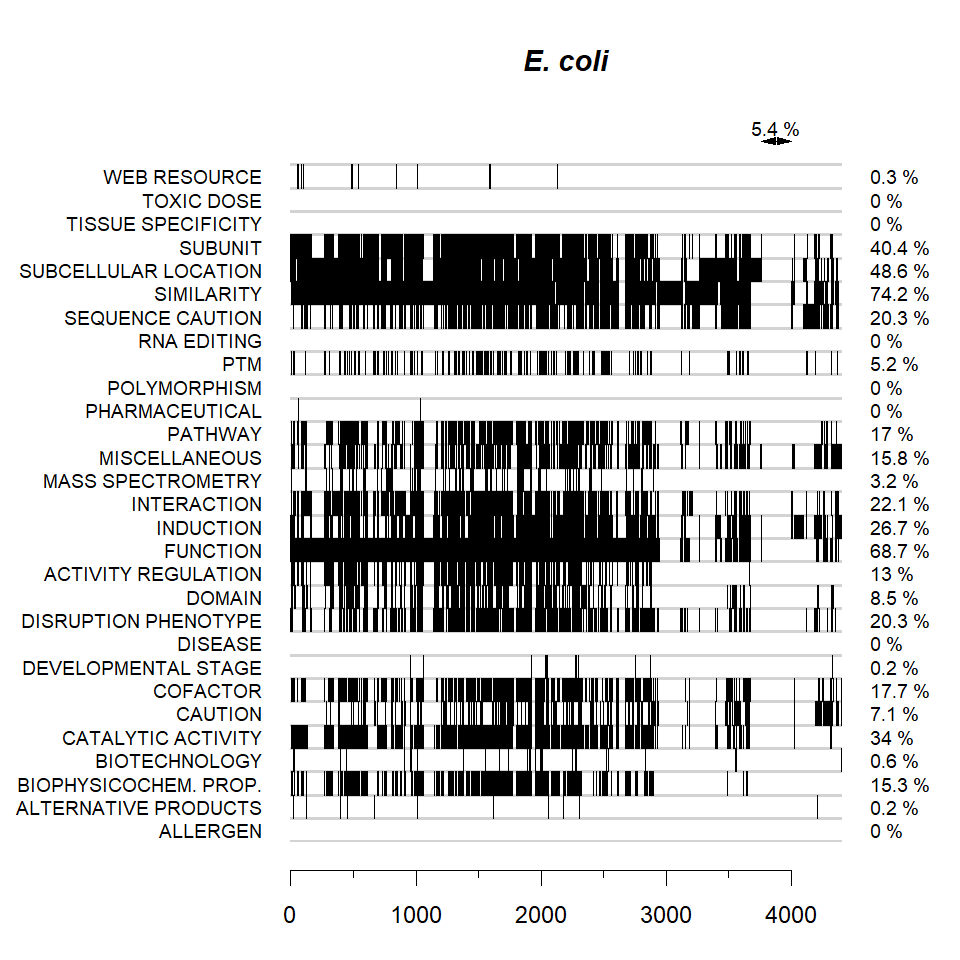
Видно, что на самом деле эти протеомы оказались довольно схожими по полноте комментариев. В среднем у V. cholerae заполненность поля больше на 1,3 %. Просто усреднение может быть не совсем удачной характеристикой изученности, потому что если, например, функция, очевидно, есть у каждого белка, и заполненность этого поля осмысленно сравнивать, то доля белков с заполненной «каталитической активностью» или «посттрансляционными модификациями» зависит не только от изученности протеома, но и от того, сколько ферментов или белков с ПТМ на самом деле есть у бактерии. Из любопытства я нарисовал такую же диаграмму для референсного протеома E. coli (UP000000625, 4403) (рис. 3). Здесь отличия гораздо принципиальнее.
Как можно было бы дополнить или улучшить это задание:
1) Рис. 1 можно было бы оформить не как простую столбчатую диаграмму, а в стиле рис. 2 и рис. 3 — он получился бы информативнее.
2) Рис. 2 и рис. 3, как раз наоборот, получились не очень информативными, потому что а) все рябит в глазах и б) из-за того, что колонка, соответсвующая одному белку, в несколько раз тоньше пикселя, могут возникать артефакты, когда на рисунке зачернена большая доля строки, чем на самом деле (я нарисовал такой же рисунок для человеческого протеома, и на таком количестве белков визуализация совсем сломалась: строки, заполненные на 4 % и на 17 %, на глаз выглядят практически одинаково). Чтобы хотя бы частично исправить обе эти проблемы, следовало бы отсортировать белки так, чтобы в строках было как можно меньше «разрывов», но ни вручную, ни перербором этого сделать нельзя (для Ph. gaetbulicola, например, 191! вариантов), а придумать алгоритм, который сделал бы это за разумное время, я не могу.
3). Можно было бы посмотреть, какие базы данных использованы как источники аннотации, и на какое количество статей ссылаются записи из протеома V. cholerae (последнее, собственно, предлагается в подсказках). Хотя для целей сравнения это было бы не очень полезно, потому что записи для Ph. gaetbulicola не ссылаются ни на одну статью.
Задание 2
Результаты представлены в табл. 3.
Для того, чтобы найти в протеоме трансмембранные белки, я использовал два подхода. Во-первых, я посчитал, у скольких белков в feature table есть хотя бы одна фича TRANSMEM. Во-вторых, я посчитал, у скольких белков в CC Subcellular location упоминается хотя бы одно из следующих subcellular location: Cell inner membrane, Cell outer membrane, Cell membrane, Membrane. Кажется, кроме этих мембран у бактерии никаких других нет, поэтому любой трансмембранный белок должен находиться в них. Понятно, что есть белки, заякоренные в мембране, но не являющиеся трансмембранными, поэтому, если бы subcellular location был аннотирован для всех белков протеома, то это число было бы верхней оценкой количества трансмембранных белков. Наличие в белке трансмембранных доменов будто бы гарантирует, что он будет трансмембранным, но можно представить себе ситуацию, когда про белок известно, что он трансмембранный, но положение трансмембранных доменов неизвестно. Поэтому это число должно быть нижней оценкой количества трансмембранных белков (и нижней оценкой оно будет независимо от того, насколько полно аннотирован протеом).
Из того, как я понимаю научный метод и задания восьмого практикума, здоровый подход к сравнению полученных значений выглядит так. В качестве нулевой гипотезы мы считаем, что на самом деле доля «функциональных групп» в протеомах бактерий одинакова. Если мы видим существенное различие, мы пытаемся объяснить его какой-то технической составляющей аннотации и проверить свое объяснение. Если отвергнуть его не удается, то мы продолжаем считать, что доля функциональных групп в протеомах одинкова. Если мы не находим такого объяснения, то мы имеем право начать спекулировать о биологической природе этих различий.
Во-первых, видно, что доли белков, расположенных в мембранах, близки у Ph. gaetbulicola и V. cholerae, и заметно меньше таковой у E. coli. Первое объяснение, которое приходит в голову — это объясняется полнотой аннотации. Можно принять максимально простую модель и предположить, что среди белков, для которых не аннотирован subcellular location, доля мембранных такая же, как среди аннотированных. Доли мембранных белков среди аннотированных у этих бактерий равны соответственно 60,4, 54,8 и 58,7 %. Видно, что отличие E. coli от Ph. gaetbulicola и V. cholerae исчезло. Я не знаю, насколько это предположение близко к правде. Кажется, по-нормальному это следовало бы сделать так: взять большое число протеомов и посчитать коэффициент корреляции: какую долю изменчивости в содержании мембранных белков можно объяснить изменчивостью в полноте аннотации. Затем можно было бы оценить вероятность увидеть те значения, которые мы увидели для данных бактерий, и, соответственно, сказать, отличаются ли они, например, от типичного представителя Vibrionaceae (о конкретных статистических методах еще нужно подумать). А, может быть, для мембранных белков определить их положение в клетке легче, или, наоборот, сложнее, чем для других белков, и тогда нужно будет использовать нелинейную корреляцию, и т. п.
Доля белков с трансмембранными доменами оказалась очень похожа у всех трех бактерий. С одной стороны, большие различия в изученности протеомов заставляют сомневаться, что она на самом деле так близка, но, с другой стороны, я могу поверить, что трансмембранные домены легко обнаруживаются автоматически, и настоящее количесвто таких белков не сильно отличается от предсказанного. В качестве итоговой оценки количества трансмембранных белков я бы использовал это число, а на CC Subcellular location не обращал внимание.
| Ph. gaetbulicola | V. cholerae | E. coli | |
|---|---|---|---|
| Трансмембранные белки | |||
| CC SUBCELLULAR LOCATION | 706 (14,2 %) | 571 (15,1 %) | 1256 (28,5 %) |
| FT TRANSMEM | 1087 (21,9 %) | 816 (21,6 %) | 955 (21,7 %) |
| Ферменты | |||
| DE EC | 909 (18,3 %) | 877 (23,2 %) | 1699 (38,6 %) |
| CC CATALYTIC ACTIVITY | 863 (17,4 %) | 822 (21,7 %) | 1495 (34,0 %) |
| FT ACTIVE SITE | 383 (7,7 %) | 347 (9,2 %) | 704 (16,0 %) |
| Всего | 1050 (21,1 %) | 954 (25,2 %) | 1827 (41,5 %) |
| Секретируемые белки | |||
| CC SUBCELLULAR LOCATION | 16 (0,32 %) | 31 (0,82 %) | 14 (0,32 %) |
Количество ферментов я попытался оценить тремя способами: количество белков, в описании которых (поле DE) указана классификация ферментов (EC), количество белков с указанной в поле CC каталитической активностью и количество белков с отмеченным в FT активным сайтом, а также объединение этих групп. Количество нашедшихся белков в этих группах довольно ожидаемо убывает (#EC > #CC > #FT); в качестве оценки количества ферментов, наверное, стоит использовать их объединение.
Почти двукратное отличие доли нашедшихся ферментов в протеомах Ph gaetbulicola и E. coli я не могу объяснить никак, кроме как различиями в полноте аннотации (но здесь проверить так же, как с трансмембранными, не получится). Пытаться придумать биологическое объяснение для различия в 4 % между Ph. gaetbulicola и V. cholerae на этом фоне кажется довольно бессмысленным.
Чтобы посчитать количество секретируемых белков, я использовал значение CC Subcellular location: Secreted.
Видно, что у V. cholerae секретируемых белков больше, чем у Ph. gaetbulicola и E. coli. Понятно, что секретируемым белкам холерного вибриона было уделено много внимания, поэтому то, что их известно больше, чем для Ph. gaetbulicola, удивлять не должно. Но секретируемые белки E. coli наверняка изучены не хуже. В Sampaio et al., 2022 написано: «Biochemically, the majority of Vibrio spp. are ... capable of ... degrading gelatin, starch, lipids, chitin, and alginate with extracellular enzymes». Возможно, большее количество секретируемых белков у V. cholerae объесняется разнообразием экологических ниш, которые может занимать эта бактерия, в т. ч. симбиотических с разнообразными хозяевами.
Задание 3
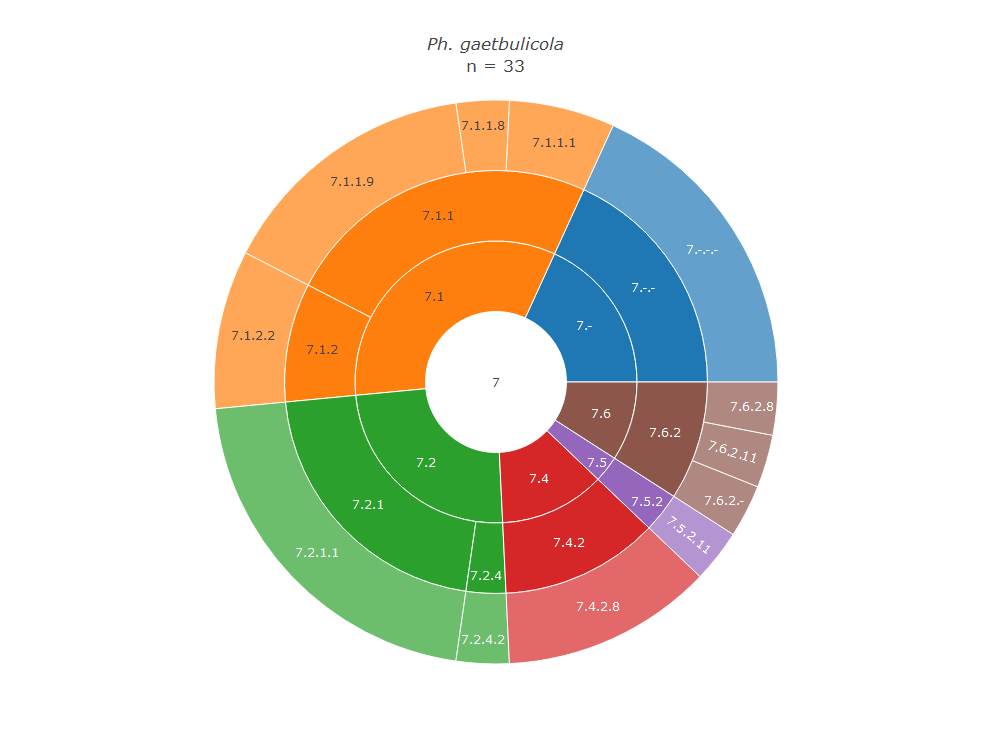
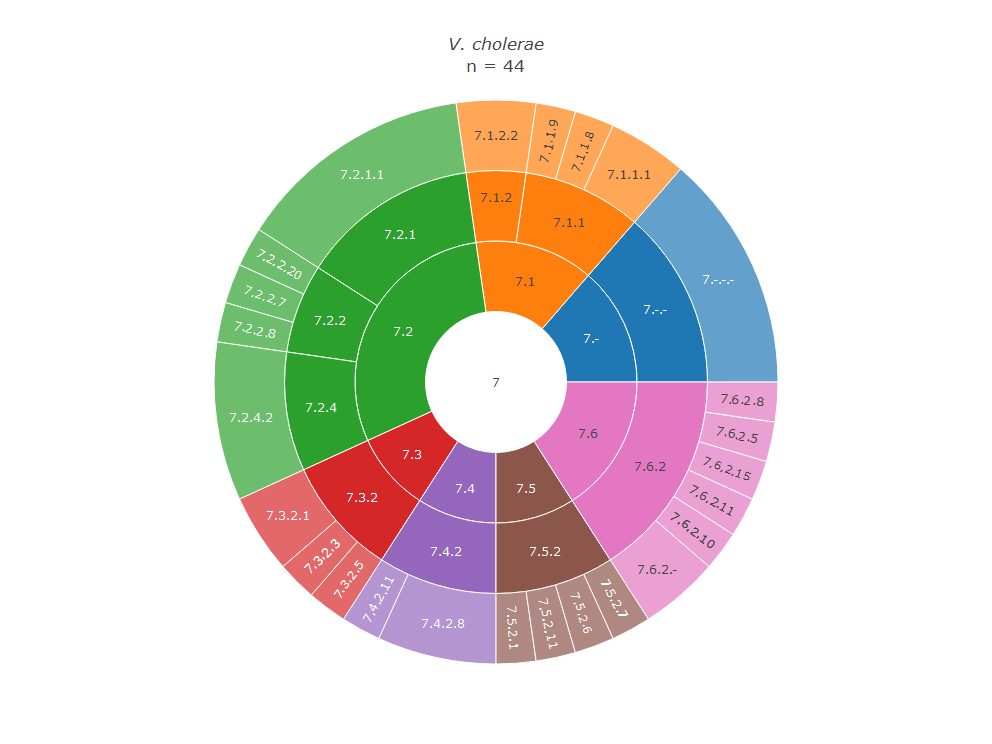
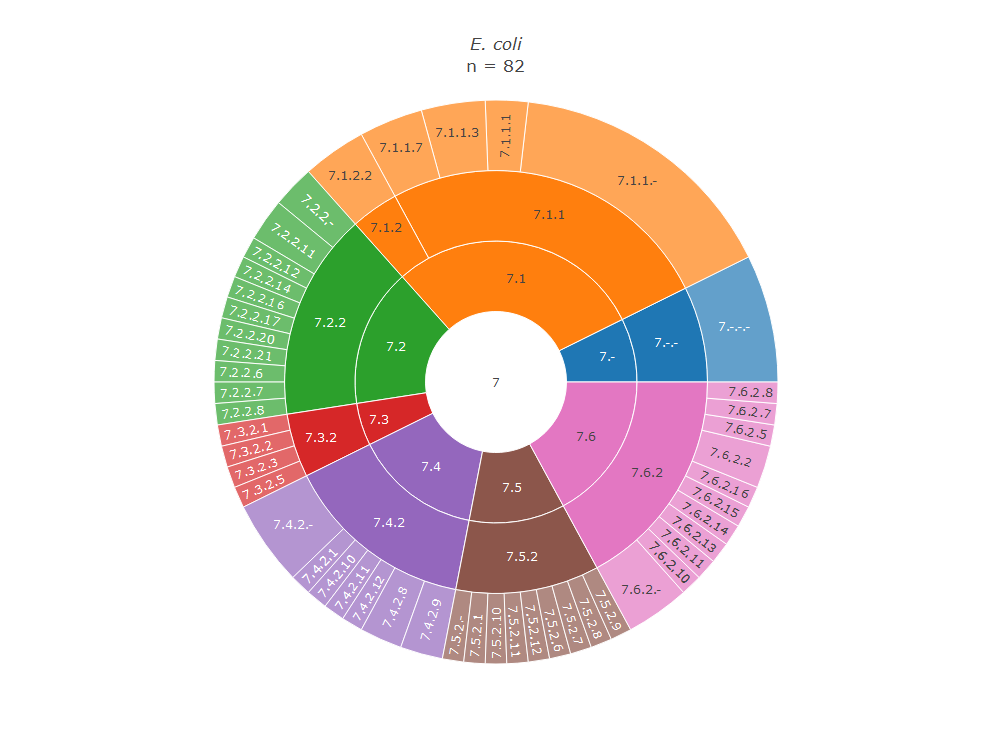
Я решил сравнить протеомы бактерий по составу имеющихся в них транслоказ (рис. 4–6, табл. 4).
| EC number | Description |
|---|---|
| EC 7.1 | Catalyzing the translocation of hydrons |
| EC 7.2 | Catalyzing the translocation of inorganic cations |
| EC 7.3 | Catalyzing the translocation of inorganic anions and their chelates |
| EC 7.4 | Catalyzing the translocation of amino acids and peptides |
| EC 7.5 | Catalyzing the translocation of carbohydrates and their derivatives |
| EC 7.6 | Catalyzing the translocation of other compounds |
| EC 7.x.1 | linked to oxidoreductase reactions |
| EC 7.x.2 | linked to the hydrolysis of a nucleoside triphosphate |
| EC 7.x.3 | linked to the hydrolysis of a diphosphate |
| EC 7.x.4 | linked to a decarboxylation reaction |
Можно заметить, что у Ph. gaetbulicola, в отличие от двух других бактерий, отсутствуют транслоказы, переносящие неорганические анионы. У нее вообще меньше белков аннотировано как транслоказы, поэтому, возможно, их просто не нашла автоматическая аннотация. Ph. gaetbulicola — свободноживущая морская бактерия, поэтому, если их действительно нет, то это можно объяснить разным ионным составом сред, в которых живут рассматриваемые бактерии. Кроме этого, у E. coli отсутствуют транслоказы, переносящие катионы и катализирующие не гидролиз АТФ, а какую-то другую реакцию. У Ph. gaetbulicola и V. cholerae все такие белки — транспортеры Na+. Я не знаю, требует ли этот факт какого-то специального объяснения.
Материалы и методы
Скрипты лежат на kodomo по адресу /home/students/y22/bakhsv/term2/pr8.
В папке pr8/outlier лежат файлы TSV, скачанные из UniProt Proteomes, и скрипт protein_count.r, считающий процентили и порогове значение для присвоения статуса Outlier.
В папке pr8/CC лежат файлы CC.txt, CC.py и CC.r. СС.py создает несколько файлов, из которых скрипт CC.r берет данные и рисует картинки (рис. 2). CC.r на kodomo не запускается, потому что нет нужных пакетов R. CC.txt содержит список возможных полей и нужен для работы CC.py (в нем нет ничего интересного).
Скрипты KW_transmembrane.py, enzymes.py и secreted.py считают доли белков из соответствующих функциональных групп. enzymes.py выводит результат в текстовый файл, остальные — в терминал.
В папке sunburst лежат скачанные из UniProt файлы со списками транслоказ ((proteome:X) AND (ec:7*)) и скрипт sunburst.r, который тоже не запускается на kodomo из-за отсутствия пакетов.
Список литературы
- Reen, F.J.; Almagro-Moreno, S.; Ussery, D.; Boyd, E.F. The genomic code: Inferring Vibrionaceae niche specialization. Nat. Rev. Microbiol. 2006, 4, 697–704, doi:10.1038/nrmicro1476.
- Machado, H.; Gram, L. Comparative genomics reveals high genomic diversity in the genus Photobacterium. Front. Microbiol. 2017, 8. doi:10.3389/fmicb.2017.01204.
- Sampaio, A.; Silva, V.; Poeta, P.; Aonofriesei, F. Vibrio spp.: life strategies, ecology, and risks in a changing environment. Diversity 2022, 14, 97, doi:10.3390/d14020097.
- Kim, Y.O.; Kim, K.K.; Park, S.; Kang, S.J.; Lee, J.H.; Lee, S.J.; Oh, T.K.; Yoon, J.H. Photobacterium gaetbulicola sp. nov., a lipolytic bacterium isolated from a tidal flat sediment. Int. J. Syst. Evol. Microbiol. 2010, 60, 2587–2591, doi:10.1099/ijs.0.016923-0.