Практикум 15
Сборка генома de novo
Чтения, с которыми я работал: https://www.ebi.ac.uk/ena/browser/view/SRR4240356.
1. Подготовка чтений
В скачанном из базы данных файле исходно было 7 511 529 чтений. Я записал все адаптеры в один файл и запустил TrimmomaticSE, чтобы их удалить:
TrimmomaticSE SRR4240356.fastq.gz removed_adapters.fastq.gz ILLUMINACLIP:adapters.fa:2:7:7
После этого осталось 7 358 438 чтений, то есть остатками адаптеорв было 2,04 % чтений. Затем запустил TrimmomaticSE еще раз, чтобы убрать некачественные нуклеотиды с конца:
TrimmomaticSE removed_adapters.fastq.gz trimmed.fastq.gz TRAILING:20 MINLEN:32
Было отброшено еще 4,15 % от оставшихся чтений, всего получилось 7 053 346.
2. Получение k-меров
Создал директорию velv/ и запустил velveth:
velveth velv/ 31 -fastq.gz -short trimmed.fastq.gz
Программа создала два текстовых файла в директории velv: Roadmaps и Sequences
3. Получение графа де Брёйна и контигов
Запустил программу velvetg. На вход этой программе подается адрес директории с результатом работы velveth, и, опционально, разные настройки, но я оставил все дефолтными. На всякий случай записал в файл stdout и stderr.
velvetg velv/ &> velvetg_outerr
Программа создала в том числе файлы contigs.fa и stats.txt. В stats.txt содержатся характеристики полученных узлов графа. По умолчанию, чтобы узел графа стал контигом, он должен быть не короче 2k. В contigs.fa содержатся последовательности получившихся контигов.
Я посмотрел в файл stats.txt. Во-первых, оказалось, что есть один узел графа длиной 1 (т.е. ни с чем не объединившийся 31-мер) с огромным покрытием. Если отсортировать этот файл по покрытию, то первые несколько значений выглядят так:
266951 1134 442 429 421 ...
На первый взгляд выглядит так, будто остался необрезанный адаптер. Наверное, можно было бы попробовать триммировать чтения еще раз, снизив пороги сходства с адаптером (хотя они, вроде, и так низкие...) В любом случае, так как эта последовательность слишком короткая, она не попала в сборку. Если оставить только те узлы графа, которые проходят по длине, то значения покрытий выглядят так (рис. 1):
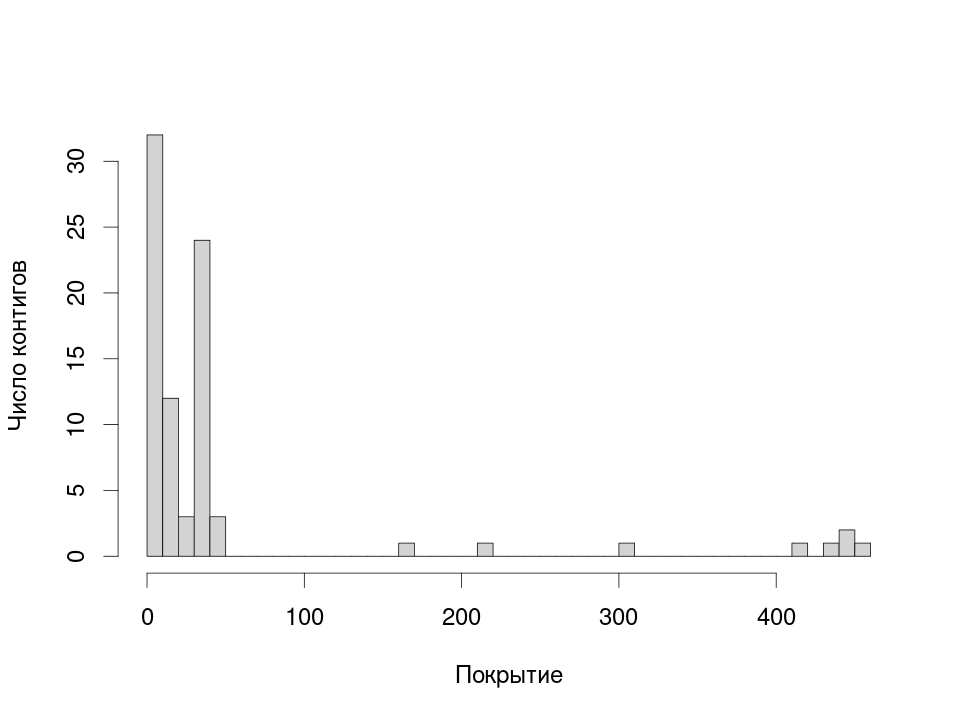
Медианное покрытие равно 14. Видимо, восемь контигов с покрытием больше 100 можно считать "аномальными". Длина этих контигов — 72–980 b.p. Первое объяснение, которое приходит в голову — это повторы, поэтому их чтений и должно быть больше. Чтобы это проверить, выровнял один из этих контигов на геном, а в качестве контроля взял контиг похожей длины с покрытием 12 (рис. 2, 3).
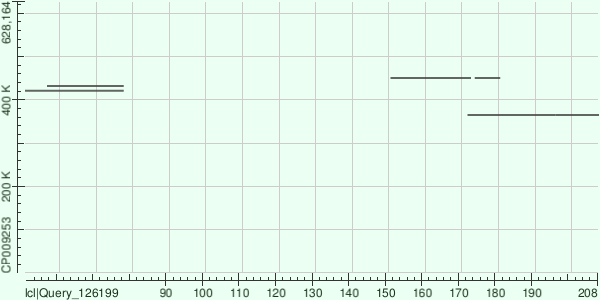
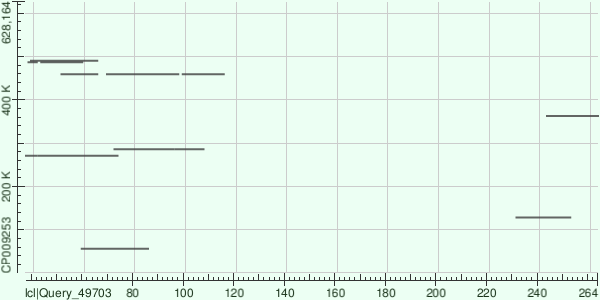
Ну, это объяснение провалилось. Возможно, это особенности фрагментации генома при подготовке бибилиотеки, или еще что-то такое — другие возможные объяснения проверить так же просто не получится.
4. Анализ
Самые длинный получившийся контиг — узел графа № 8, 111 992 b. p. длиной. Выровнял этот контиг на геном (CP009253) с помощью megablast, параметры оставил дефолтными (в т.ч. длина слова — 28). Всего нашлось 16 выравниваний. Дотплот приведен на рис. 4.
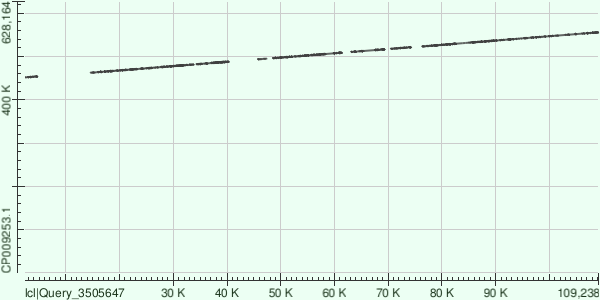
Как видно, эти 16 выравниваний не противоречат друг другу. Это выравнивания разных фрагментов контига, которые помещают его в одной и той же ориентации в одно и то же место генома. Между ними, видимо, находятся более вариабельные участки, которые не нашлись алгоритмом.
Начало первого выравнивания из шестнадцати — нуклеотид 2390 контига, конец последнего — 109 238, т.е. между ними лежит 95,4 % длины контига. Оставшиеся 5,6 % — это концы, которые не выровнялись. Кроме того, в промежутках между этими выравниваниями находится еще 19,7 % длины контига. Всего в этих выравниваниях 16 054 мисматча и 2092 гэпа. Понятно, что участки контига и хромосомы между нашедшимися выравниями тоже гомологичны друг другу, поэтому, если бы хотелось сравнить геном со сборкой лучше, можно было бы сделать глобальное выравнивание контига с соответсвующим регионом хромосомы.
Для следующих двух самых длинных контигов аналогичная информация приведена на рис. 5 и 6 и в табл. 1.
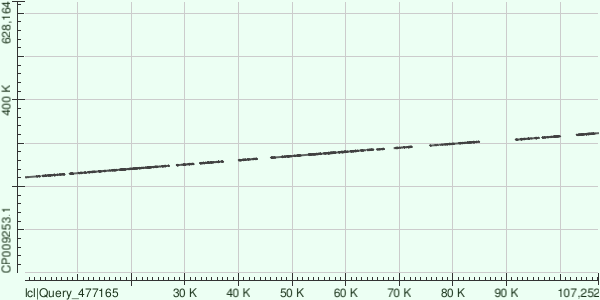
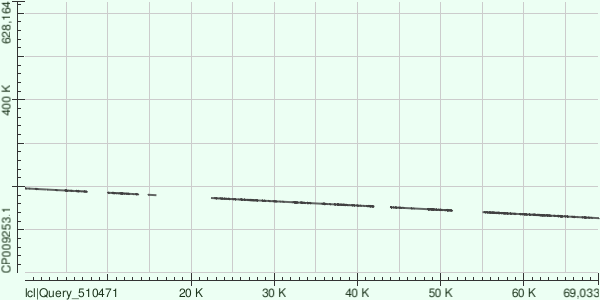
| Номер контига | Длина контига | Количество нашедшихся выравниваний | Начало первого выравнивания (контиг) | Конец последнего выравнивания (контиг) | Начало первого выравнивания (хромосома) | Конец последнего выравнивания (хромосома) | Доля контига, покрытая выравниваниями, % | Мисматчи | Гэпы |
|---|---|---|---|---|---|---|---|---|---|
| 8 | 111 992 | 16 | 2390 | 109 238 | 451 729 | 555 905 | 75,71 | 16 054 | 2092 |
| 6 | 107 518 | 18 | 146 | 107 252 | 220 869 | 323 043 | 78,31 | 16 294 | 2171 |
| 10 | 80 969 | 11 | 37 | 69 033 | 126 623 | 195 400 | 61,55 | 10 414 | 1202 |