Практикум 3
A- и B-формы ДНК. Структура РНК.
Задание 1
В этом задании нужно было построить модели структур ДНК в A-, B- и Z-формах с помощью программы fiber из пакета 3DNA. Вот ссылки на соответствующие файлы: gatc-a.pdb, gatc-b.pdb, gatc-z.pdb. В задании предлагалось построить структуры ДНК с последовательностью GATC, повторенной пять раз. Для Z-формы программа fiber посторила структуру, состоящую из пяти повторов только GC, поэтому она короче — десять пар основнаний вместо двадцати.
Задание 2
В этом задании нужно было сравнить экспериментальные и смоделированные структуры ДНК.
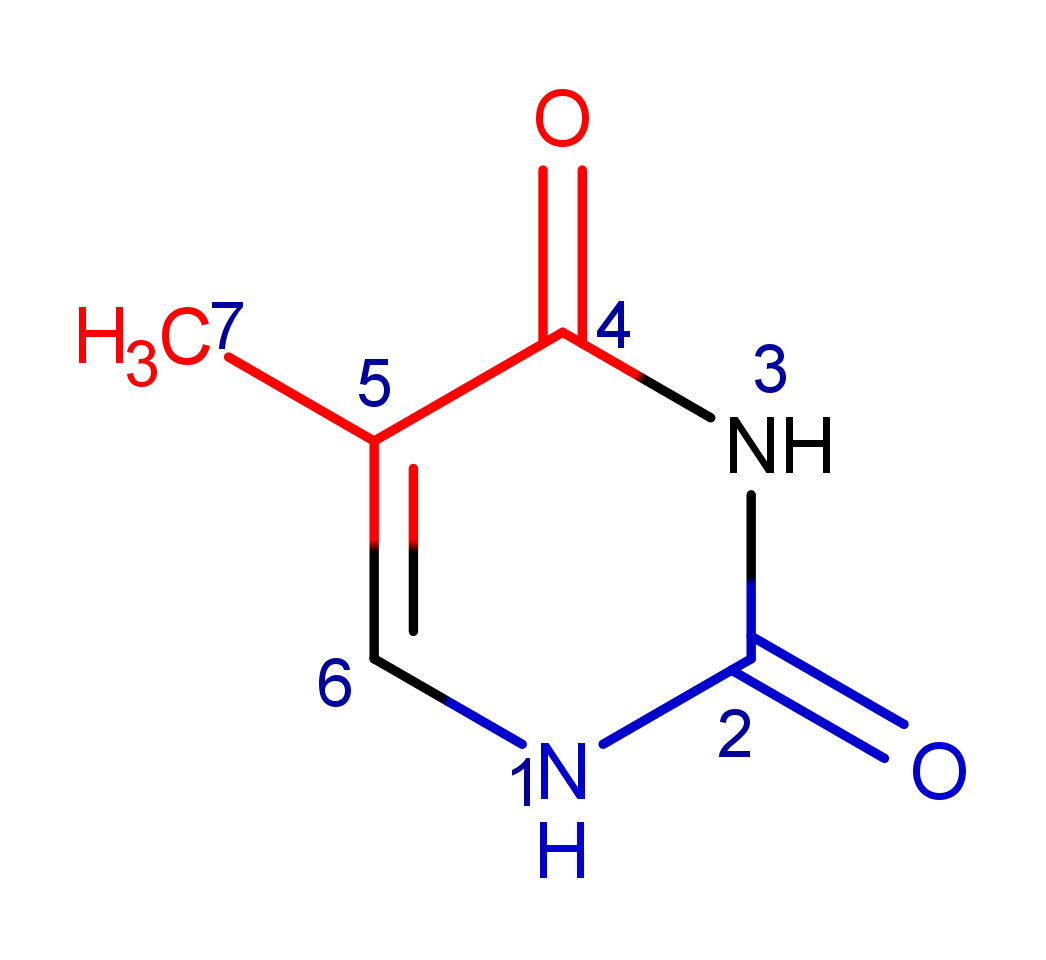
В экспериментально полученной структуре A-ДНК (PDB ID 3V9D) в сторону большой бороздки обращены атомы 4, 5 и 7 тимина, в сторону малой — атомы 1 и 2 (рис. 1). Тимин, который я рассматривал — основание 7 в структуре.
С помощью PyMol я измерил харктеритистики структур, которые требовались в этом задании (табл. 1). Для экспериментальной структуры Z-ДНК не привожу значения, так как предложенная в задании струкутра (PDB ID 1TNE) слишком короткая. Для смоделированной Z-ДНК значения измерены не для файла из предыдущего пункта, а для файла с вдвое более длинной цепочкой (gatz-z-10.pdb). Ширину бороздок я считал по определению из задания: ширина бороздки — расстояние от атома фосфора данного нуклеотида до атома фосфора нуклеотида на комплементарной цепочке, такого, что расстояние до его соседей больше, чем до него. Большой бороздкой считается более глубокая (а не более широкая).
Кроме того, программа analyze из пакета 3DNA, которую я запускал на этих файлах для выполнения следующих заданий, также считает ширину бороздок. Я решил убедиться, что я вручную посчитал ширину бороздок верно, сравнив мои значения с теми, которые вернула программа. В табл. 1 в скобках приведена усредненная ширина бороздок, посчитанная программой analyze.
| Форма ДНК | PDB ID | Тип спирали | Шаг спирали, Å | Число оснований на виток | Ширина большой бороздки, Å | Ширина малой бороздки, Å | Номер основания, для которого измерена ширина бороздок |
|---|---|---|---|---|---|---|---|
| A | 3V9D | Правая | 29,2 | 11 | 11,8 (17,6) | 15,8 (16,6) | 7 |
| A | Модель | Правая | 28,0 | 11 | 8,0 (15,2) | 16,8 (18,5) | 30 |
| B | 1BNA | Правая | 36,7 | 10 | 18,1 (17,5) | 11,1 (10,9) | 7 |
| B | Модель | Правая | 33,8 | 10 | 17,2 (17,2) | 11,7 (11,7) | 30 |
| Z | Модель | Левая | 43,5 | 12 | 9,9 (20,0) | 18,3 (9,9) | 30 |
Как видно, значения совпали только для B-ДНК. Для Z-ДНК мы с программой разошлись во мнениях, какую бороздку считать малой, а какую — большой. Для A-ДНК ширины обеих бороздок, посчитанные программой, примерно равны получившийся у меня ширине малой бороздки, хотя у меня большая бороздка вышла значительно уже.
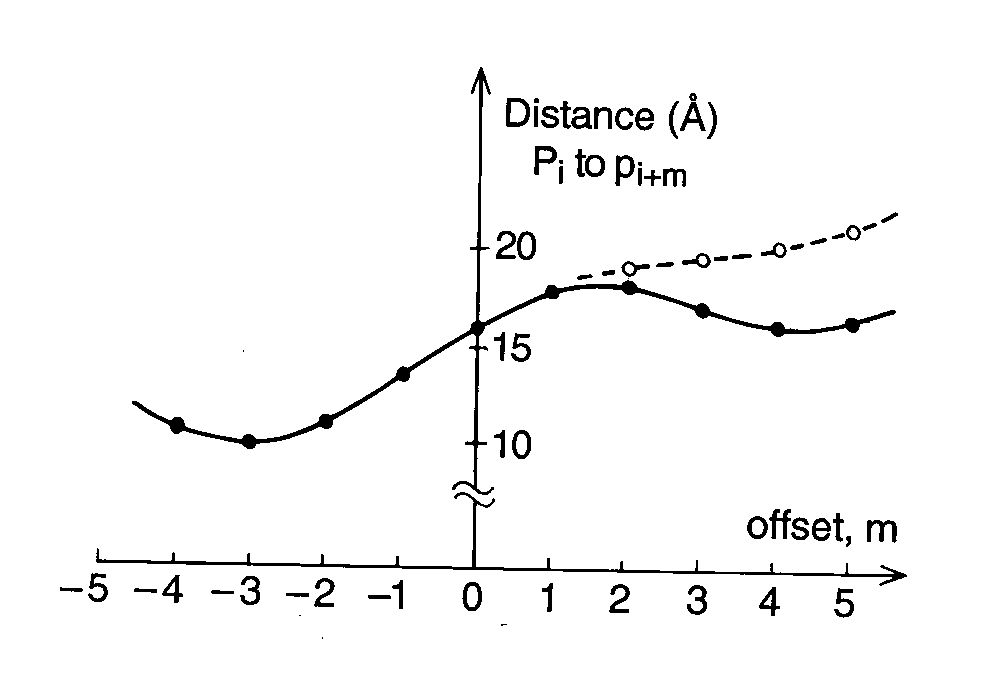
Чтобы понять, почему это произошло, я почитал статью, на которую ссылается мануал программы (Hassan, Calladine, 1998). Во-первых, авторы статьи пишут не об атомах фосфора, а о фосфатах. В статье написано, что малая бороздка всегда явственно выражена — расстояние от данного фосфата до фосфата нуклеотида, расположенного на другой цепи на три позиции ближе к 5'-концу, чем комплементарный данному, всегда меньше, чем до соседних (рис. 2). А вот минимум расстояний, соответствующий большой бороздке, хотя обычно находится на четыре нуклеотида ближе к 3'-концу, чем комплементарный данному нуклеотид, может находиться и в другом месте, либо отсутствовать вовсе. Поэтому в качестве определения ширины большой бороздки авторы взяли расстояние до фосфата +4-го нуклеотида от комплементарного — так было удобнее для целей статьи. Как я понял, в статье речь идет только о B-ДНК.
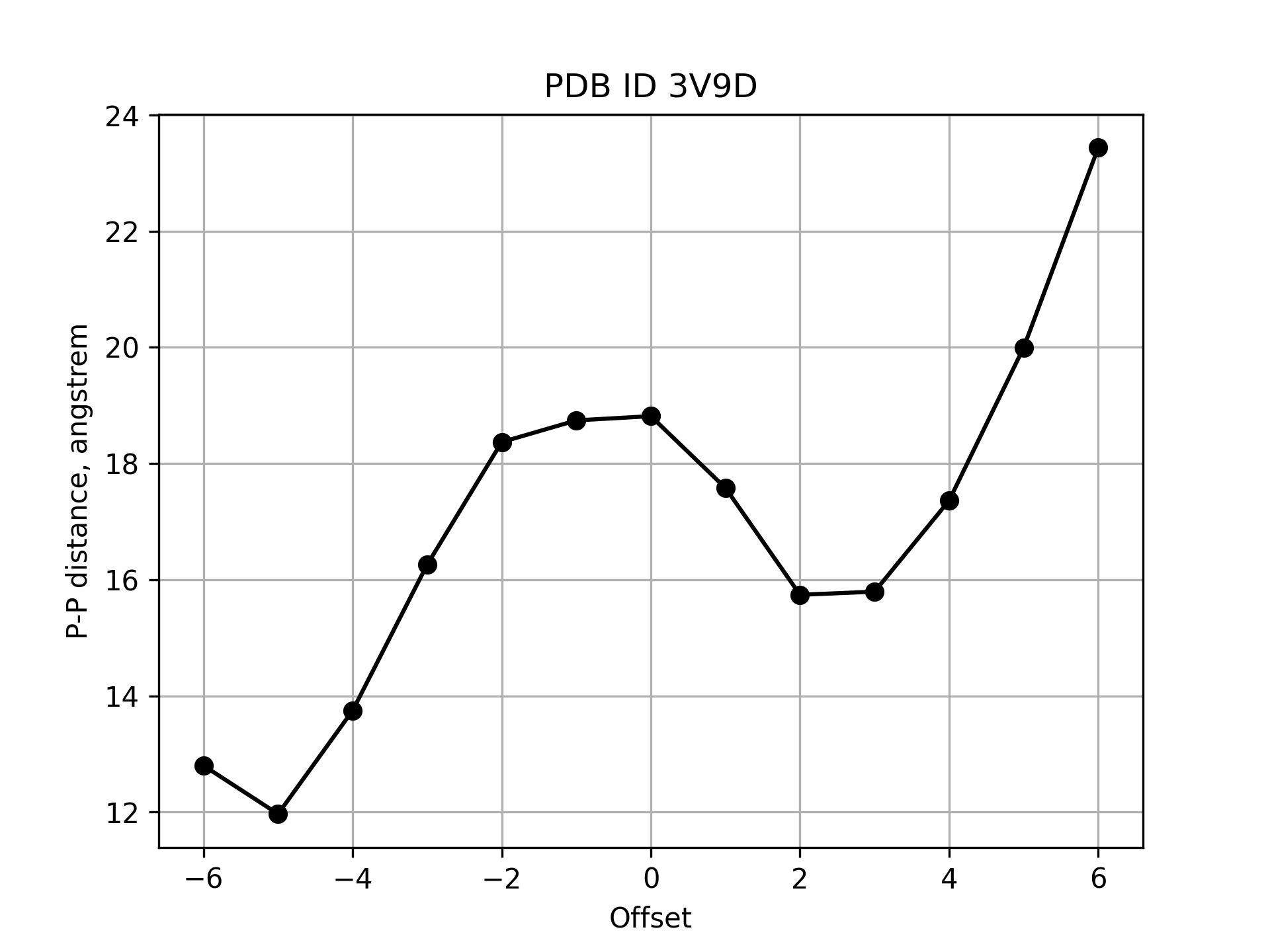
Очевидно, автор пакета 3DNA использовал это определение для любой формы ДНК. Поэтому для B-ДНК измерения ширины бороздок совпали, а для остальных форм — нет. Кажется, будто такое определение малоосмысленно для A- и Z-ДНК (рис. 3).
Я написал скрипт, считающий ширину бороздок по определению, предложенному в задании (лежит по ссылке; описание см. в комментариях внутри).
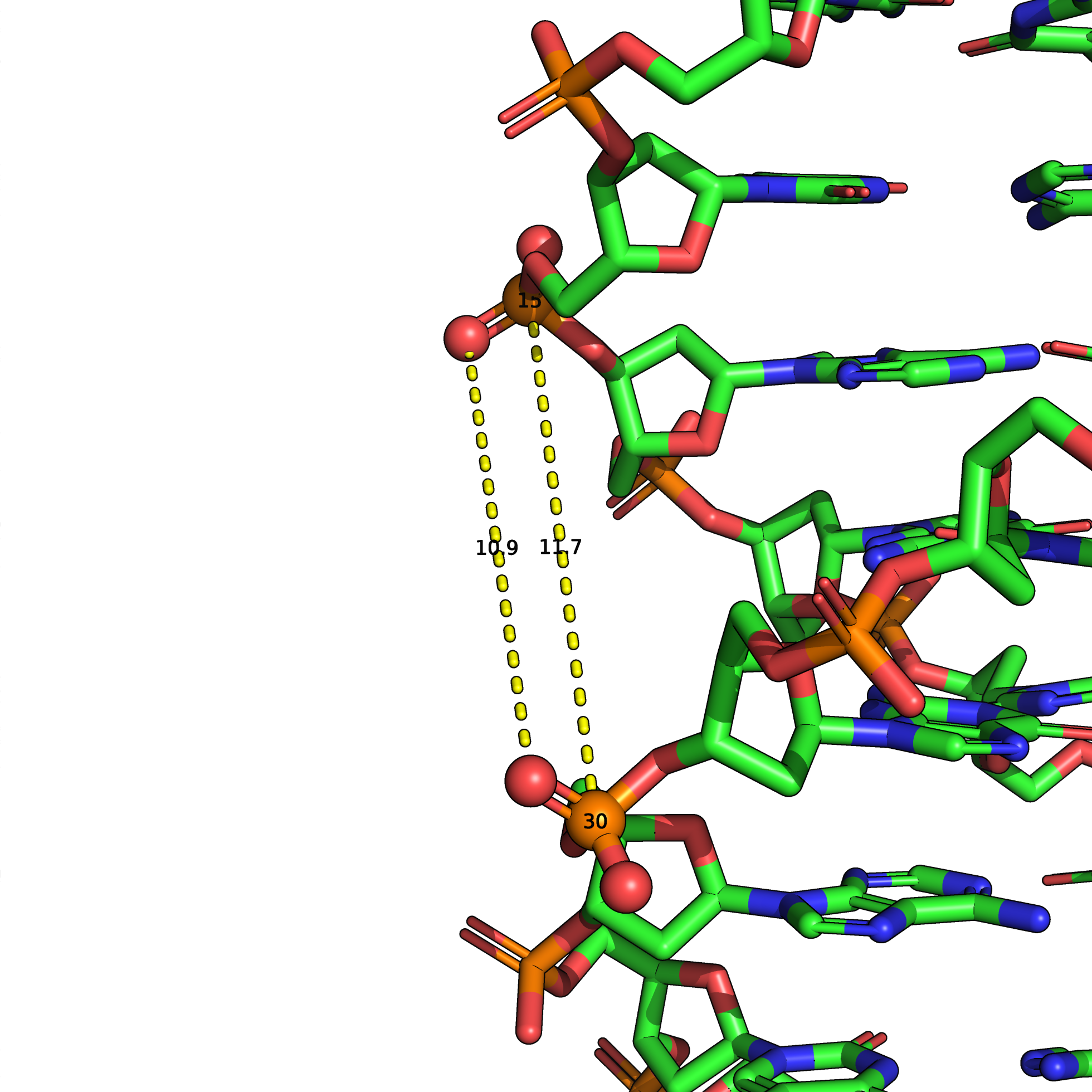
Кстати, кроме этого, видимо существует и путаница между "фосфором" и "фосфофатом". Статья, на которую ссылается мануал 3DNA, пишет о расстоянии между фосфатными группами. При этом программа analyze, очевидно, определяет расстояние между атомами фосфора: если посмотреть на структуру, видно, что оно совпадает с определенной программой шириной бороздки, но в фосфатной группе есть атомы кислорода, расположенные ближе, то есть, если считать ширину бороздки в полном соответсвии с определением из статьи, оно будто бы должно быть меньше (рис. 4).
Задание 3
Упражнение 1
В этом упражнении предлагалось сравнить торсионные углы в тРНК (PDB ID 1GTR) с торсионными углами разных форм ДНК.
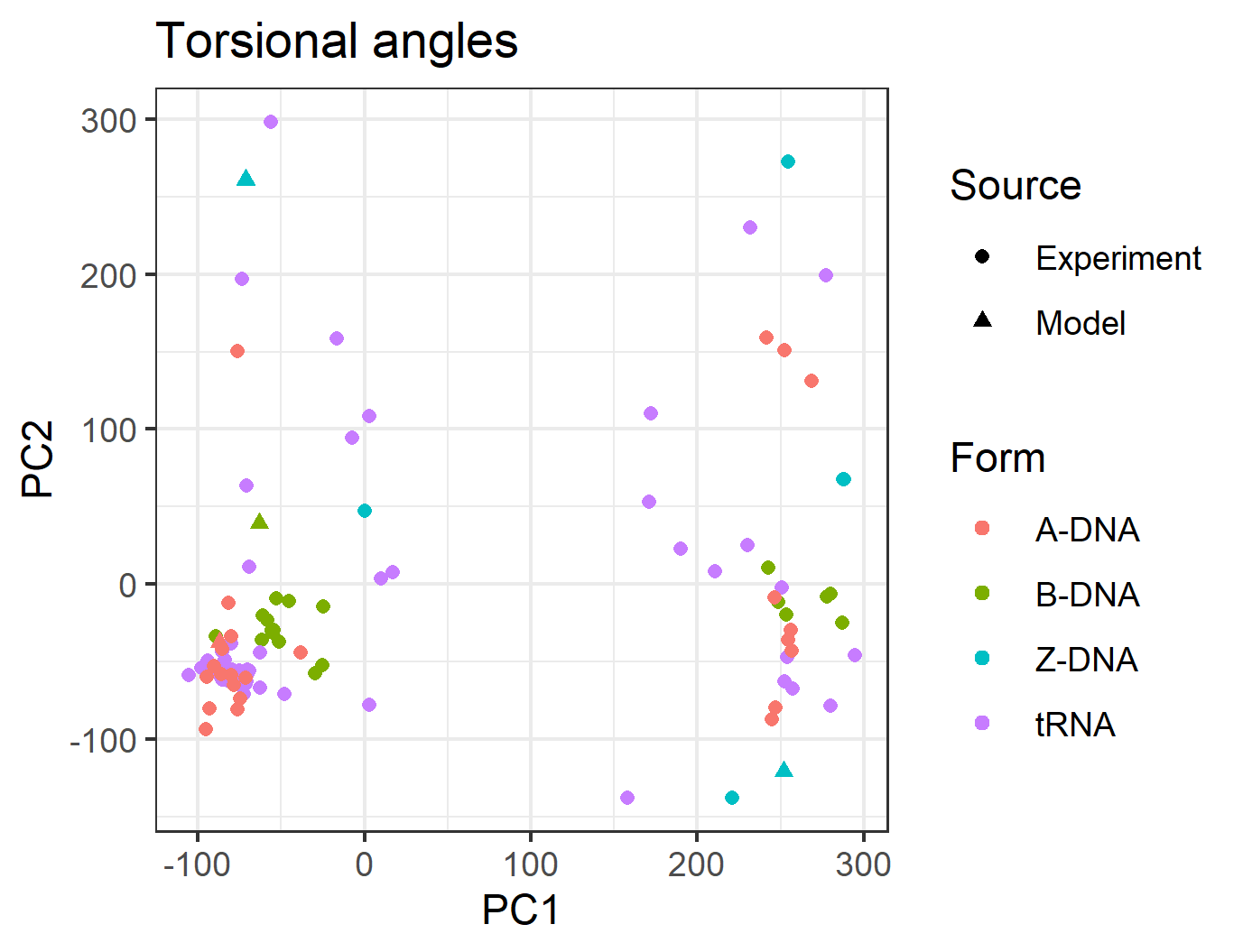
Торсионные углы посчитала программа analyze. Всего в нуклеотиде определяют семь торсионных углов. Чтобы изобразить различия между формами ДНК наглядно, я спроецировал семимерные данные на плоскость методом главных компонент (скрипт) (рис. 5).
Видно, что большинство нуклеотидов находится в кластере с левом нижнем углу картинки. Я думаю, эти нуклеотиды составляют типичную спираль, а меньшее количенство разбросанных точек — это нуклеотиды в изгибах спирали, вблизи концов и т.п. Видно, что тРНК больше всего напоминает A-форму ДНК.
Упражнение 2
В этом упражнении нужно рассмотреть вторичную структуру предложенной тРНК.
Вот координаты стеблей этой тРНК (один столбик — это стебель; через три дефиса указаны комплементарные пары в начале и в конце стебля):
2---71 49---65 39---31 10---25 . . . . . . . . . . . . . . . . 7---66 53---61 43---27 12---23
В структуре есть неканонические пары оснований. Они приведены в табл. 2.
| Основание 1 | Основание 2 |
|---|---|
| U 54 | A 58 |
| U 55 | A 18 |
| A 37 | U 33 |
| U 38 | U 32 |
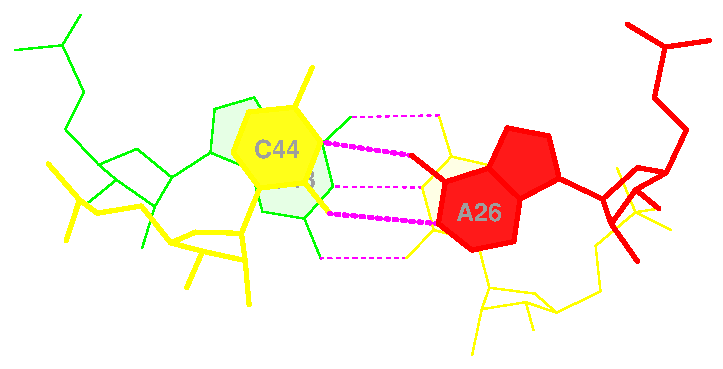
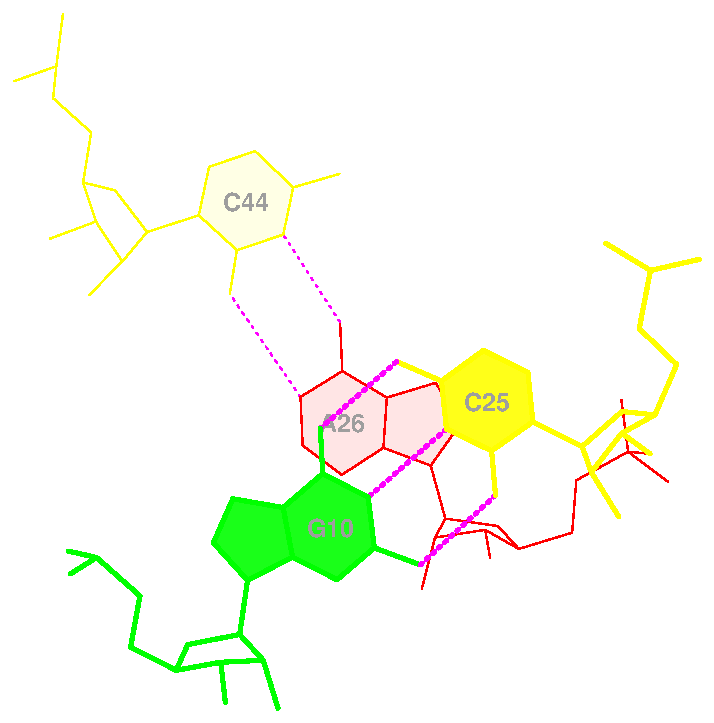
| C 44 | A 26 |
| A 13 | A 45 |
| A 14 | A 21 |
| G 15 | C 48 |
В структуре есть пары взаимодействующих оснований, не входящие в стебли ("дополнительные водородные связи"). Такие пары оснований приведены в табл. 3.
| Основание 1 | Основание 2 |
|---|---|
| U 54 | A 58 |
| U 55 | A 18 |
| A 37 | U 33 |
| U 38 | U 32 |
| C 44 | A 26 |
| A 13 | A 45 |
| A 14 | A 21 |
| G 15 | C 48 | G 19 | C 56 |
Упражнение 3
В этом упражнении нужно было получить изображения для иллюстрации стекинг-взаимодействий с помощью программы stack2img пакета 3DNA для тРНК из предыдущего упражнения. На рис. 6 приведено изображения для динуклеотидной пары с максимальной суммарной площади перекрывания проекций азотистых оснований, на рис. 7 — с минимальной ненулевой такой площадью.
Если честно, из задания и подсказок непонятно, как это помогает выполнить главную часть в упражнении, т.е. "научиться находить возможные стекинг-взаимодействия". Видимо, предполагается, что большая площадь перекрывания говорит о возможном стекинг-взаимодействии. Но насколько большая? И какой, например, площадь перекрывания должна быть при параллельном стекинге со смещением? Непонятно, как именно мы должны найти возможные стекинг-взаимодействия, использую площадь перекрывания азотистых осонований.
Список литературы
- M. A. El Hassan, C. R. Calladine. Two Distinct Modes of Protein-induced Bending in DNA. J. Mol. Biol. (1998) 282, 331–343.