Практикум 11
Домены и профили
Я выбрал домен PF00030 — бета-гамма кристаллин. Кристаллин — это водорастворимый белок, слагающий прозрачные части глаз животных (но, кажется, это его не единственная функция). Я взял двухдоменную архитектуру PF00030-PF05433, второй домен — это "глициновая молния". В этой архитектуре 132 белка.
Я выровнял fasta-файл с ними, используя Muscle with defaults из Jalview (последующие выравнивания я делал так же). Я обрезал выравнивание с краев, чтобы оно совпадало с началом первого и концом второго домена у репрезентативного представителя архитектуры, B9TBH6. Выровнял еще раз, убрал несколько последовательностей, которые были слишком короткими и не имели целой последовательности какого-то из доменов. Затем убрал последовательности с больше 90% идентичности. Вот получившееся после этого выравнивание. Затем собрал fasta-файл из последовательностей, не вошедших в это выравнивание.
В качестве контроля я взял архитектуру PF00030-PF00030-PF00652, т.е. два домена кристаллина и потом рициноподобный домен. В Pfam есть 36 белков с такой архитектурой.
Запустил на kodomo следующие программы:
hmm2build hmm2build_out crystallin-rick_cut_redund_removed.fasta hmm2calibrate hmm2build_out hmm2search --cpu=1 hmm2build_out leftout.fasta > search_positive.txt hmm2search --cpu=1 hmm2build_out crystallin-ricin_raw.fasta > search_negative.txtВот получившиеся файлы: оставшиеся белки с той же архитектурой (44 штуки), контроль.
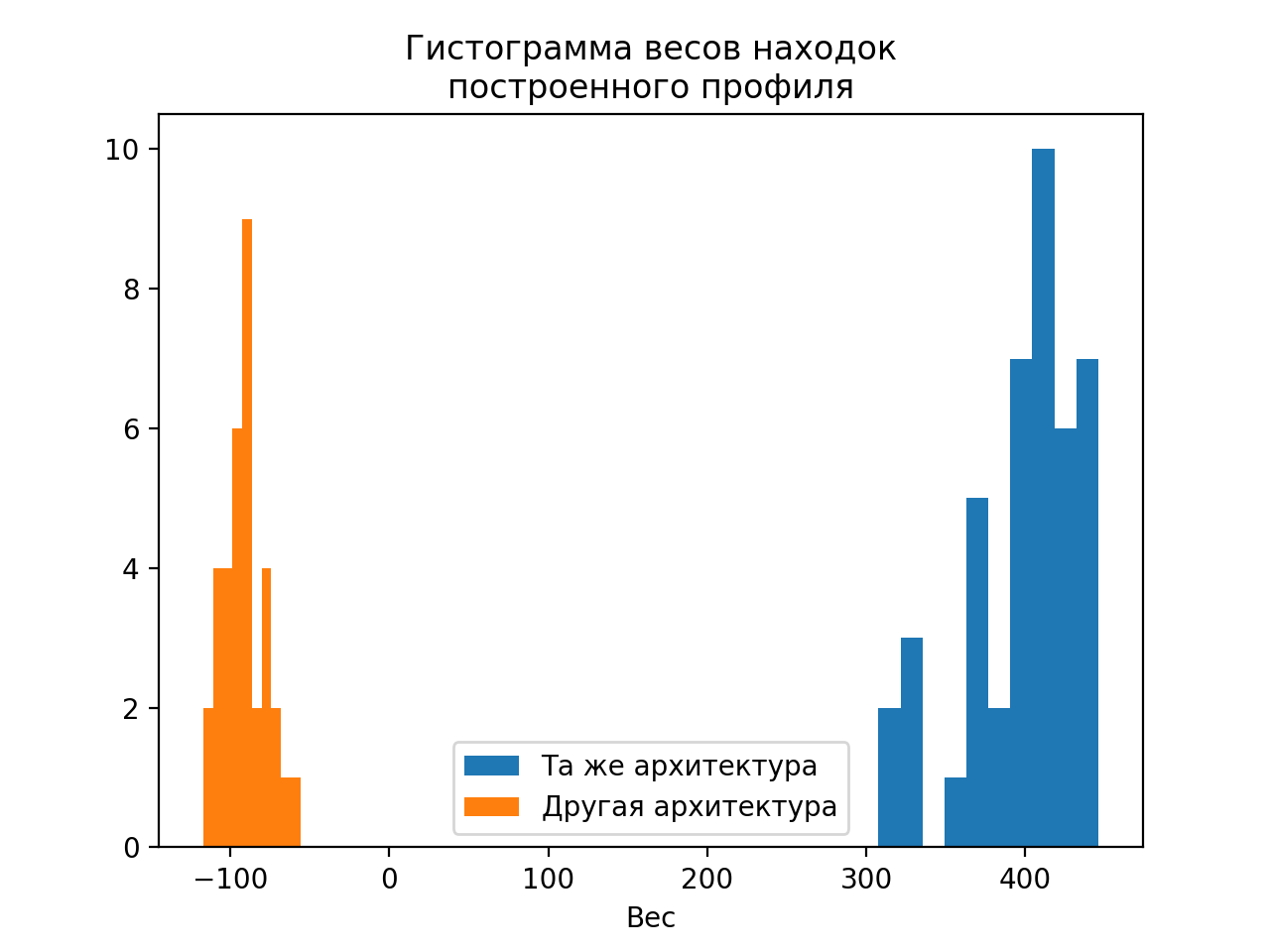
Затем я построил гистограмму весов находок (рис. 1)
Как видно, веса не пересекаются, причем с очень широким гэпом, т.е. построенный таким образом профиль позволяет точно различить архитектуры.
В задании, кроме того, предлагается построить кривые ROC и F1, но в этом случае это, очевидно, избыточно, потому что при любом разумном значении порога (например, 0) и F1, и "accuracy" будут равны единице.