Практикум 4
Задание 1
Поиск гомологов CLPX_ECOLI
Подготовил базу данных из протеомов выбранных бактерий и запустил blastp с длиной слова 2 и E-value 0,001. Выбрал формат выдачи «6» (таблица без заголовка). Вот файл.
Задание 2
Реконструкция и визуализация дерева
По найденным белкам построил дерево, используя параметры, указанные в задании. Вот файл с деревом.
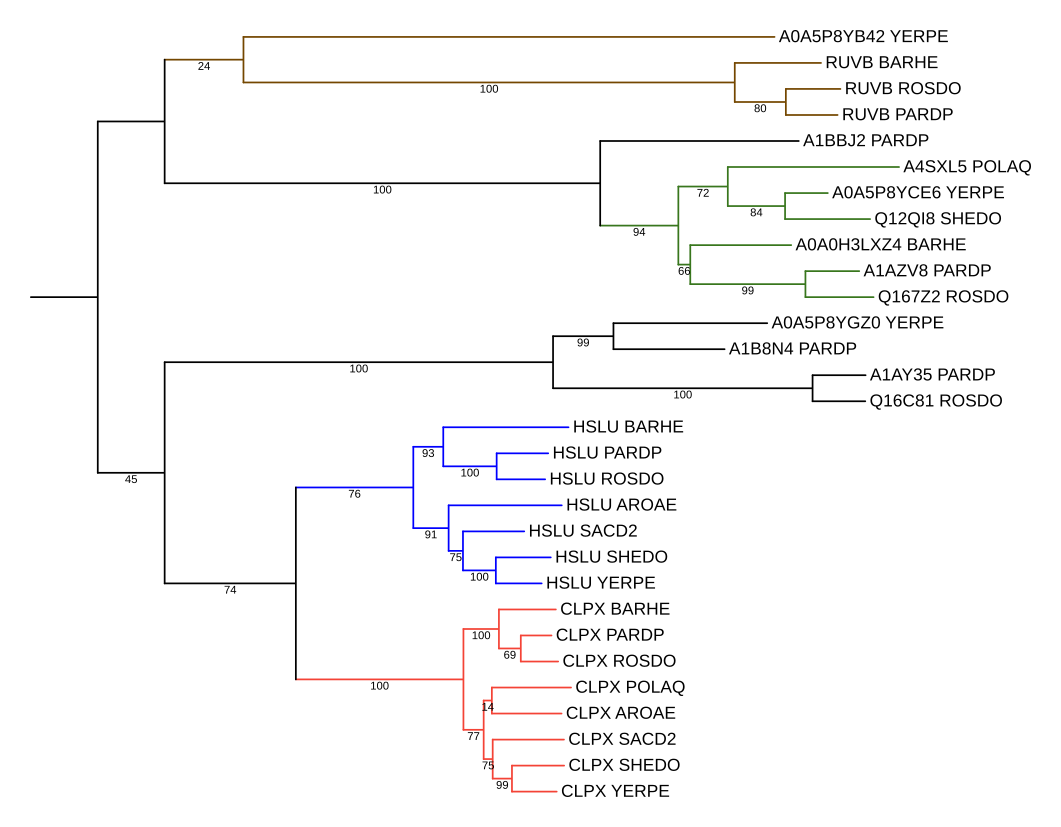
В задании требуется указать три пары паралогов и три пары ортологов, считая дерево реконструированным верно. Можно выбрать, например, такие:
Ортологи: CLPX_BARHE и CLPX_PARDP; HSLU_BARHE и HSLU_PARDP; A4SXL5_POLAQ и Q12QI8_SHEDO.
Паралоги: CLPX_BARHE и HSLU_BARHE; RUVB_BARHE и HSLU_BARHE; A1BBJ2_PARDP и A1AZV8_PARDP.
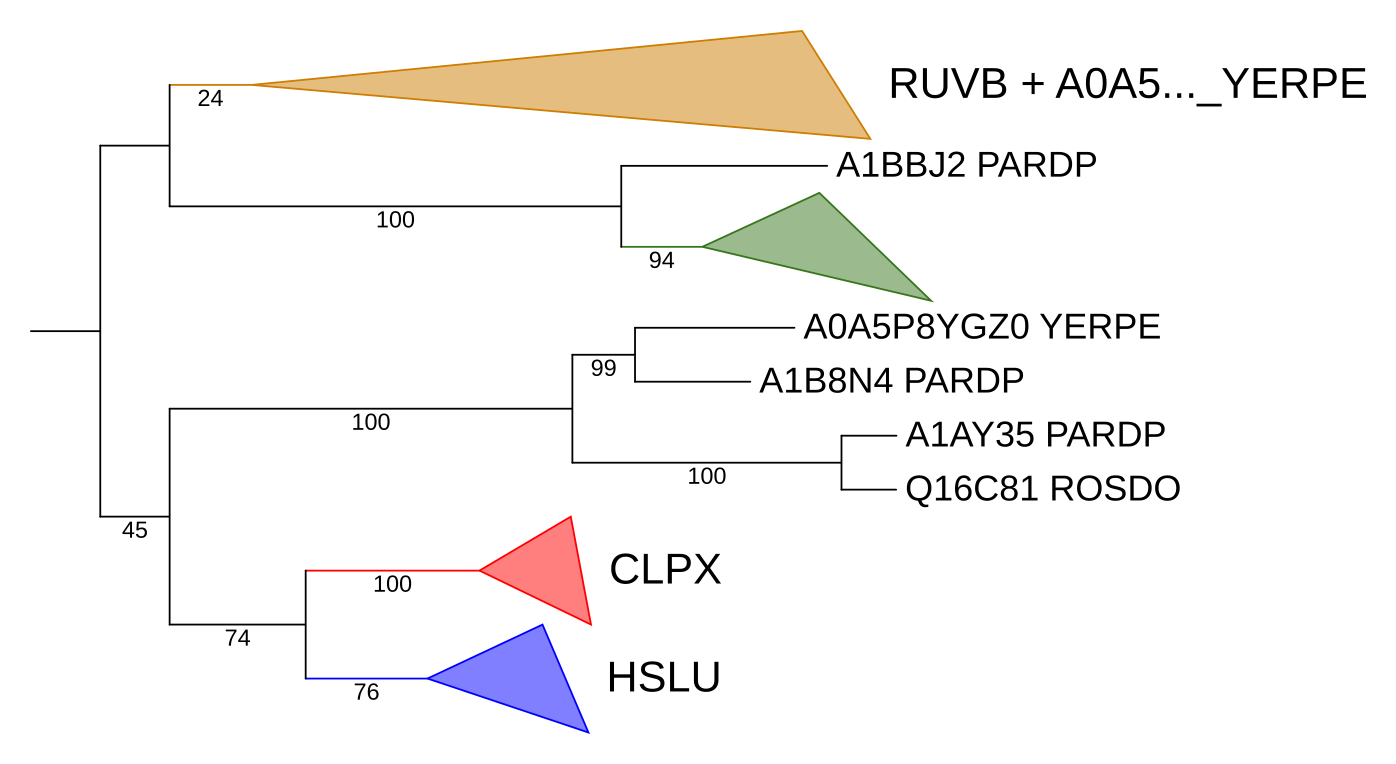
Как видно из рис. 1 и 2, на получившемся дереве есть четыре основные группы ортологов. Во-первых, это белки ClpX и HslU. Это субъединицы некоторых протеаз, очевидно, гомологичные друг другу и разделившиеся в результате дупликации у общего предка рассматриваемых бактерий. Эти белки — самые достоверные находки blast, для всех них E-value < 10−17, для остальных — E-value > 10−8. ClpX присутсвует во все бактериях, HslU — во всех, кроме Polynucleobacter asymbioticus (POLAQ). Реконструкция филогении этих белков соответствует филогении бактерий.
Третья ортологичная группа — бекли RuvB и A0A5P8YB42_YERPE. RuvB — это субъединица комплекса RuvABC, разрешающего структуры Холидея. Меня удивило, что RuvB нашелся не у всех бактерий, поэтому я просто поискал в UniProt белки с id вида RUVB_аббревиатура_моей_бактерии. Они нашлись для всех, кроме Polynucleobacter asymbioticus (POLAQ). Для нее потом нашелся тоже, просто не в SwissProt, а в trEMBL, поэтому с некрасивым id. Я добавил эти белки в fasta-файл и перестроил дерево (рис. 3).
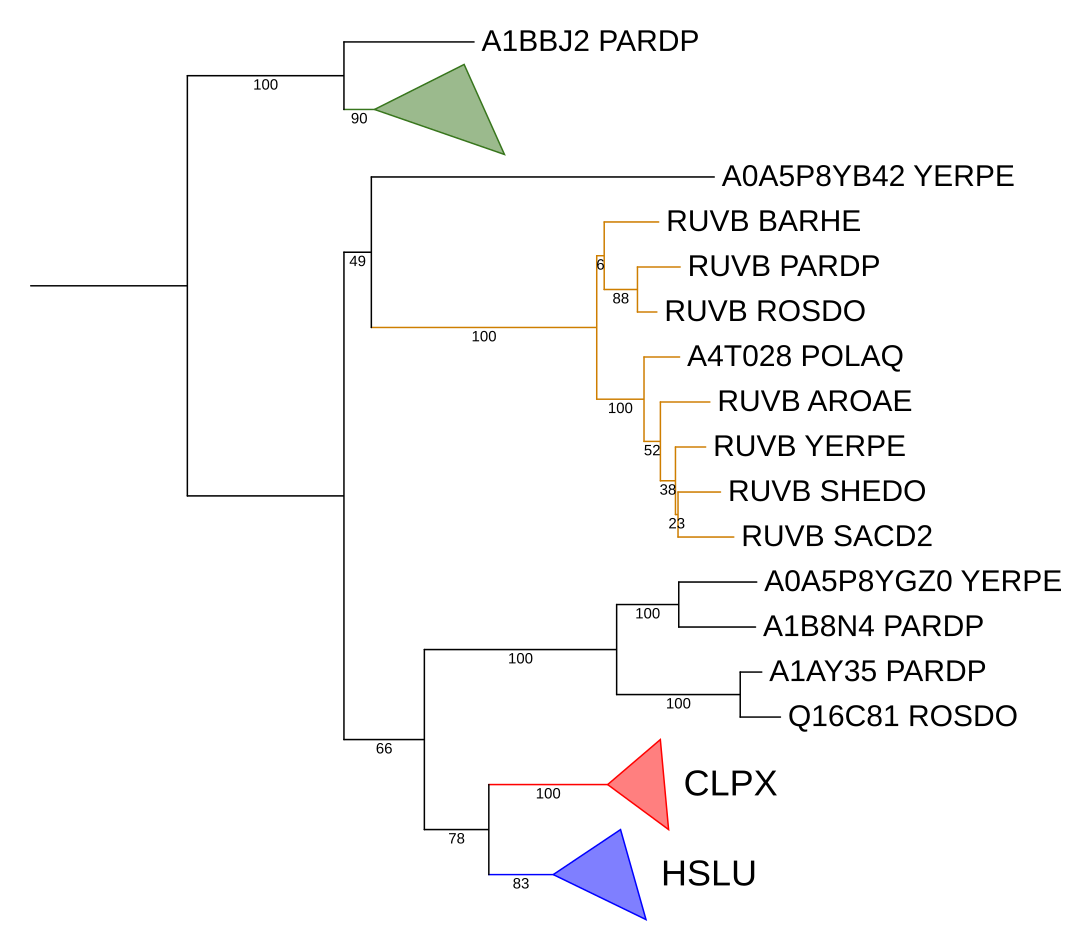
Во-первых, если мы формулируем задачу как «построить дерево гомологов ClpX E. coli, присутствующих в протеомах выбранных бактерий», то это дерево, конечно, лучше, потому что полнее. Кроме того, если мы думаем, что все нашедшиеся blast белки действительно гомологичны ClpX E. coli, то надо то же самое сделать и для других «одиноких» белков, которые не попали в группы ортологов, включающих белки из всех или почти всех бактерий, потому что некоторые находки имели E-value, близкие к указанному порогу в 0,001, а значит, какая-то часть находок, похожих по качеству и, вероятно, гомологичных, не попала в это дерево. (Но я не буду это делать).
Во-вторых, видно, что из-за неполноты дерева на предыдущем рисунке ортологичная группа указана на самом деле неправильно, т.к. A0A5P8YB42_YERPE отделился от общего предка RuvB не в результате видообразования, а в результате дупликации.
В-третьих, видно, что реконструкция филогении RuvB на этом дереве не соответсвует филогении бактерий (на рис. 2 — соответсвует).
В-четвертых, видно, что на таком дереве средняя точка попала в другую ветвь.
Четвертая ортологичная группа показана зеленым. Она содержит белки FtsH, Zn2+-зависимые протеазы. В ней нет белков из Saccharophagus degradans (SACD2) и Polynucleobacter asymbioticus (POLAQ); для остальных бактерий топология соответсвует реальной филогении.
Сопроводительные материалы
Это не есть работающий скрипт, потому что я что-то перекладывал и переименовывал вручную, но какой-то набор команд, которые я использовал. Пусть тут лежит.