Практикум 6
Задание 1
Сравнение предсказаний трансмембранных участков в бета-листовом белке
В качестве белка с трансмембранной β-бочкой я выбрал нуклеозид-специфичный порин Tsx из E. coli. Он расположен во внешней мембране и является каналом, проницаемым (только) для нуклеозидов и дезоксинуклеозидов. Варианты английского названия этого белка — bacterial nucleoside transporter Ts (OPM), nucleoside-specific channel-forming protein Tsx (UniProt), nucleoside-specific porin (Wikipedia). Идентификаторы — P0A927 в UniProt, 1TLY в PDB.
Первичная последовательность этого белка в UniProt и в PDB различается: в UniProt она на 22 аминокислоты длиннее с N-конца и на 6 короче с C-конца. Первые 22 аминокислоты в UniProt размечены как сигнальный пептид; т.к. белок для РСА выделяли из E. coli, логично, что он зрелый и сигнального пептида в нем нет. Лишние 6 аминокислот на конце — это гистидины, потому что авторы струткуры использовали 6-His таг, чтобы выделить белок (Jiqing, van den Berg, 2004). Для DeepTMHMM я решил использовать последовательность из PDB.
Трансмембранные участки, приведенные в OPM и предсказанные DeepTMHMM, перечислены в табл. 1.
| TM участок | OPM | DeepTMHMM |
|---|---|---|
| 1 | 13–22 | 14–27 |
| 2 | 34–44 | 32–43 |
| 3 | 48–57 | 48–58 |
| 4 | 77–85 | 75–86 |
| 5 | 101–110 | 96–110 |
| 6 | 122–132 | 119–131 |
| 7 | 137–146 | 139–151 |
| 8 | 163–173 | 161–171 |
| 9 | 181–189 | 181–193 |
| 10 | 215–224 | 211–222 |
| 11 | 227–235 | 227–241 |
| 12 | 262–272 | 260–270 |
Графическое представление результата работы DeepTMHMM приведено на рис. 1, текстовая выдача в формате gff лежит здесь.
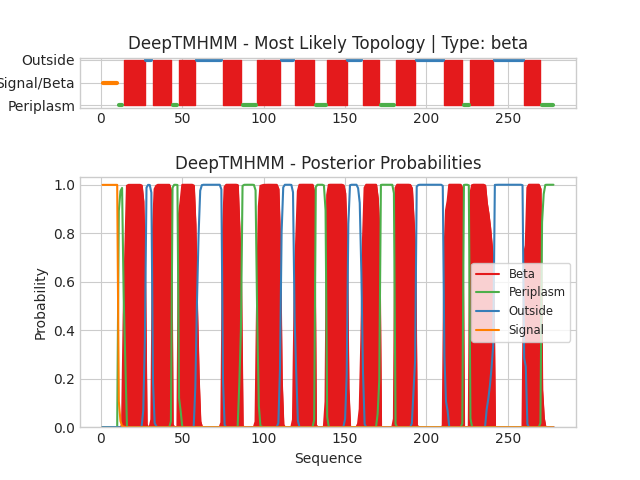
Сначала меня смутило, что на странице и в выдаче DeepTMHMM везде написано просто «Beta» или «Beta sheet», но нигде не сказано, что они имеют в виду именно трансмембранный, но потом я посмотрел в препринт (Hallgren et al, 2022), и там написано, что, конечно, подразумеваются именно такие (ну просто про трансмембранные α-спирали у них написано наоборот только «Membrane»... могли бы быть и последовательнее).
Во-первых, видно, что она нашла кусочек сигнального пептида в начале последовательности, хотя на самом деле его там уже нет. Забавно, что если подать ей всю последовательность белка, то она определит сигнальный пептид правильно (gff).
По табл. 1 видно, что DeepTMHMM нашла все трансмембранные участки, и они все хорошо перекрываются с перечисленными в OPM. Так как какие-то отличия все-таки есть, но просто глядя на цифры что-то понять сложно, я их нарисовал (рис. 2).
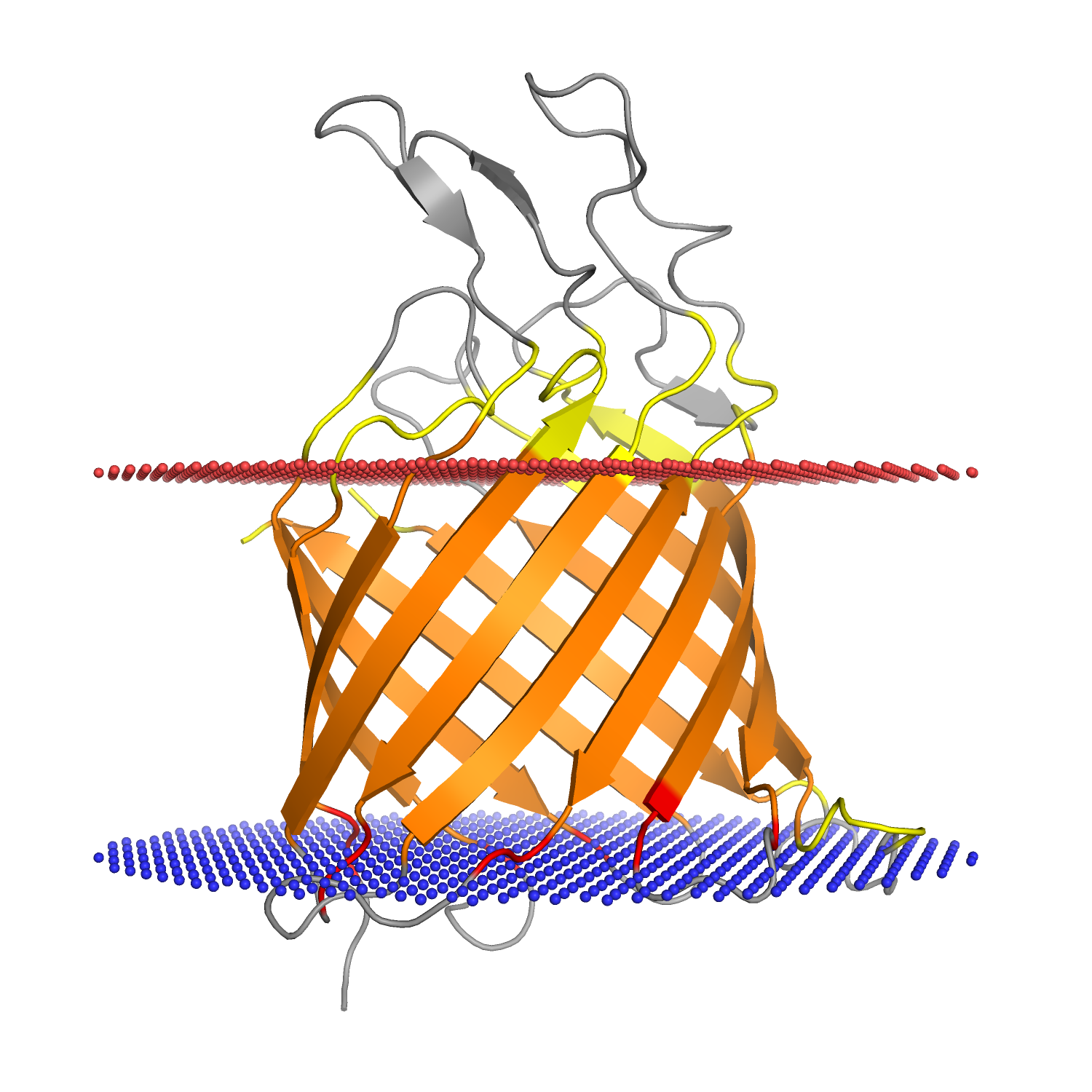
Границы трансмембранных участков, обращенные к внутренней стороне мембраны, либо совпадают у двух методов, либо находятся чуть ниже (относительно ориентации, как на рисунке) у OPM, за исключением двух кусочков петель, на рисунке справа снизу. Наружная же граница находится всегда выше у DeepTMHMM. В общем, DeepTMHMM решила, что мембрана потолще и белок утоплен в нее поглубже.
Алгоритм из OPM работает, минимизируя энергию Гиббса переноса белка из воды в мембрану, где мембрана представлена как растворитель с меняющимися в пространстве свойствами (рис. 3).

Но периплазма наверное может довольно сильно отличаться по этим параметрам от чистой воды; я не знаю, использует ли алгоритм разные модели для разных мембран (наверное использует, зачем иначе спрашивать?); не знаю, где именно проводится граница, по которой разделяются трансмембранные и не-трансмембранные участки. Но если искать причину, по которой результаты DeepTMHMM могут оказаться ближе к правде, чем OTM, я бы предположил некоторую неадекватнось используемой физической модели — что-то вроде «какие-нибудь полисахариды в периплазме образуют водородные связи не так, как вода, поэтому самое выгодное положение белка оказывается на 5 Å глубже».
К сожалению, авторы DeepTMHMM вообще ничего не написали в препринте о том, откуда взялся датасет, на котором они учили модель, поэтому что можно сказать здесь, я не знаю.
Задание 2
Сравнение предсказаний трансмембранных участков в альфа-спиральном белке
Белок, который мне выдали — «неохарактериpизованный белок MG120» из Mycoplasma genitalium (Uncharacterized protein MG120, Y120_MYCGE). Судя по нашедшемся в белке доменам, он транспортный. Там были слова «ABC transporter» и «permease», но это, кажется, противоречит друг другу, поэтому точнее сказать я не могу.
Результаты работы DeepTMHMM представлены на рис. 4, в табл. 2 и в файле.
Я запустил PPM со следующими параметрами: Type of membrane — Gram-positive bacteria inner membrane; Allow curvature — no; Topology (N-ter) — in (потому что так сказала DeepTMHMM). Результаты есть в табл. 2 и в pdb-файле.
| TM участок | PPM | DeepTMHMM |
|---|---|---|
| 1 | 32–54 | 34–55 |
| 2 | 77–100 | 84–98 |
| 3 | 105–122 | 107–122 |
| 4 | 136–162 | 138–158 |
| 5 | 166–181 | 168–185 |
| 6 | 221–240 | 222–240 |
| 7 | 270–291 | 270–290 |
| 8 | 312–324 | 314–324 |
| 9 | 329–344 | 330–341 |
| 10 | 354–376 | 358–372 |
Как видно, оба алгоритма нашли одни и те же трансмембранные участки, и они всегда почти полностью перекрываются; совпадение результатов очень хорошее. Для этого белка я не рисовал трехмерную визуализацию, поэтому ничего нового о причинах тех несущественных отличий, которые все-таки есть, сказать не могу.
