3 Результаты и обсуждения
3.1 Длины белков, закодированных в геноме бактерии Klebsiella quasipneumoniae
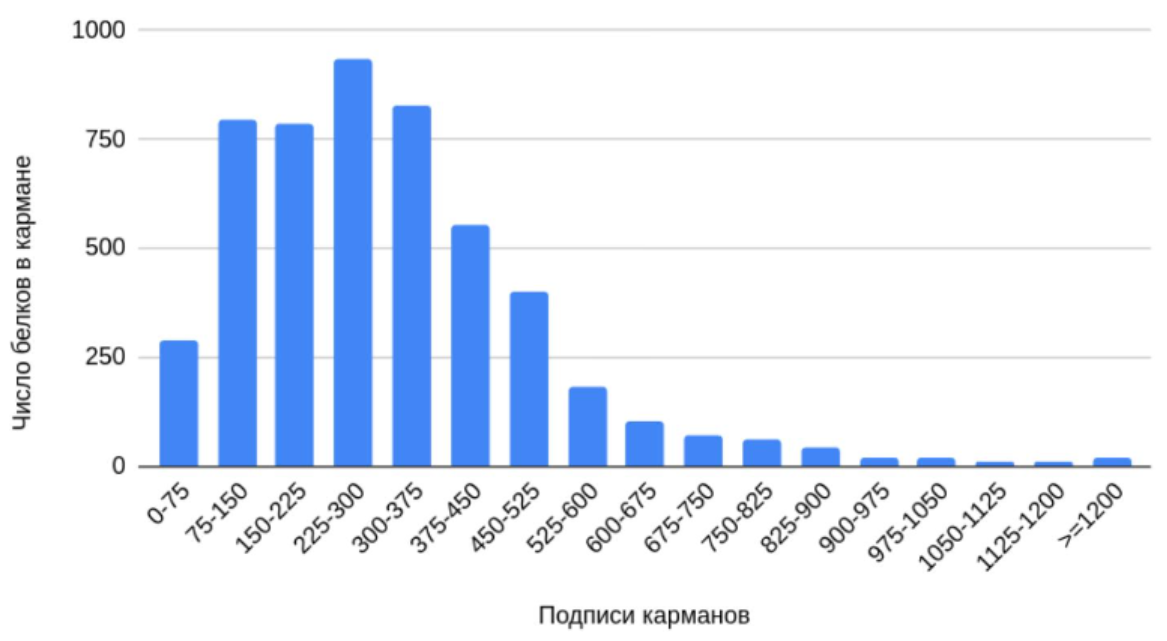
Как видно из рис 1 больше всего белков имеют длины 75-375 аминокислот, в данной бактерии присутствуют даже белки, длиной более 1200 аминокислот.
3.2 GC состав генома бактерии Klebsiella quasipneumoniae
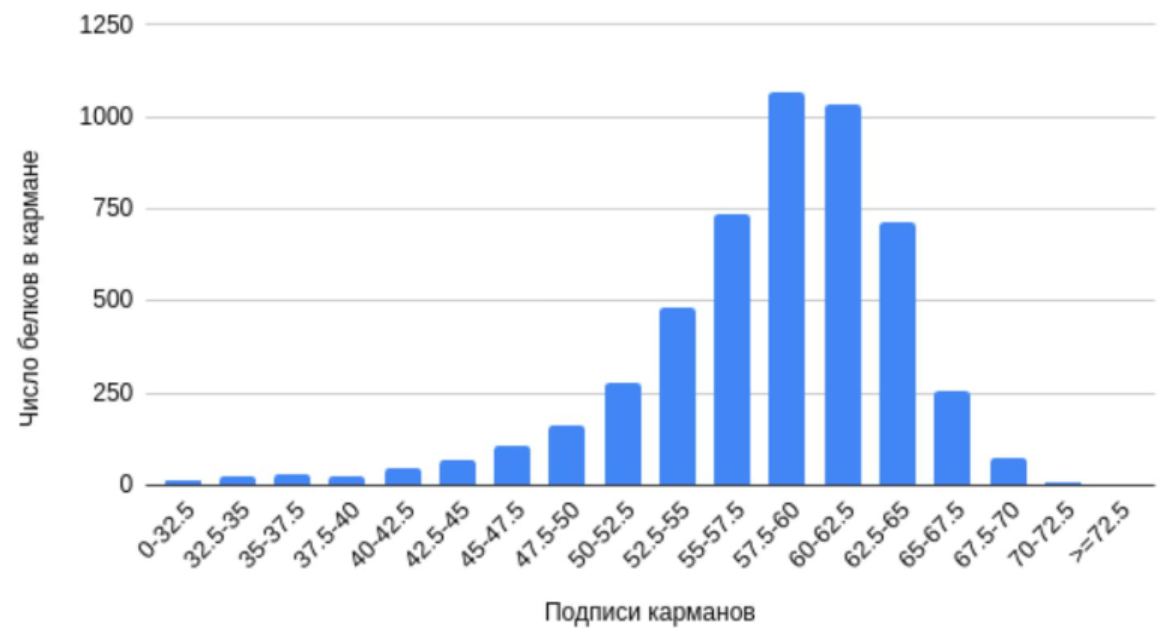
Из рис 2 можно сделать вывод, что в геноме данной бактерии в основном 57.5 - 62.5% нуклеотидов GC. Кроме того, подавляющая часть последовательностей CDS имеет процент GC больше 50.
3.3 Частоты встречаемости старт кодонов в геноме бактерии Klebsiella quasipneumoniae
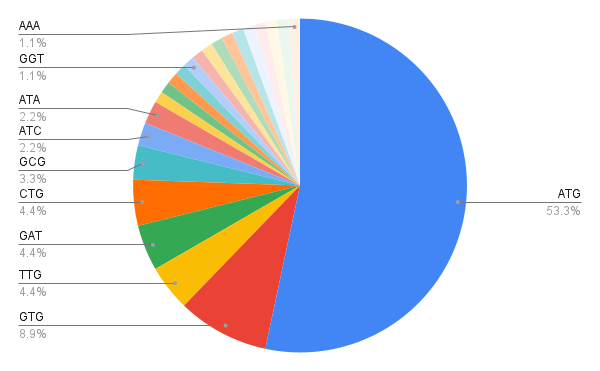
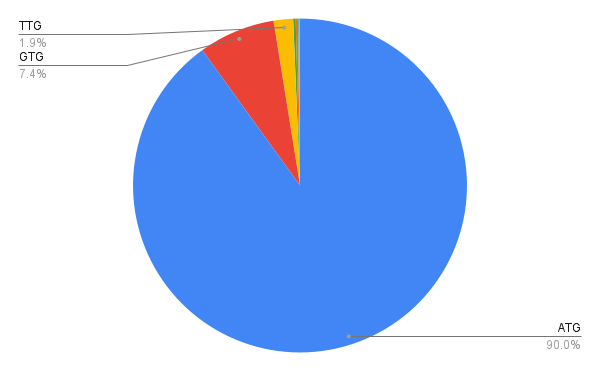
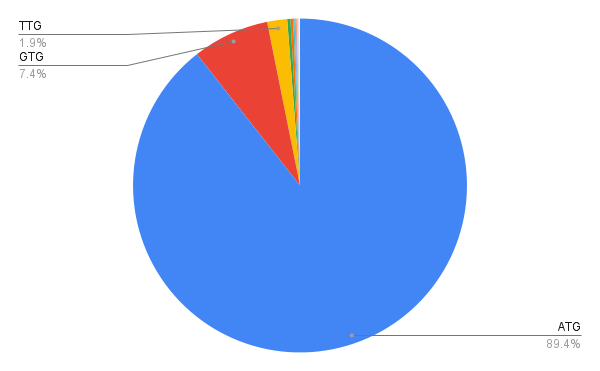
В белок кодирующих CDS гены в основном начинаются со старт кодона AUG, однако достаточно распространены также старт кодоны GTG и TTG (рис 3). В псевдогенах наблюдается гораздо большая вариативность старт кодонов (рис 4), что неудивительно, так как регуляция трансляции псевдогенов не так важна, как регуляция трансляции белок кодирующих генов. Однако, так как в геноме псевдогенов гораздо меньше, чем белок кодирующих генов, в общем рассмотрении старт-кодонов CDS все равно превалируют ATG, GTG и TTG (рис 5). Последние два являются достаточно распространенными в геномах бактерий. На пример, не каноничные старт кодоны могут компенсироваться мутациями в других инициаторных элементах, для более сильного сигнала инициации трансляции [4]. Так же другие варианты старт кодонов могут служить механизмом дополнительной регуляции [3].
3.4 Изучение генов, кодирующих металло-бета-лактамазы
Были рассмотрены гены, кодирующие один из типов карбапенемаз — металло-бета-лактамазы. Оказалось, что все 8 таких генов находятся в основном геноме, а не в плазмидах. При анализе последовательности этих генов, было обнаружено, что такие же гены присутствуют в хромосомных геномах Klebsiella pneumoniae и Klebsiella variicola (таблица 1).
| Идентификатор | Организм, где чаще всего встречается наиболее близкая к этому последовательность |
|---|---|
| WP_004205106.1 | Klebsiella pneumoniae |
| WP_064150901.1 | Klebsiella pneumoniae |
| WP_048324480.1 | Klebsiella sp. |
| WP_043519632.1 | Klebsiella pneumoniae |
| WP_167875398.1 | Klebsiella variicola |
| WP_004201404.1 | Klebsiella variicola |
| WP_025999133.1 | Klebsiella pneumoniae |
| WP_004206319.1 | Klebsiella pneumoniae |
Такое совпадение может быть получено по некоторому ряду причин. Во-первых, эти бактерии очень близкие родственники, поэтому схожесть генома неудивительна. Во-вторых, как известно, у этих бактерий очень развит горизонтальный перенос генов [7], поэтому вполне вероятно, что эти гены передаются между данными бактериями.
3.5 Изучение плазмид бактерии Klebsiella quasipneumoniae
Данная бактерия содержит 2 плазмиды. Анализ последовательностей этих плазмид показал, что они обе очень похожи на плазмиды многих образцов Klebsiella pneumoniae. Более длинная плазмида (NZ_CP065839.1 - 144959 bp) на 100% процентов совпадает с участком последовательности лишь одного образца Klebsiella pneumoniae, в остальных случаях только на 60-70%. Такие результаты могут быть обусловлены их родством или горизонтальным переносом генов, так как, как известно, данные 2 вида обмениваются плазмидами [2,7]. Также, последовательность этой плазмиды на 75% совпадает с участком генома плазмиды Escherichia coli в одном из образцов. Это достаточно неожиданный результат, который может быть вызван случайностью или горизонтальным переносом генов. Вторая плазмида короче (NZ_CP065840.1 - 3478). Она была обнаружена в последовательностях плазмид многих образцов Klebsiella pneumoniae, также не маленький процент схожести с этой последовательностью имели образцы плазмид других Klebsiella. Однако вновь наблюдается несколько неожиданных образцов в выдачи BLAST. Pseudomonas aeruginosa содержит в своем хромосомном геноме ровно такую же последовательность, а эти бактерии совершенно не родственники (Pseudomonas aeruginosa - g-proteobacteria, Klebsiella quasipneumoniae - Enterobacteriaceae). Есть информация, что устойчивость к карбапенемам встречается у Pseudomonas aeruginosa [9,13], возможно поэтому такая схожесть плазмид не случайна. Немаленький процент схожести наблюдался у нескольких образцов других Enterobacteriaceae, в том числе Escherichia coli. Вновь становится непонятно, это горизонтальный перенос или случайность.