Рис 1. Предсказанная структура.
Проанализируем структуру тРНК 1FFY.pdb с помощью программы einverted из пакета EMBOSS, которая позволяет найти инвертированные участки в нуклеотидных последовательностях.
Код:
! echo "GGGCUUGUAGCUCAGGUGGUUAGAGCGCACCCCUGAUAAGGGUGAGGUCGGUGGUUCAAGUCCACUCAGGCCCAC" > rna.seq
! einverted -sequence rna.seq -gap 12 -threshold 10 -match 3 -mismatch -3 -outfile outfile -outseq seqout
Предскажем вторичную структуру тРНК по алгоритму Зукера с помощью ViennaRNA.
На рис 1 изображена структура, которую предсказала программа. Т.к. она не выглядит как тРНК, была найдена наиболее похожая на адекватный результат структура их субоптимальных структур (рис 2).
Заполним общую таблицу о реальной и предсказанной вторичной структуре тРНК из файла 1FFY.pdb
| Участок структуры | Позиции в структуре (по результатам find_pair) |
Результаты предсказания с помощью einverted |
Результаты предсказания по алгоритму Зукера |
|---|---|---|---|
| Акцепторный стебель | 5' 1-7 3' 5' 66-72 3' |
5' 1-7 3' 5' 57-63 3' |
5' 1-7 3' 5' 67-73 3' |
| D-стебель | 5' 10-13 3' 5' 22-25 3' |
- | 5' 10-13 3' 5' 23-26 3' |
| T-стебель | 5' 49-53 3' 5' 61-65 3' |
- | 5' 50-54 3' 5' 62-66 3' |
| Антикодоновый стебель | 5' 26-32 3' 5' 38-44 3' |
5' 23-32 3' 5' 40-49 3' |
5' 28-32 3' 5' 40-44 3' |
| Общее число канонических пар нуклеотидов | 20 | 14 | 21 |
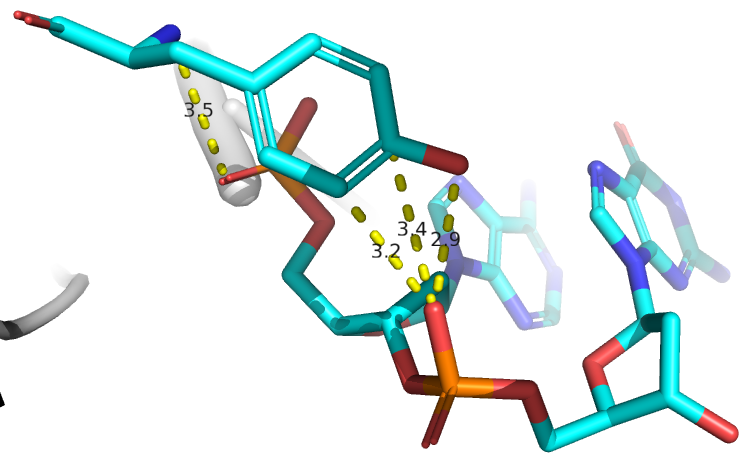
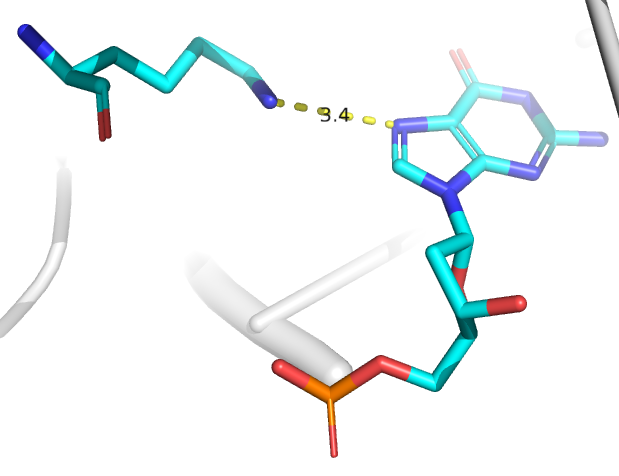
Провизуализируем в pymol структуру 1R4O.pdb (рис 3, 4, 5, 6, 7)
Скрипт



Опишем ДНК-белковые контакты в заданной структуре 1R4O.pdb. Сравним количество контактов разной природы.
Полярные атомы: кислород и азот
Неполярные атомы: углерод, фосфор и сера
Назовем полярным контактом ситуацию, в которой расстояние между полярным атомом белка и полярным атомом ДНК меньше 3.5Å.
Аналогично, неполярным контактом будем считать пару неполярных атомов на расстоянии меньше 4.5Å.
Скрипт
| Контакты атомов белка с | Полярные | Неполярные | Всего |
|---|---|---|---|
| остатками 2'-дезоксирибозы | 1 | 12 | 13 |
| остатками фосфорной кислоты | 11 | 19 | 30 |
| остатками азотистых оснований со стороны большой бороздки | 4 | 7 | 11 |
| остатками азотистых оснований со стороны малой бороздки | 0 | 1 | 1 |
Как видно из таблицы, преобладают неполярные контакты с фосфатным остовом и дезоксирибозой, что может указывать на преобладание гидрофобного характера связывания белка с ДНК.
С помощью программы nucplot получили популярную схему ДНК-белковых контактов для 1R4O.pdb (рис 8, 9).
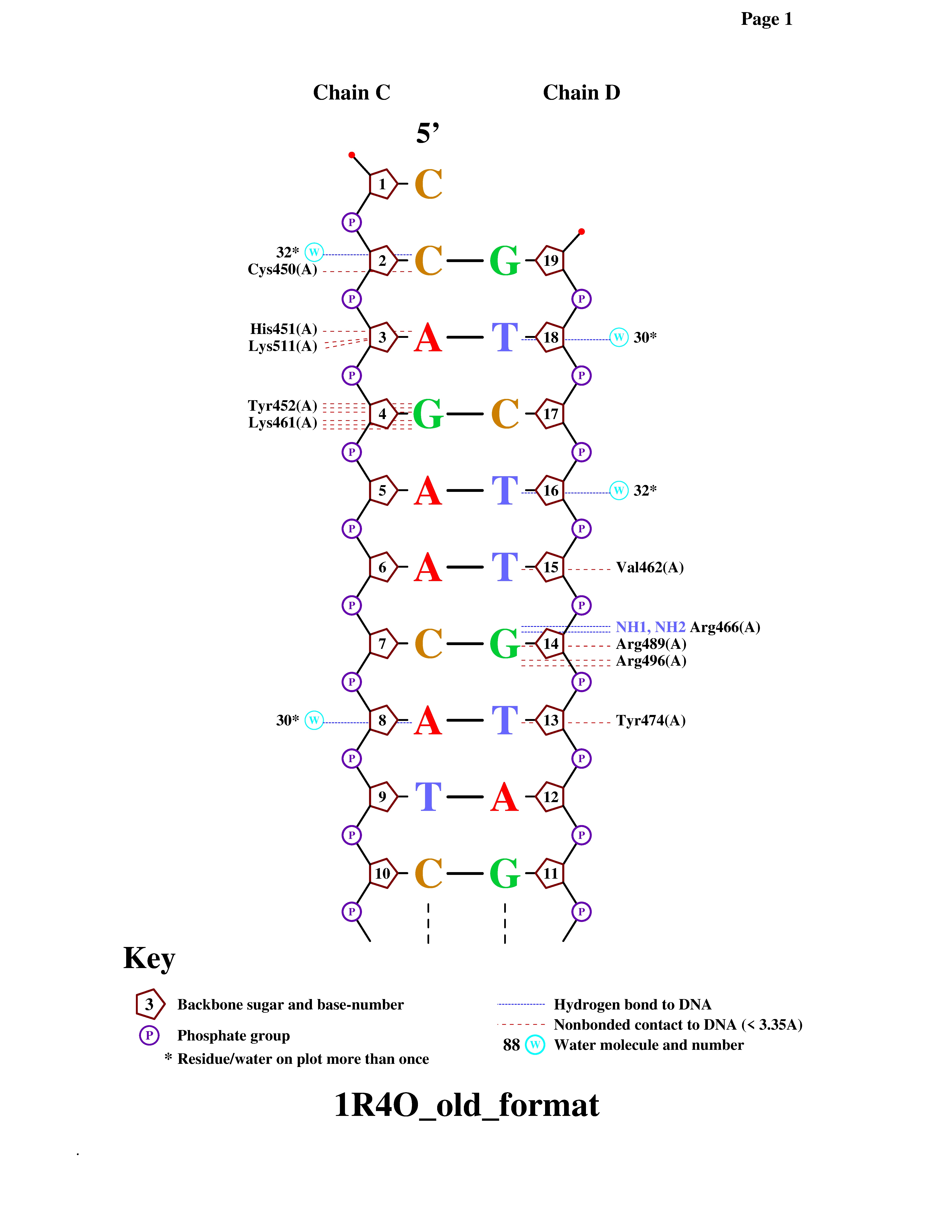
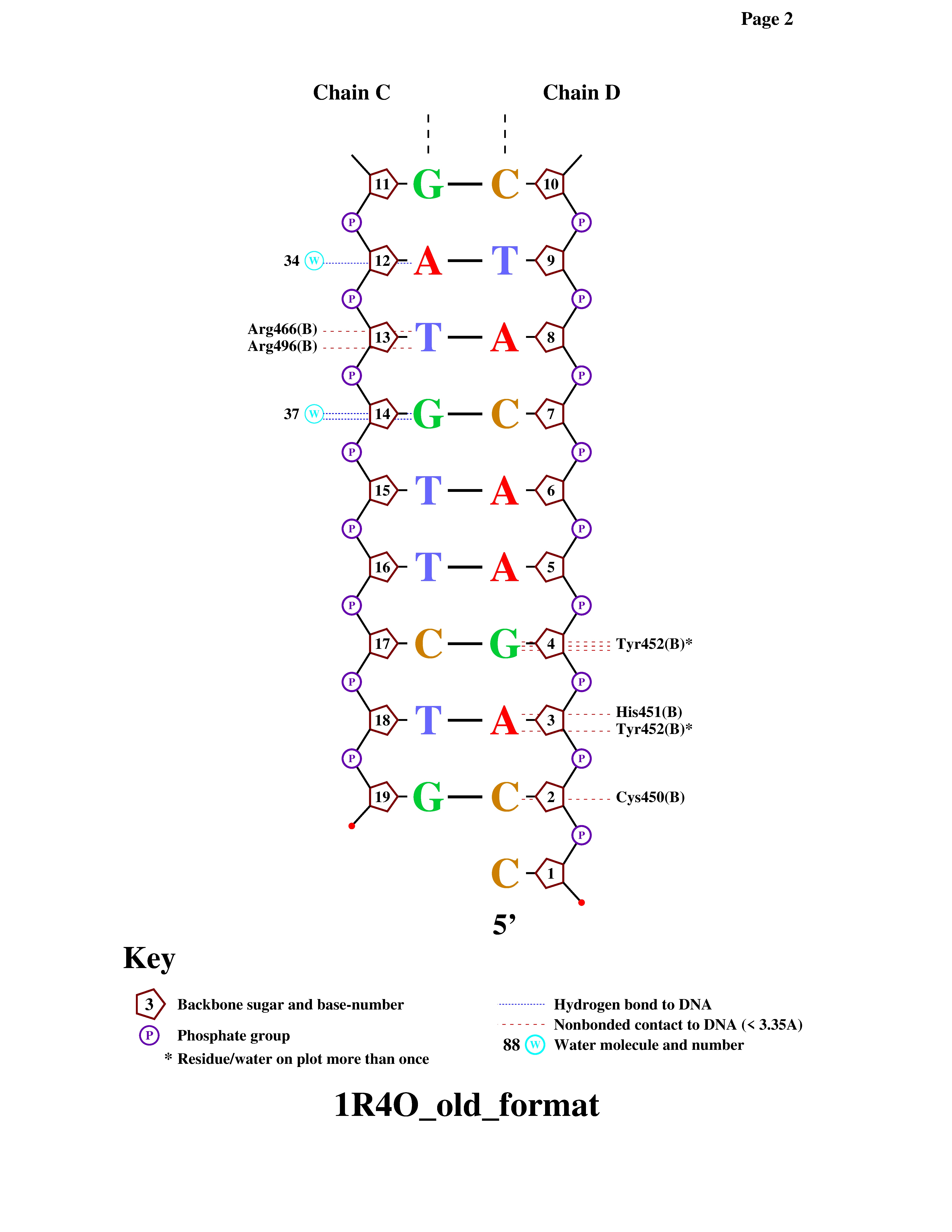
Как мы видим из рис 8 и 9, аминокислотные остатки с наибольшим числом указанных на схеме контактов с ДНК: Tyr452, Lys461.
Аминокислотный остаток, наиболее важный для распознавания последовательности ДНК: Tyr452 (т.к. этот остаток имеет наибольшее число водородных связей и связывается напрямую с азотистым основанием).
Покажем в pymol выбранные остатки (рис 10, 11).