В файле с последовательностями белков, который мы скачале в прошлом практикуме (GCF_003254725.2_ASM325472v2_protein.faa), найдем белок, который аннотирован как δ-субъединица АТФ-синтазы. Он имеет идентификатор XP_025312481.1, сохраним его последовательность отдельно в fasta файл.
Найдем идентификатор нуклеотидной записи, к которой относится ген этого белка. Для этого в файле с аннотацией (GCF_003254725.2_ASM325472v2_genomic.gbff) проведем поиск по названию белка и по строчке "LOCUS". Найдем последний LOCUS до идентификатора нашего белка. Получаем локус NC_064262, который находится на 20ой хромосоме.
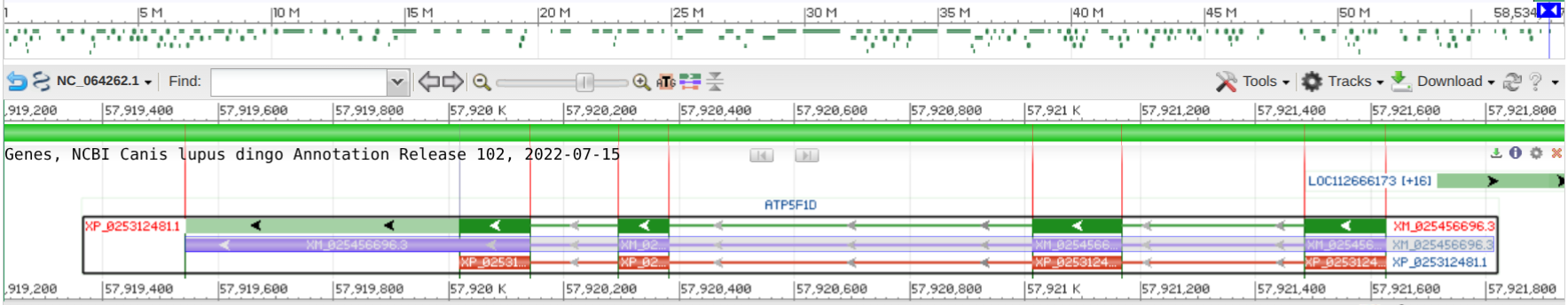
Получим последовательность ДНК, которая содержит белок кодирующую часть гена δ-субъединицы АТФ-синтазы, через режим геномного браузера (рис 1). Сохраним последовательность гена с окрестностью: файл.

Для оценки консервативности кодирующей последовательности δ-субъединицы АТФ-синтазы Canis lupus dingo был проведен blastn и tblastx.
Для сравнения я выбрала таксон Пчелы (Apoidea), т.к. он достаточно сильно удален от нашего организма. Поэтому провести поиск BLAST по последовательностям из этого таксона для последовательности белок-кодирующей области δ-субъединицы АТФ-синтазы нашего организма, будет интересно.
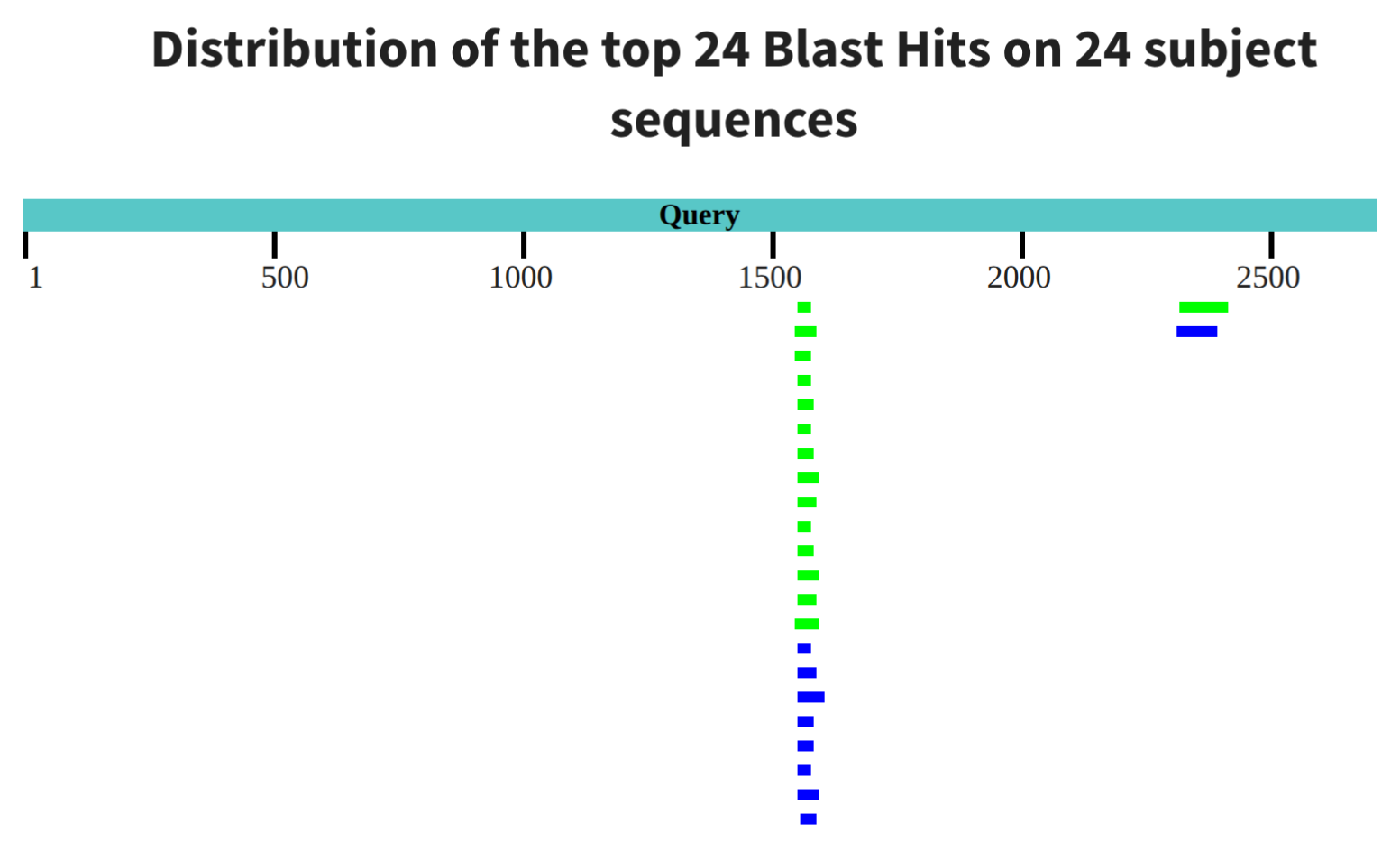
Был проведен blastn (рис 2), а не megablast, т.к. megablast оптимизирован для высокогомологичных последовательностей, а Canis lupus dingo сильно удален от пчел и высокой гомологии не ожидается.
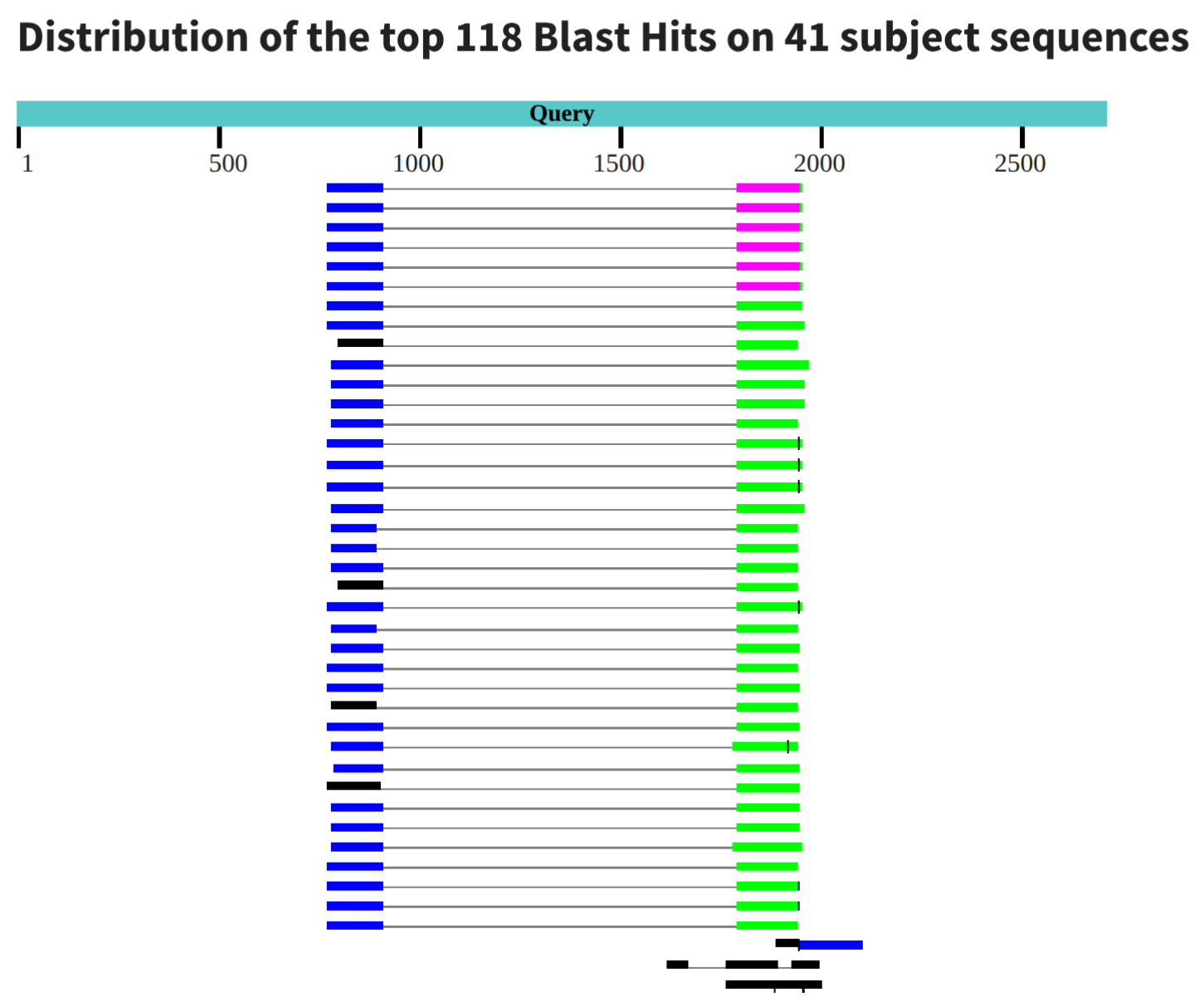
Провела tblastx (рис 3), который выявляет гомологию на уровне транслированных нуклеотидных баз данных. Я выбрала его, а не tblastn, т.к. на мой взгляд он более чувствительный и может выдать больше находок.
Оба метода blast я запускала по базе данных: refseq_genomes, в ней 38 сборок, входящих в рассматриваемый таксон. Когда я искала этот таксон через NCBI Datasets, в RefSeq включено 37 референсных сборок. Такая разница может быть обусловлена тем, что ncbi обновил данные, а бласт еще не успел.
Проиндексируем последовательность генома Canis lupus dingo:
makeblastdb -in GCF_003254725.2_ASM325472v2_genomic.fna -dbtype nucl
Проведем blastn для 16S рРНК и 23S рРНК E.Coli по полученной базе данных:
blastn -task blastn -query rRNA_ecoli_16S.fasta -db GCF_003254725.2_ASM325472v2_genomic.fna -out 16S_blast_results.out -word_size 7 -outfmt 7 -evalue 0.01
blastn -task blastn -query rRNA_ecoli_23S.fasta -db GCF_003254725.2_ASM325472v2_genomic.fna -out 23S_blast_results.out -word_size 7 -outfmt 7 -evalue 0.01
При запуске blastn я поменяла размер слова на 7, для большей чувствительности, а так же поставила порог на evalue 0.01, чтобы совсем маловероятные находки не выводились. А формат выдачи я поставила 7 - таблица с комментариями.
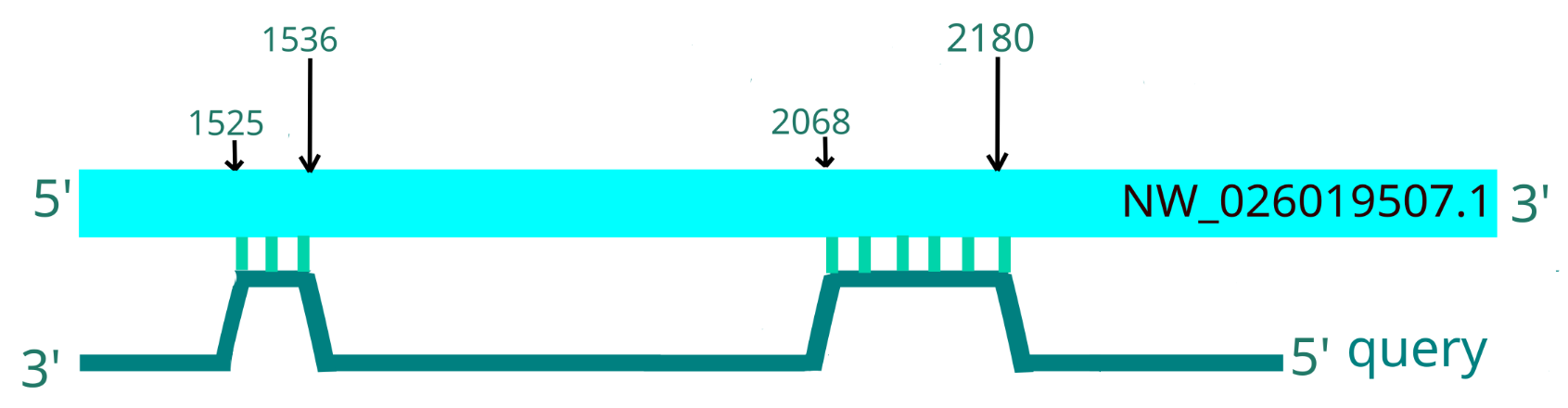
Выдача blastn для 16S
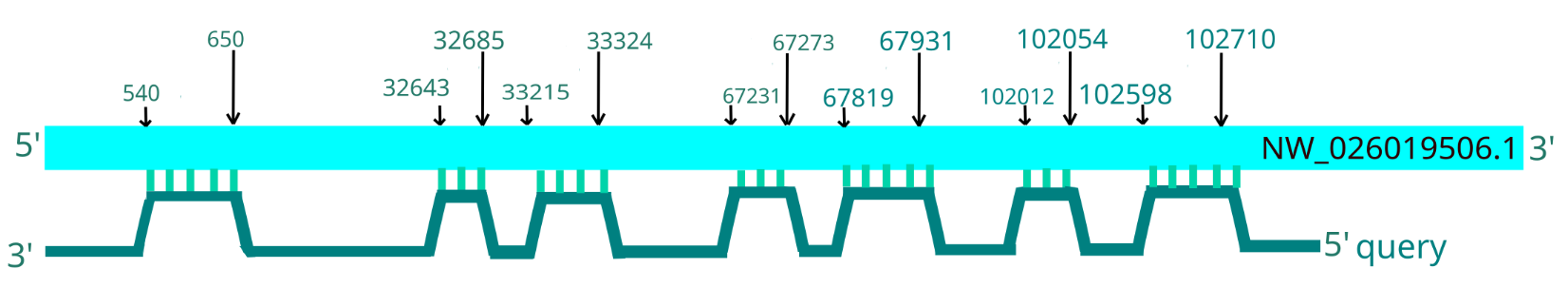
Выдача blastn для 23S
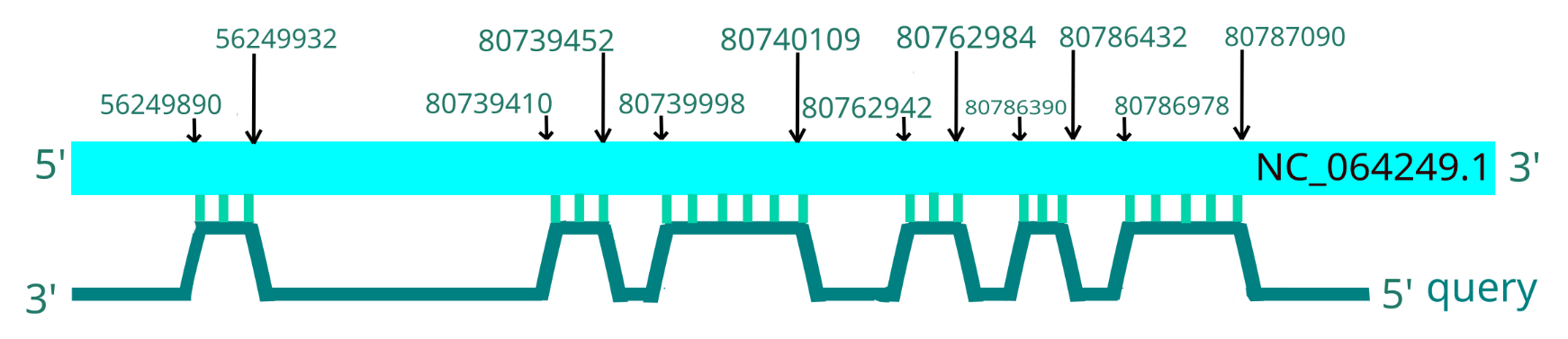
Проанализируем выдачу blastn для 16S рРНК c помощью визуализации в графическом редакторе Inksape (рис 4, 5, 6).
Для анализа я выбрала Mycoplasmoides pneumoniae M129 (сборка RefSeq: NC_000912.1) и Mycoplasmoides genitalium G37 (сборка RefSeq: NC_000908.2). Поиск проводился по базе данных NCBI Nucleotide с помощью запроса:
(complete[Title]) AND Mycoplasmoides[Organism]
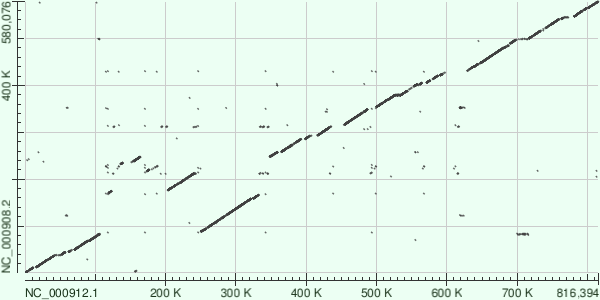
Для этих двух геномов был проведен megablast, blastn и tblastx, в результате чего были получены карты локального сходства (рис 7, 8, 9).
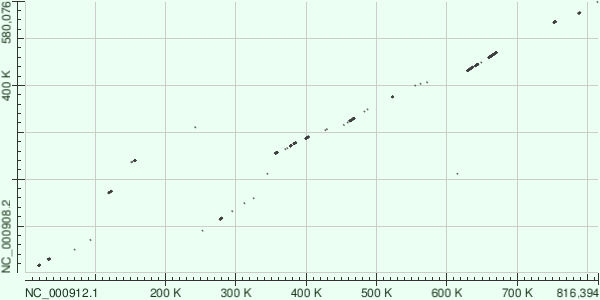
В данном случае, как мы можем видеть из рисунков 7 и 8, blastn говорит о схожести последовательностей больше, чем megablast. Это и логично, т.к. blastn имеет большую чувствительность, чем megablast, но за счет этого меньшую скорость.
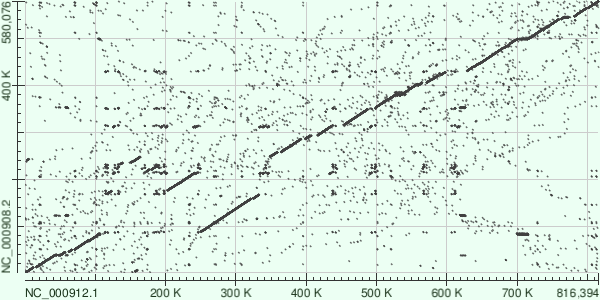
При построении карты локального сходства с помощью tblastx (рис 9) добавляется еще больше точечных совпадений, и видно близкое родство организмов. В целом, при анализе данной карты можно заметить тостаточно крупную дупликацию в геноме Mycoplasmoides pneumoniae (либо это произошла делеция в Mycoplasmoides genitalium), и много не очень больших делеций в геноме Mycoplasmoides genitalium.