Были построены карты локального сходства геномов архей: Halorhabdus tiamatea SARL4B и Halorhabdus utahensis DSM 12940 (рисунок 1) и Haloferax mediterranei ATCC 33500 и Haloferax volcanii DS2 (рисунок 2). Крупные изменения в геномах этих близкородственных организмов отмечены на рисунках разными цветами (синим - инверсии, красным - делеции и вставки).
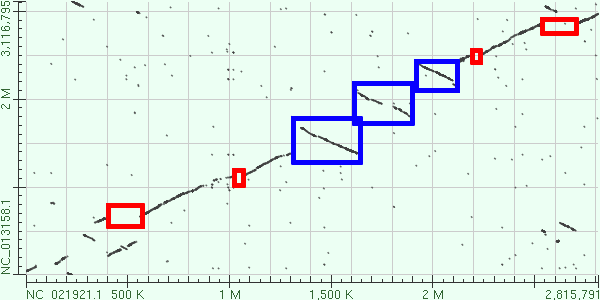
Рисунок 1. Карта локального сходства Halorhabdus tiamatea SARL4B (по горизонтали) и Halorhabdus utahensis DSM 12940 (по вертикали). Красным отмечены делеции в Halorhabdus utahensis (или вставки в Halorhabdus tiamatea).
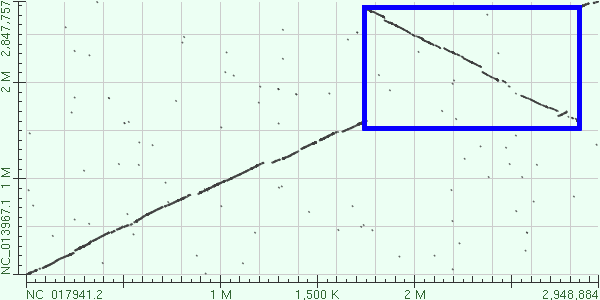
Рисунок 2. Карта локального сходства Haloferax mediterranei ATCC 33500 (по горизонтали) и Haloferax volcanii DS2 (по вертикали).
Пангеном был построен для бактерий Rickettsia rickettsii, а именно для штаммов Iowa, Brazil, Morgan и 'Shelia Smith'.
g-блоков 7, их порядок на хромосомах изображены на рисунке 3. Видно, что 2 блока есть во всех геномах (g4x1180482 и g4x86570). В геномах
Iowa, Morgan и Shelia порядок расположения g-блоков совпадает, при этом в Morgan и Shelia совпадает также i-блок между ними (поэтому можно считать,
что различных g-блоков не 7, а 6). Геном Brazil взят в обратном направлении, и в нем произошла перестановка двух g-блоков. Если перевернуть этот
геном, то расположение g-блоков будет совпадать с остальными штаммами, однако различия в расположении i-блоков останутся.

Рисунок 3. Расположение g-блоков на хромосомах.
s-блоков 209
суммарная длина s-блоков 1226970, процент от длины генома в среднем 97.159%
сходство геномов (процент консервативных позиций в объединенном выравнивании s-блоков): 99.8953%
h-блоков 9, r-блоков 105.
В таблице 1 представлено описание двух r-блоков и двух h-блоков. r23x140 встречается 3 раза в Brazil, 6 раз в Iowa, 7 раз в Morgan и
7 раз в Shelia. r10x112 встречается 3 раза в Brazil, 2 раза в Iowa, 2 раза в Morgan и 3 раза в Shelia. Блок h2x9671 есть только в
Morgan и Iowa. Блока h3x115 нет только в Brazil.
Таблица 1. Описание некоторых r-блоков (с повторома) и h-блоков (содержащихся не во всех геномах).
| Блок | r23х140 | r10х112 | h2x9671 | h3x115 |
| Число фрагментов | 23 | 10 | 2 | 3 |
| Число генов (во всех фрагментах из данного блока) | 23 | 6 | 9 | 3 |
| Длина | 140 | 112 | 9671 | 115 |
| Процент консервативных колонок | 93,57% | 87,5% | 99,98% | 100% |
Уникальных блоков найдено не было.
Примеры расхождений в аннотации:
1. В блоке s4x2113 у штаммов Shelia и Brazil отмечен гипотетический белок , в то время как у штаммов Morgan и Iowa этого белка нет (рисунок 4).
Это также отмечено в визуализаторе NPGE (рисунок 5). Так как в данном месте последовательности имеют высокое сходство, можно аннотировать этот
белок и в последних штаммах, хотя его функция не выяснена.
2. В блоке s4x2027 у штамма Brazil аннотирован гипотетический белок, а у остальных штаммов этого белка нет (рисунок 6). NPGE показывает немного не
так: по данным визуализатора этот белок не аннотирован только для Iowa (рисунок 7).
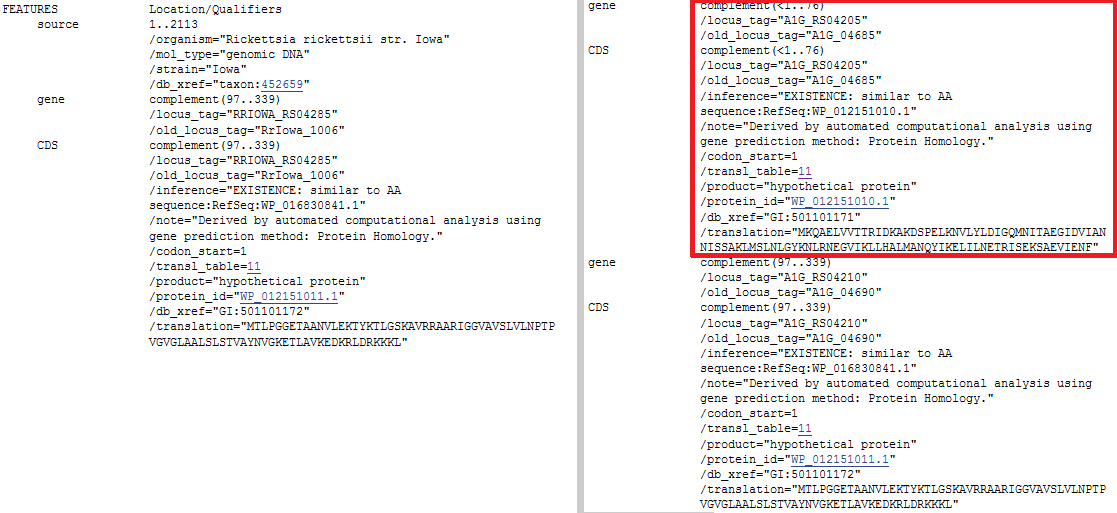
Рисунок 4. Слева - аннотация генов из блока s4x2113 для штамма Morgan, справа - для штамма Brazil. Красным выделен ген, который есть у Brazil и Shelia, но не у Iowa и Morgan.
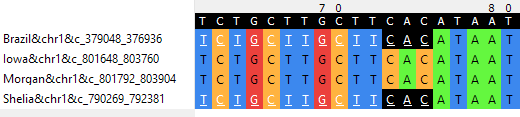
Рисунок 5. Изображение из визуализатора NPGE для гена, показаного на предыдущем рисунке.
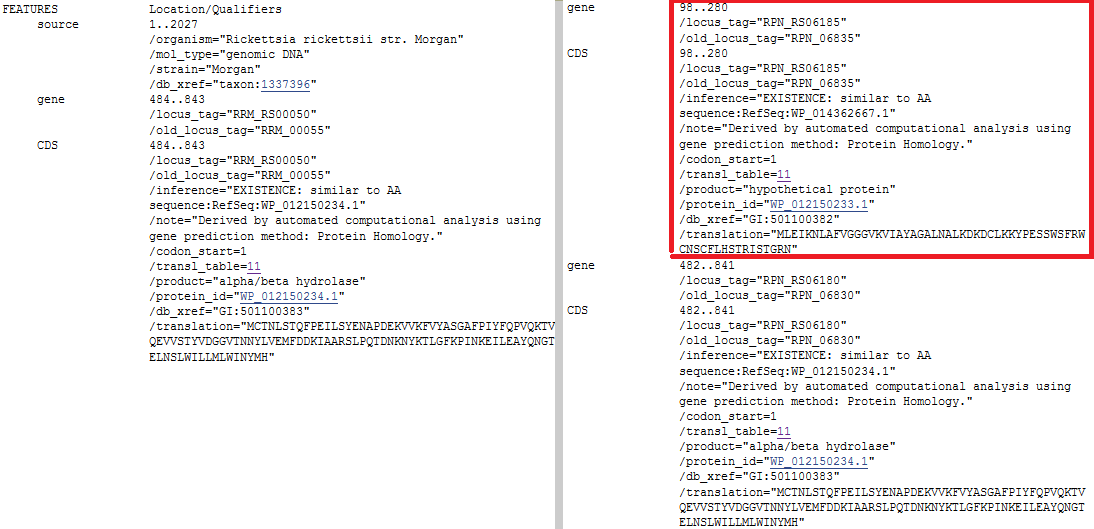
Рисунок 6. Слева - аннотация генов из блока s4x2027 для штамма Morgan, справа - для штамма Brazil. Красным выделен ген, который есть только у Brazil.

Рисунок 7. Изображение из визуализатора NPGE для гена, показаного на предыдущем рисунке.
В целом, построение нуклеотидных пангеномов - полезный способ, который позволяет отследить и описать глобальные изменения, произошедшие при эволюции различных видов или штаммов близкородственных бактерий или архей, такие как инверсии или дупликации генов. С его помощью можно выделить отдельные эволюционные группы штаммов. Также этот метод позволяет откорректировать и дополнить аннотации генов.
© Батырша Бостанов