Задание 1

Рис. 1 ДНК в A-форме. Пять раз повторена последовательность GATC. Ссылка на pdb
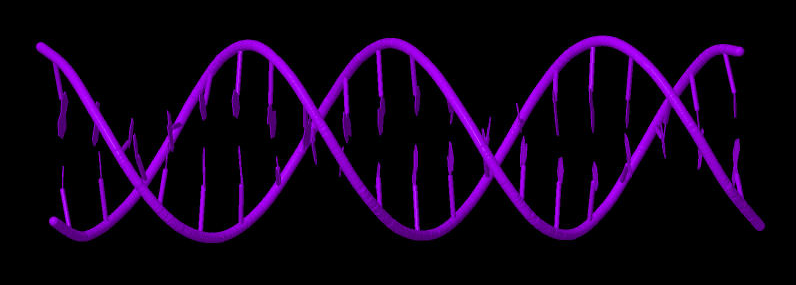
Рис. 2 ДНК в B-форме. GATC * 5. Ссылка на pdb

Рис. 3 ДНК в Z-форме. GC * 10. Ссылка на pdb
A-, B- и Z-формы были построены с помощью программы fiber из пакета X3DNA.
Задание 2
Упражнение 1
На всех рисунках показана А-форма ДНК.
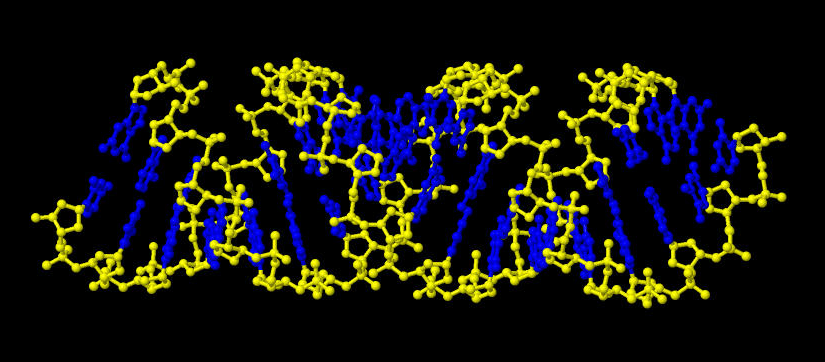
Рис. 4 Сахарофосфатный остов ДНК выделен жёлтым, а основания синим. Jmol-команда select backbone.

Рис. 5 Все нуклеотиды выделены фиолетовым. Jmol-команда select all .
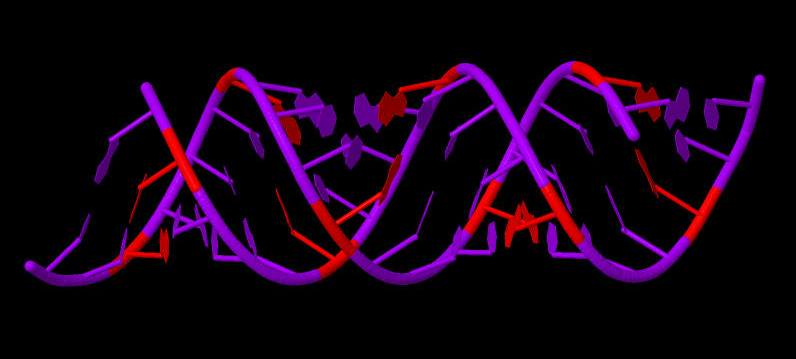
Рис. 6 Все нуклеотиды, содержащие основание аденин выделены красным. Всё остальное жёлтым. Jmol-команда select A.
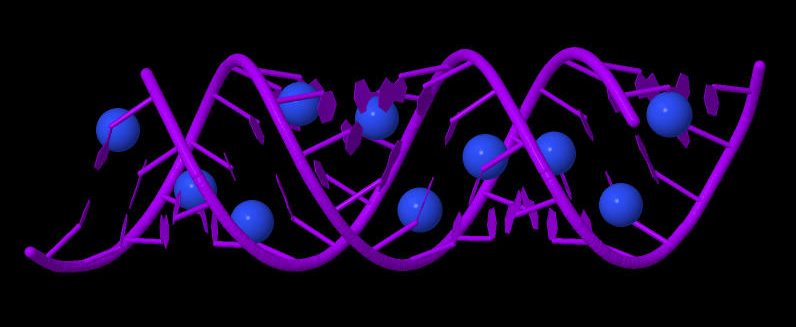
Рис. 7 Синим цветом выделены атомы N7 всех гуанинов.
Упражнение 3
В выданных файлах разрывов не обнаружено.
Задание 3
Упражнение 1
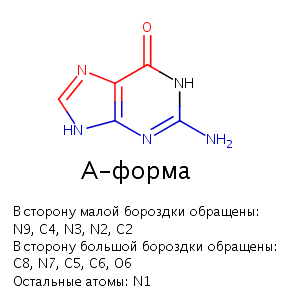
Рис. 9 Гуанин, нарисованный в Marvin sketch. Красным выделены атомы, направленные в сторону малой бороздки, а синим - в сторону большой. А-форма ДНК.
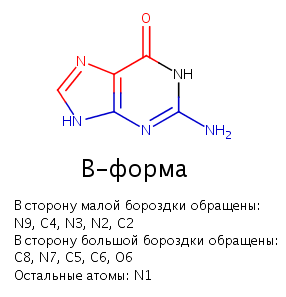
Рис. 10 Гуанин, нарисованный в Marvin sketch. B-форма ДНК.
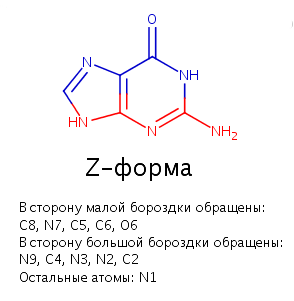
Рис. 11 Изображение цитозина, полученное в Marvin sketch. Обозначения такие же, что и на предыдущем рисунке. Z-форма.
Упражнение 2
|
|
A-форма
|
В-форма
|
Z-форма
|
|
Тип спирали (правая или левая)
|
Правая
|
Правая
|
Левая
|
|
Шаг спирали (Å)
|
28.03
|
33.75
|
43.5
|
|
Число оснований на виток
|
11
|
10
|
12
|
|
Ширина большой бороздки
|
7.98 [DC]28 - [DT]7
|
17.21 [DA]34 - [DC]4
|
9.87 [DC]28 - [DG]17
|
|
Ширина малой бороздки
|
16.81 [DG]37 - [DA]6
|
11.69 [DG]13 - [DC]32
|
16.08 [DC]8 - [DC]32
|
Упражнение 3
Таблица 1. Торсионные углы, измеренные вручную разных форм ДНК.
|
Торсионный угол
| |
A-форма
| |
B-форма
| |
|
α
| |
-51.7
| |
-29,9
| |
|
β
| |
174.8
| |
136,4
| |
|
γ
| |
41.7
| |
31.1
| |
|
δ
| |
79.1
| |
143.4
| |
|
ε
| |
-147.8
| |
-140.8
| |
|
ζ
| |
-75.1
| |
-160.5
| |
|
χ
| |
-157.2
| |
-98
| |
Задание 4
Упражнение 1
Торсионные углы в тРНК 1N78.pdb
Strand I
base alpha beta gamma delta epsilon zeta chi
1 G --- 170.0 50.9 86.6 -146.4 -80.0 -172.7
2 G -79.4 -175.8 54.9 84.0 -147.5 -70.4 -161.8
3 C -66.9 169.8 54.4 82.7 -150.0 -72.7 -164.3
4 C -67.9 173.7 53.9 81.6 -152.4 -79.4 -161.7
5 C -61.6 169.7 54.8 81.2 -151.3 -72.3 -166.5
6 C -61.7 171.4 58.2 87.3 -156.2 -79.4 -164.9
7 A -51.5 174.8 59.9 137.6 --- --- -122.1
8 G --- 142.7 40.0 81.9 -148.5 -73.1 -173.8
9 G -61.7 -179.6 48.0 81.3 -153.7 -69.3 -170.1
10 G -59.4 -178.8 44.5 81.1 -171.6 -78.4 -159.2
11 G 160.5 -157.2 168.6 85.9 -107.1 -47.3 176.5
12 G -108.7 90.8 151.8 88.8 -117.5 -68.2 163.3
13 U -68.6 -180.0 45.5 82.5 -125.2 -77.6 -162.3
14 U -52.7 155.6 50.9 81.0 --- --- -153.5
15 A --- 167.5 48.8 85.3 -141.5 -63.3 -167.8
16 G -66.0 173.7 52.0 81.7 -152.2 -77.5 -164.9
17 G -59.7 174.8 48.9 82.0 -149.3 -71.5 -166.0
18 C -65.0 174.3 53.2 82.6 -155.0 -68.8 -159.4
19 C -70.3 174.5 57.1 78.3 -153.2 -69.3 -166.3
20 G -57.1 174.6 54.7 81.1 -155.7 -81.6 -164.7
21 A -63.2 173.9 50.5 82.4 --- --- -154.3
22 G --- 178.1 52.4 88.3 -148.4 -67.7 -178.6
23 U -70.4 -175.8 48.0 81.6 -154.2 -71.0 -164.7
24 C -65.9 -174.2 48.5 81.5 -152.9 -66.4 -159.7
25 U 140.4 -155.7 -167.0 83.4 -153.3 -77.5 -171.5
26 A -57.7 -174.8 45.3 79.8 -139.4 -66.7 -170.7
27 G -43.4 161.1 58.8 129.6 --- --- -119.6
28 G --- -131.3 48.7 114.2 --- --- -64.1
29 G --- -172.0 45.6 87.6 -148.4 -76.0 -168.5
30 G -72.6 -177.1 47.3 85.4 -149.1 -69.6 -163.9
31 C -72.8 172.1 55.8 84.4 -148.2 -75.4 -157.8
32 C -54.9 171.7 42.9 81.4 -152.6 -78.8 -158.0
33 C -60.9 170.1 54.8 79.6 -150.6 -70.8 -166.3
34 C -63.3 169.5 59.0 84.2 -160.7 -75.5 -160.5
35 A -42.2 -171.5 39.8 98.9 --- --- -115.4
36 G --- 147.1 33.3 82.1 -147.3 -73.4 -168.1
37 G -60.5 179.2 46.7 80.6 -154.6 -70.2 -167.0
38 G -58.3 177.2 45.9 82.3 -162.1 -89.1 -161.4
39 G 156.9 -149.8 166.7 85.5 -103.1 -52.8 175.5
40 G -101.2 90.6 151.4 88.2 -111.0 -74.5 160.1
41 U -68.1 176.7 44.9 84.2 -126.0 -77.3 -160.5
42 U -52.7 157.2 50.2 81.7 --- --- -152.3
43 A --- 164.4 53.8 85.4 -139.0 -60.9 -169.5
44 G -67.7 174.0 49.9 79.9 -153.5 -73.6 -161.3
45 G -60.5 173.5 48.6 79.9 -151.6 -72.9 -168.6
46 C -63.4 177.6 50.8 82.4 -155.8 -67.8 -157.5
47 C -67.4 171.7 59.1 79.4 -153.6 -70.3 -167.5
48 G -59.2 174.4 55.1 81.4 -152.5 -80.6 -164.1
49 A -65.8 170.3 54.6 79.7 --- --- -155.4
50 G --- 177.3 48.1 88.1 -154.0 -63.5 -172.9
51 U -66.6 -176.2 47.6 82.4 -152.0 -71.8 -165.9
52 C -68.6 -174.3 48.6 81.6 -154.7 -69.3 -158.6
53 U 144.9 -154.1 -169.6 84.6 -149.2 -81.2 -174.1
54 A -54.7 -176.8 43.7 78.4 -142.7 -62.5 -168.6
55 G -49.0 168.8 61.4 126.3 --- --- -121.4
56 G --- -130.7 46.6 113.9 --- --- -59.7
Strand II
base alpha beta gamma delta epsilon zeta chi
1 C 157.9 -143.8 161.4 87.5 --- --- -168.9
2 U -70.0 176.4 52.4 83.8 -168.0 -94.2 -160.5
3 G -47.2 160.9 54.1 82.8 -149.9 -65.8 -174.1
4 G -64.4 -178.5 51.0 84.1 -153.2 -79.1 -169.2
5 G -67.4 178.8 47.4 79.5 -163.2 -65.5 -166.9
6 G -64.8 178.2 47.2 83.1 -160.2 -67.8 -159.0
7 U -73.6 179.7 54.5 83.4 -157.5 -67.0 -158.2
8 C -65.0 177.1 55.6 84.4 -156.9 -60.6 -163.4
9 C -63.4 168.9 55.7 82.2 -154.5 -65.1 -162.6
10 C -73.5 -169.5 49.6 84.4 -160.3 -76.4 -155.6
11 C -70.7 -175.7 51.4 83.8 -159.3 -67.7 -163.7
12 C --- -150.3 47.5 82.2 -158.3 -74.2 -165.5
13 A --- -96.6 179.1 81.6 --- --- -149.1
14 G --- -149.9 -64.9 108.9 --- --- -115.4
15 C -61.1 167.7 51.8 85.0 --- --- -162.5
16 C -63.2 173.9 53.7 82.8 -141.4 -70.0 -159.9
17 C -66.3 172.8 55.1 83.9 -154.5 -70.5 -164.9
18 G -64.4 178.8 53.5 83.2 -158.3 -72.1 -165.6
19 G -72.3 -179.5 52.5 79.0 -153.2 -71.9 -169.1
20 C -64.4 178.2 50.5 78.2 -146.9 -73.6 -159.7
21 G -67.1 169.9 50.2 81.8 -148.3 -65.8 -164.9
22 C -60.0 176.0 52.8 84.1 -144.6 -61.6 -163.0
23 A -63.5 176.3 50.3 81.0 -152.1 -76.5 -164.6
24 G -54.3 170.7 53.6 81.0 -153.0 -69.0 -166.0
25 G --- 142.1 45.6 80.2 -147.5 -81.7 -172.6
26 U --- -156.2 59.0 85.8 --- --- -152.1
27 C --- 108.0 172.3 133.5 --- --- -123.6
28 C --- 162.2 52.6 83.3 --- --- -160.1
29 C 157.5 -150.9 163.6 85.5 --- --- -162.5
30 U -63.1 171.0 52.5 86.4 -168.3 -90.1 -156.2
31 G -56.1 171.2 54.6 85.2 -143.8 -72.2 -169.4
32 G -54.3 169.2 49.6 81.1 -156.8 -73.5 -169.4
33 G -63.1 171.0 47.3 77.2 -151.0 -77.4 -172.0
34 G -73.4 -177.5 49.0 83.5 -154.9 -71.6 -159.9
35 U -77.4 -171.2 52.1 82.4 -160.5 -60.5 -160.0
36 C -62.9 176.1 57.2 85.2 -161.1 -55.5 -168.4
37 C -64.0 174.5 51.3 81.7 -157.4 -67.1 -171.4
38 C -73.2 -177.2 53.8 83.0 -157.3 -74.5 -161.6
39 C -75.2 -174.5 54.3 83.3 -156.2 -73.4 -167.2
40 C --- -145.8 56.5 86.1 -160.2 -72.0 -172.6
41 A --- -96.4 -179.2 81.5 --- --- -148.8
42 G --- -149.3 -60.2 106.3 --- --- -112.0
43 C -63.4 167.4 52.5 85.8 --- --- -157.1
44 C -63.7 175.3 54.6 83.3 -145.3 -67.5 -162.4
45 C -70.8 175.4 58.7 84.5 -154.5 -70.6 -164.0
46 G -56.5 172.3 51.3 80.5 -157.4 -68.9 -166.0
47 G -59.6 172.5 45.4 78.3 -150.3 -73.9 -167.9
48 C -74.0 -177.1 53.2 81.2 -141.6 -81.6 -161.3
49 G -70.8 176.4 51.0 81.3 -153.6 -61.3 -161.9
50 C -58.3 175.2 53.0 83.5 -144.5 -58.8 -167.0
51 A -72.6 178.9 53.6 81.1 -150.8 -76.8 -169.1
52 G -53.4 170.5 53.2 80.8 -152.7 -70.8 -169.6
53 G --- 143.1 42.4 81.2 -148.7 -79.8 -172.0
54 U --- -173.4 44.3 84.2 --- --- -154.1
55 C --- 109.0 170.8 132.1 --- --- -126.9
56 C --- 162.8 52.7 83.0 --- --- -154.3
Таблица 3. Средние значения торсионных углов в тРНК 1N78.pdb.
|
Торсионный угол
| |
Значение
| |
|
α
| |
-49.5
| |
|
β
| |
55.3
| |
|
γ
| |
48.6
| |
|
δ
| |
85.7
| |
|
ε
| |
-150
| |
|
ζ
| |
-71.7
| |
|
χ
| |
-147
| |
Самый "деформированный" нуклеотид - G11 первой цепи.
Торсионные углы ДНК 1I3J.pdb
Strand I
base alpha beta gamma delta epsilon zeta chi
1 T --- 166.7 60.3 146.6 -171.5 -102.4 -105.7
2 C -50.8 -174.0 31.0 150.4 175.8 -114.2 -85.3
3 T -16.9 -174.6 13.5 150.0 -161.2 -114.5 -99.4
4 T 18.2 -162.5 -58.8 151.2 -159.9 -95.2 -95.8
5 G -60.2 175.4 34.8 153.6 -122.6 -179.0 -65.5
6 G -67.3 137.9 49.4 147.5 -149.2 -150.8 -89.9
7 G -71.5 157.8 56.9 147.8 -174.3 -92.2 -122.1
8 T -55.9 -159.4 25.3 144.7 161.1 -82.0 -95.7
9 C 151.3 150.4 -171.3 135.6 -154.7 -113.1 -129.7
10 T -38.9 -174.8 29.9 144.0 -170.3 -109.5 -105.1
11 A -48.8 -178.3 40.5 147.1 -166.1 -113.6 -93.0
12 C 22.8 -169.9 -55.1 153.1 -178.1 -115.7 -91.8
13 C 27.5 -161.8 -61.8 149.8 -159.9 -93.1 -124.5
14 G -55.8 -169.5 37.6 148.3 170.5 -108.5 -99.3
15 T -43.8 -171.3 39.0 147.8 -168.4 -120.7 -103.4
16 T 26.1 -168.3 -53.3 154.4 179.4 -89.1 -107.1
17 T -50.1 -171.0 29.7 156.8 -166.8 -154.0 -84.6
18 A -44.9 159.2 47.7 146.3 177.3 -90.7 -92.8
19 A -37.0 -155.4 25.0 151.9 -162.2 -112.0 -86.8
20 T 24.7 -126.9 -75.8 161.3 --- --- -119.5
Strand II
base alpha beta gamma delta epsilon zeta chi
1 A 16.3 -176.4 -51.6 159.8 --- --- -94.6
2 G -52.4 164.8 49.0 151.0 -161.1 -112.2 -107.6
3 A -38.5 160.2 37.6 152.6 -163.9 -149.8 -85.8
4 A -48.7 157.8 48.3 145.1 -168.7 -123.3 -83.3
5 C -41.4 175.4 31.3 144.7 -156.6 -144.8 -86.6
6 C -27.9 -172.7 17.8 142.9 -164.0 -105.2 -105.6
7 C 41.2 174.7 -57.0 151.5 -176.1 -98.0 -112.2
8 A -35.2 167.6 23.9 147.1 -172.2 -120.7 -111.5
9 G -49.4 -177.4 29.3 152.5 -166.5 -150.0 -85.4
10 A -33.9 175.0 46.1 149.0 -175.2 -99.2 -96.8
11 T -48.9 -167.7 35.9 146.1 -171.5 -123.0 -99.1
12 G 32.9 -177.8 -52.9 151.4 165.5 -100.4 -125.1
13 G -42.4 -161.1 33.1 154.1 177.5 -130.9 -94.6
14 C 32.0 -161.0 -57.9 150.1 -178.5 -93.9 -100.8
15 A -21.7 162.3 31.9 147.7 -170.7 -111.2 -96.5
16 A -35.0 -173.9 24.6 151.0 170.0 -131.3 -84.8
17 A -36.7 174.5 42.1 147.1 175.8 -101.8 -85.5
18 T -31.1 178.3 26.2 144.4 -176.8 -119.8 -93.6
19 T 6.0 -151.1 -80.2 143.3 -167.5 -112.0 -108.4
20 A --- 177.9 47.4 155.3 -132.9 -137.0 -85.2
Таблица 4. Средние значения торсионных углов в ДНК 1I3J.pdb.
|
Торсионный угол
| |
Значение
| |
|
α
| |
-21.91
| |
|
β
| |
-29.51
| |
|
γ
| |
17.55
| |
|
δ
| |
148.66
| |
|
ε
| |
-80.84
| |
|
ζ
| |
-115.98
| |
|
χ
| |
-98.19
| |
Самый "деформированный" нуклеотид - C9 первой цепи.
Упражнение 2
Стебли: [501-507] - [566-572]; [549-553] - [561-565]; [538-544] - [526-532];[510-513] - [522-525].
Неканонические пары оснований: G[502]-U[571], U[555]-G[518], A[538]-C[532], A[544]-G[526], U[513]-G[522]
Стабилизирующие пары оснований: G[519]-C[556].
Упражнение 3
Данные о перекрывании нуклеотидов.
step i1-i2 i1-j2 j1-i2 j1-j2 sum
1 TC/GA 3.16( 0.25) 0.00( 0.00) 0.00( 0.00) 5.36( 2.46) 8.52( 2.72)
2 CT/AG 6.25( 0.73) 0.00( 0.00) 0.00( 0.00) 1.04( 0.10) 7.29( 0.82)
3 TT/AA 6.06( 0.34) 0.00( 0.00) 0.00( 0.00) 4.42( 2.62) 10.49( 2.96)
4 TG/CA 6.11( 1.21) 0.00( 0.00) 0.00( 0.00) 3.11( 0.00) 9.22( 1.21)
5 GG/CC 5.03( 2.14) 0.00( 0.00) 0.00( 0.00) 6.50( 1.37) 11.53( 3.51)
6 GG/CC 2.63( 0.16) 0.00( 0.00) 0.00( 0.00) 3.25( 0.27) 5.89( 0.43)
7 GT/AC 6.58( 1.94) 0.00( 0.00) 0.00( 0.00) 4.51( 2.95) 11.08( 4.88)
8 TC/GA 3.60( 1.20) 0.00( 0.00) 0.00( 0.00) 2.00( 0.04) 5.60( 1.24)
9 CT/AG 5.12( 0.00) 0.00( 0.00) 0.00( 0.00) 5.78( 4.19) 10.91( 4.20)
10 TA/TA 2.35( 0.00) 0.00( 0.00) 0.00( 0.00) 3.71( 0.35) 6.06( 0.35)
11 AC/GT 5.49( 3.82) 0.00( 0.00) 0.00( 0.00) 4.21( 0.71) 9.70( 4.53)
12 CC/GG 0.58( 0.00) 0.00( 0.00) 0.05( 0.00) 3.35( 1.65) 3.98( 1.65)
13 CG/CG 0.83( 0.00) 0.00( 0.00) 0.73( 0.00) 2.73( 0.68) 4.30( 0.68)
14 GT/AC 5.22( 1.24) 0.00( 0.00) 0.00( 0.00) 5.40( 3.97) 10.62( 5.21)
15 TT/AA 5.30( 0.15) 0.00( 0.00) 0.00( 0.00) 2.80( 1.45) 8.10( 1.59)
16 TT/AA 6.26( 0.41) 0.00( 0.00) 0.00( 0.00) 4.53( 2.80) 10.80( 3.21)
17 TA/TA 0.46( 0.00) 0.00( 0.00) 0.00( 0.00) 3.02( 0.00) 3.48( 0.00)
18 AA/TT 6.51( 4.71) 0.00( 0.00) 0.00( 0.00) 5.07( 0.00) 11.58( 4.71)
19 AT/AT 6.85( 3.37) 0.00( 0.00) 0.00( 0.00) 6.05( 2.29) 12.90( 5.66)
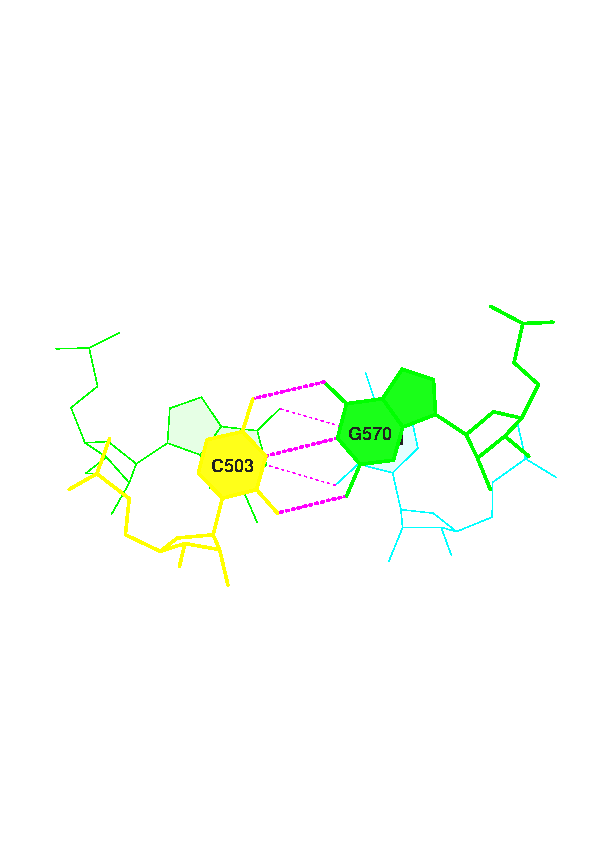
Рис. 12 Изображение стекинг-взаимодействия между наиболее сильно перекрывающимися парами нуклеотидов.










