AC: 00115
ID: COX1
Название: Cytochrome C and Quinol oxidase polypeptide I
Число последовательностей среди бактерий: 13661
Требуется выбрать два белковых домена.
Выбранные домены — PF00115, PF00510. Ниже приведены данные о них.

AC: 00115
ID: COX1
Название: Cytochrome C and Quinol oxidase polypeptide I
Число последовательностей среди бактерий: 13661
AC: 00510
ID: COX3
Название: Cytochrome c oxidase subunit III
Число последовательностей среди бактерий: 8528
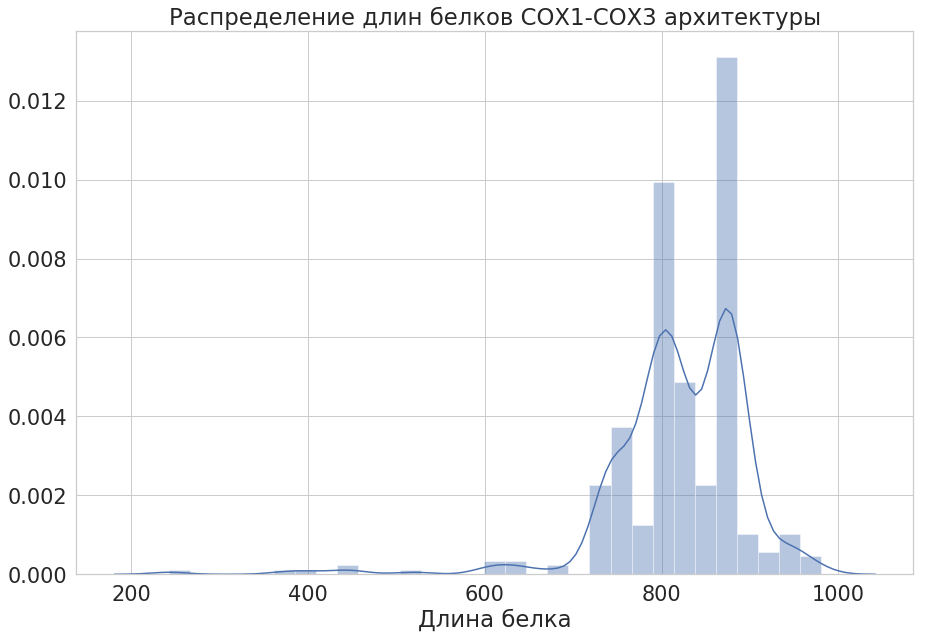
Pfam выдал 153 белка, составленных только из данных двух доменов.

Uniprot же выдал 372 белка.
Поиск по UniProt был проведён со следующим запросом:
taxonomy:"Bacteria [2]" database:(type:pfam pf00115) database:(type:pfam pf00510)
Была составлена таблица с информацией о всех бактериальных
белках с выбранной архитектурой.
В настройках Columns выбирал те колонки, которые были указаны на занятии:
Organism (Names & Taxonomy)
Protein names (Names & Taxonomy)
Length (Sequences)
Taxonomic lineage (FAMILY) (Taxonomic lineage)
Taxonomic lineage (GENUS) (Taxonomic lineage)
Taxonomic lineage (PHYLUM) (Taxonomic lineage)
Pfam (Family and domain)
Скачать таблицу в Excel-файле можно здесь.