Выравнивание последовательностей
Для выполнения задания я получила список идентификаторов Swiss-Prot с заданной мнемоникой организма (*_ecoli и *_bacsu) командой, представленной ниже:
infoseq 'sw:*_ecoli ( или *_bacsu)' -only -name -nohead -out ecoli.txt ( или bacsu.txt)
Далее командой
cut -f 1 -d '_' ecoli.txt bacsu.txt | sort | uniq -d > file.txt,
где "file" - имя файла, заданное пользователем, формируется итоговый список тех мнемоник, ччто содержатся у обеих бактерий. Из них я выбрала 3 произвольные, которые будут рассмотрены в заданиях ниже.
1. Глобальное парное выравнивание гомологичных белков
Используемая команда:
needle sw:*_ecoli sw:*_bacsu *.needle -auto
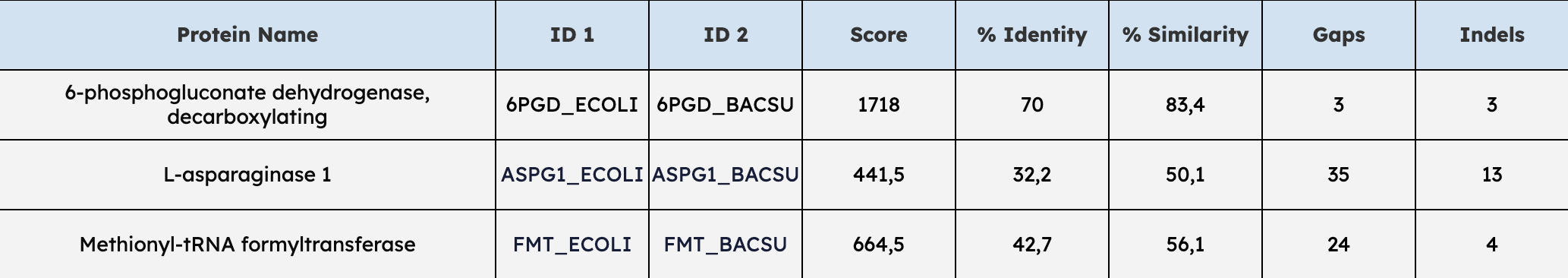 Таблица 1. Глобальное парное выравнивание гомологичных белков
Таблица 1. Глобальное парное выравнивание гомологичных белков
 Таблица 1. Глобальное парное выравнивание гомологичных белков
Таблица 1. Глобальное парное выравнивание гомологичных белков
2. Локальное парное выравнивание гомологичных белков
Используемая команда:
water sw:*_ecoli sw:*_bacsu *.water -auto
 Таблица 2. Локальное парное выравнивание гомологичных белков
Таблица 2. Локальное парное выравнивание гомологичных белков
 Таблица 2. Локальное парное выравнивание гомологичных белков
Таблица 2. Локальное парное выравнивание гомологичных белков
3. Результат применения программ выравнивания к неродственным белкам
Для выполнения этого задания я снова выбрала 2 произвольных белка предложенных бакткрий, но на этот раз один из них принадлежал бактерии Escherichia coli (strain K12) [СBL_ECOLI], а другой - Bacillus subtilis (strain 168) [ССА_BACSU]. Затем я применила к соответствующим файлам программы глобального и локального выравнивания, как это было сделано в заданиях 1 и 2, и на основании полученных данных составила сравнительную таблицу (см. Таблица 3).
 Таблица 3. Сравнение неродственных белков
Из данных в Таблице 3 видно, что белки имеют низкие индексы идентичности и схожеси при обоих типах выравнивания, а также сравнительно больше количество гэпов, что подтверждает выбор этих белков, как удовлетвояющий заданным условиям. К тому же, можно предположить, что именно из-за этих отличий в том числе белки выполняют различные функции. Например, Например, HTH-type transcriptional regulator cbl (СBL_ecoli) может быть дополнительным регуляторным белком в составе цис-регулона, а CCA-adding enzyme (ССA_bacsu) катализирует добавление и восстановление необходимой 3’ коцевой последовательности ССА в тРНК без использования матричной нуклеиновой кислоты. Однако также можно заметить, что процент схожести в локальном выравнивании выше, чем в глобальном. Возможно, существует какая-то совокупность свойств этих белков, позволяющая проявлять частично схожие функции.
Таблица 3. Сравнение неродственных белков
Из данных в Таблице 3 видно, что белки имеют низкие индексы идентичности и схожеси при обоих типах выравнивания, а также сравнительно больше количество гэпов, что подтверждает выбор этих белков, как удовлетвояющий заданным условиям. К тому же, можно предположить, что именно из-за этих отличий в том числе белки выполняют различные функции. Например, Например, HTH-type transcriptional regulator cbl (СBL_ecoli) может быть дополнительным регуляторным белком в составе цис-регулона, а CCA-adding enzyme (ССA_bacsu) катализирует добавление и восстановление необходимой 3’ коцевой последовательности ССА в тРНК без использования матричной нуклеиновой кислоты. Однако также можно заметить, что процент схожести в локальном выравнивании выше, чем в глобальном. Возможно, существует какая-то совокупность свойств этих белков, позволяющая проявлять частично схожие функции.
 Таблица 3. Сравнение неродственных белков
Таблица 3. Сравнение неродственных белков
4. Множественное выравнивание белковых последовательностей. Jalview.
Для выполнения данного задания я выбрала 1 из белков, рассматриваемых в 1 задании: FMT_*, где * - принадлежность к какому-либо организму (полное имя белка см. в задании1). Через базу UniProt KB и ее расширенный поиск я посмотрела, у каких организмов встречается данный белок. Учитывая банк Swiss-Prot, запрос выдал 696 результатов, из которых я выбрала 7 следующих:
FMT_HUMAN, FMT_ECOLI, FMT_YEAST, FMT_BOVIN, FMT_SALTY, FMT_SCHPO, FMT_BACSU;
соответственно принадлежащие организмам:
-Homo sapiens (Human), Escherichia coli (strain K12), Saccharomyces cerevisiae, Bos taurus (Bovine), Salmonella typhimurium (strain LT2), Schizosaccharomyces pombe (strain 972), Bacillus subtilis (strain 168).
Затем я создала файл FMT_txt, строками которого являются sw:FMT_*. Далее командами
seqret @FMT.txt FMT.fastа
muscle -in FMT.fasta -out FMT_alignment.fasta
формируется нужный файл в формате fasta. Полученный файл был открыт в Jalview, ссылка на который представлена ниже:
Исходя из результатов, сложно судить о гомологии всех 7 выбранных белков, так как визуально можно оценить большое количество гэпов, например блоки [347-358], [283-295], крупный блок [1-49], а также малую протяженность консервативных блоков. Наряду с ними можно увидеть схожие блоки: [52-72], [85-92], [121-138], но самое заметное сходство наблюдается в блоке [160-262], в котором практически нет даже единичных гэпов и заметны консервативные колонки.
Также можно рассмотреть белки попарно. В упомянутом блоке с 1 по 49 аминокислотный остаток наблюдается практически абсолютное сходство между белковыми последовательностями FMT_HUMAN и FMT_BOVIN, принадлежащими таксиномически близким организмам, поэтому между ними наблюдается наибольшее сходство, что говорит о гомологии данных белков. Подобную ситуацию можно также наблюдать при сравнении последовательностей FMT_ECOLI и FMT_SALTY имеющих одинаковые длину последовательноти,консервативные участки и даже количество гэпов.