Алгоритмы поиска BLAST
1. Ген, кодирующий дельта-субъединицу АТФ-синтазы
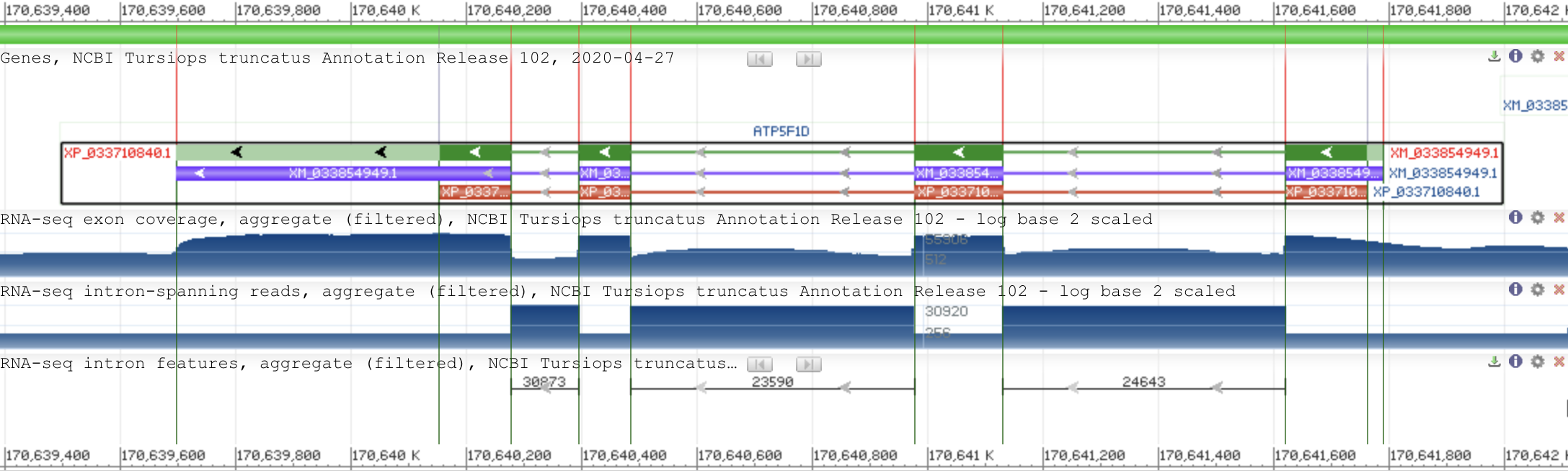
Рисунок 1.Иллюстрация фрагмента, содержащего ген дельта-субъединицы АТФ-синтазы. Весь исследуемй ген изображен в 3 цветах: зеленым (интроны и экзоны, серым - некодирующие фрагменты), фиолетовым (весь ген) и красным (только интроны и экзоны). Тонкими линиями изображены интроны.
2. Алгоритмы поиска BLAST для фрагмента ДНК
3. Поиск генов основных рибосомальных РНК по далекому гомологу
makeblastdb -in GCF*fna -dbtype nucl,
где GCF*fna - файл с нуклеотидной последовательностью генома. Далее был проведен локальный поиск BLAST по двум видам рРНК: 16s (файл с последовательностью: 16s.txt) и 23s (23s.txt). Использованные команды:для 16s: blastn -task blastn -query 16s.txt -db GCF*fna -out 16s_result.txt -outfmt 7
для 23s: blastn -task blastn -query 23s.txt -db GCF*fna -out 23s_result.txt -outfmt 7
16s рРНК выступает в роли идентификатора прокариот, то есть, является характерной именно для них. Помимо этого, данная рРНК связывается с последовательностью Шайна-Дальгарно мРНК, стабилицирует правильное расположение кодона и антикодона, а также способствует связыванию малой и большой рибосомальных субъединиц путем взаимодействия с 23s рРНК.
Результаты локального поиска для каждой рРНК: 16s_result.txt и 23s_result.txt. Для 23s рРНК был найден 1 гомолог (как гомолог был помечен еще один фрагмент, но он представляет сбой L-rRNA, охарактеризованный, как 16s, то есть гомологом не является). Для 16s рРНК было найдено 3 гомолога, один из них аннотирован, как 12s рРНК.