Филогенетическая реконструкция и сравнение деревьев
В ходе выполнения данного практикума были построены филогенетические деревья на основе выравнивания аминокислотных последовательностей цитохромов B выбранных в практикуме 1 организмов. Деревья далее визуализированы в программе ITol.
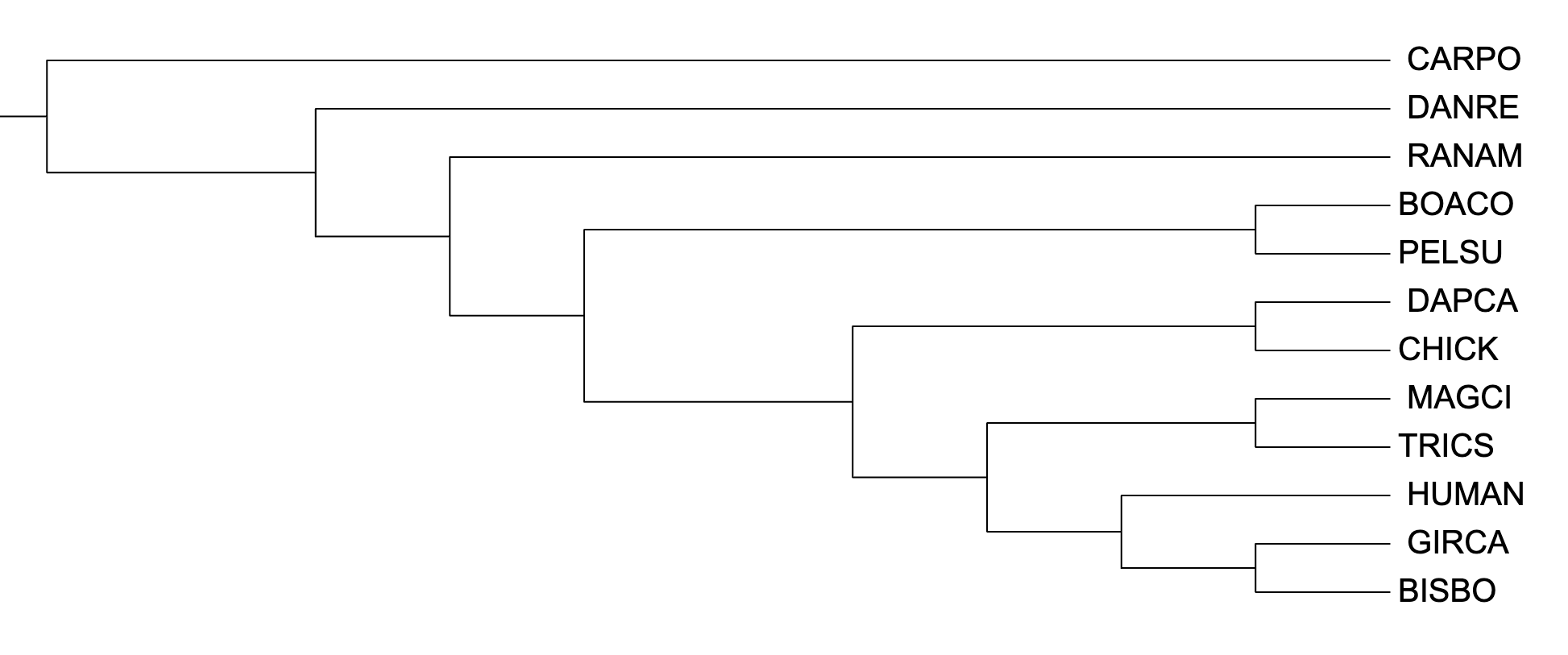

Изображение 1.Филогенетическое дерево, построенное на основании таксономической принадлежности организмов (из пр1)

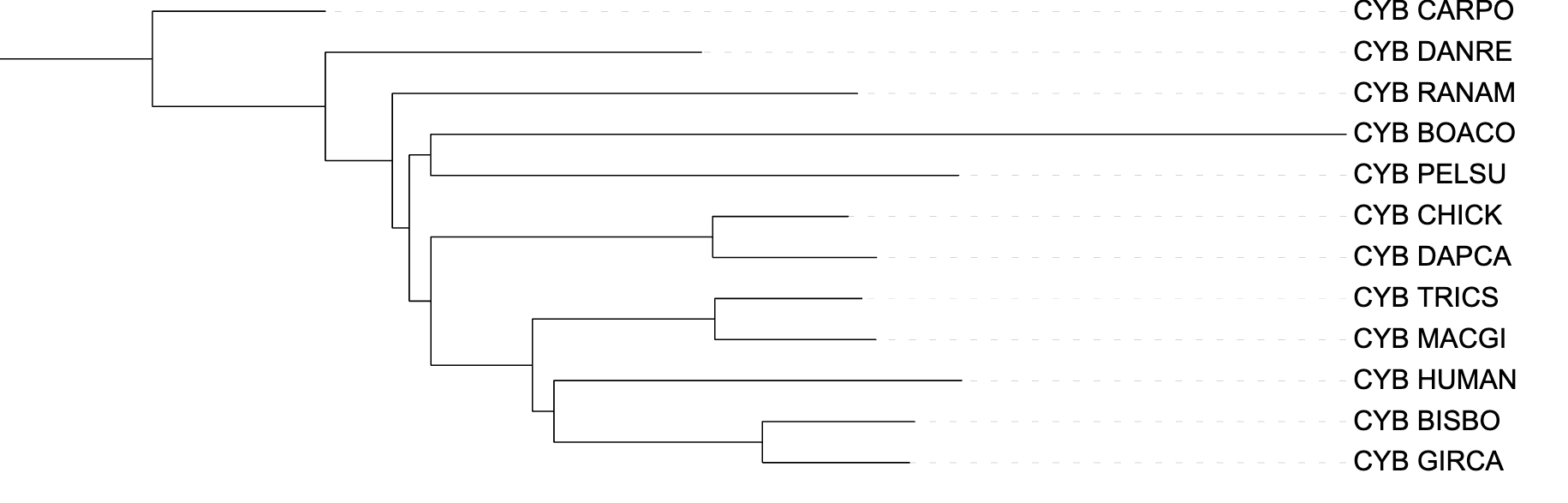
Изображение 2.Филогенетическое дерево, реконструированное на основе множественного выравнивания программой fastme с параметром -p p-distance

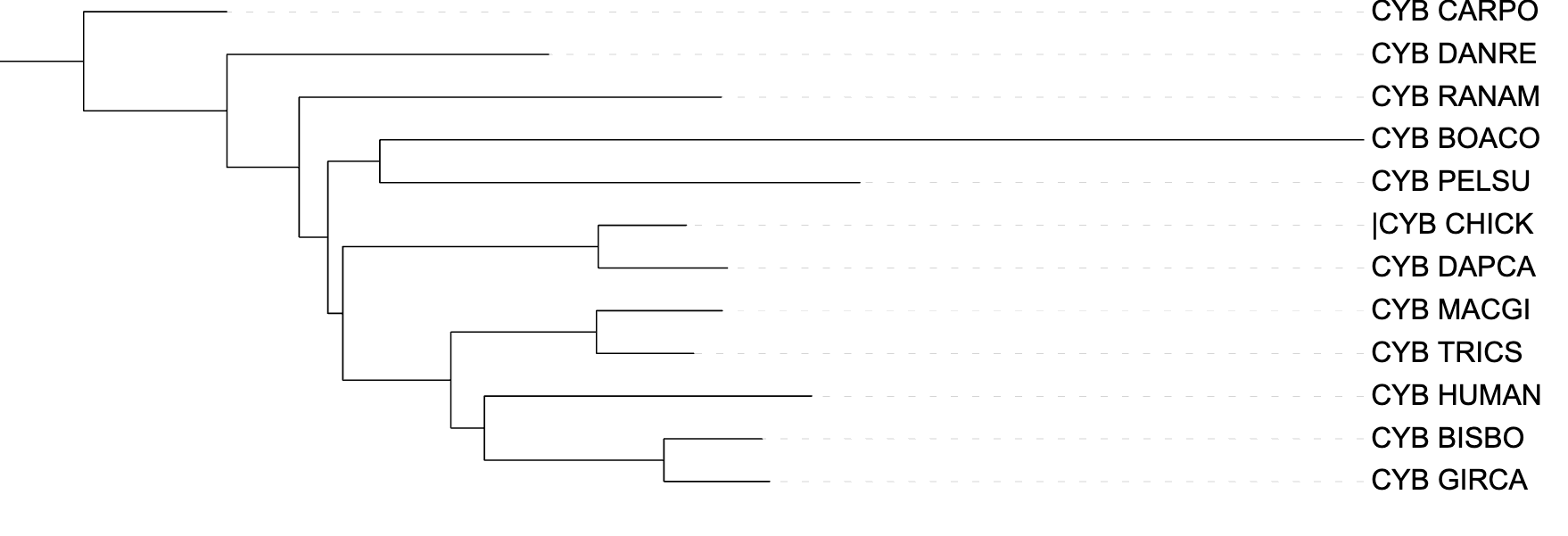
Изображение 3.Филогенетическое дерево, реконструированное на основе множественного выравнивания программой fastme с параметром -p MtREV

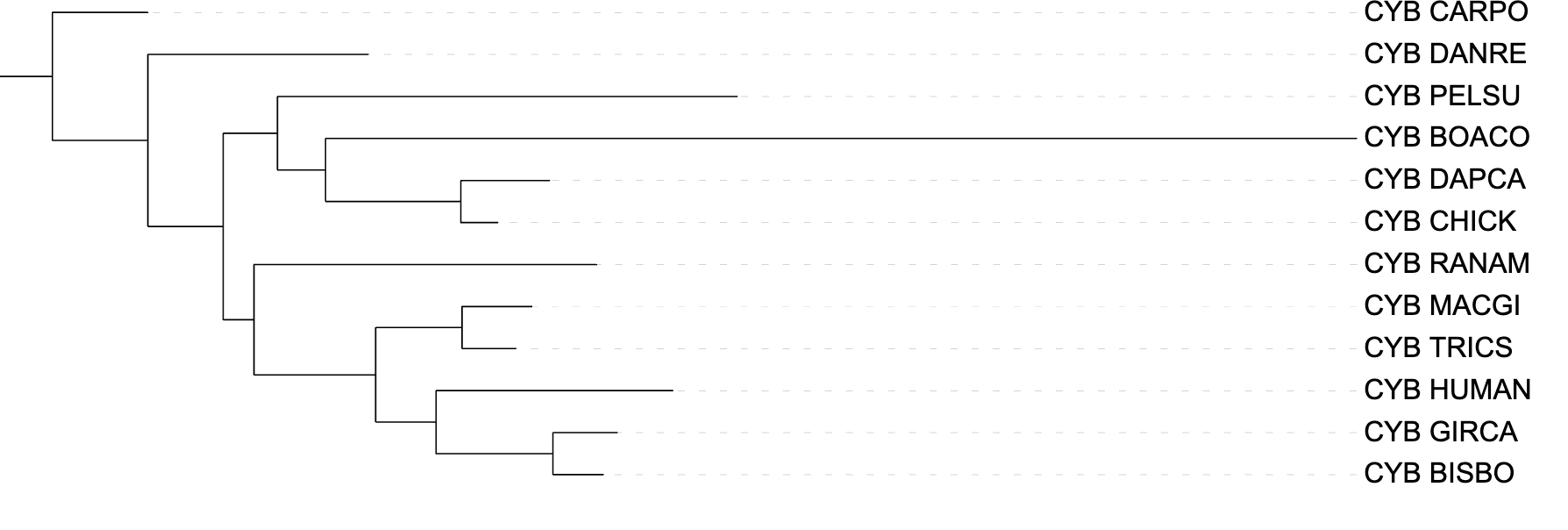
Изображение 4.Филогенетическое дерево, реконструированное на основе множественного выравнивания программой IQ-tree
Интерпретация результатов
Деревья были укоренены в определенные ветви в соответствии с изображением 1 по возможности. Деревья, реконструированные программой fastme (изображения 2, 3) c параметрами p-distance и mtrev соответственно, топологически идентичны дереву, рекоструированному на основе таксономической принадлежности организмов. Значительно от этих деревьев отличается дерево, для рекнострукции которого была использована команда iqtree (изображение 4). Это дерево меньше всего соответствует таксономической действительности. Здесь можно увидеть новую кладу ((((GIRCA, BISBO), HUMAN), (MACGI, TRCKS)), RANAM), а также ((DAPCA, CHICK), BOACO), а клада (PELSU, BOACO) - отсутствует.