Базы данных KEGG, GO и другие
Для анализа мне был предложен список следующих генов человека: FOLH1, GOT2, FOLH1B, GOT1, ASPG, ASNS, NAALAD2, GADL1, ASPA, SLC25A13, NAT8L, SLC25A12. Я решила предварительно не смотреть, что это за гены, и "вслепую" посмотреть (как-то странно звучит) их в базе данных STRING (потому что в презентации хорошо рассказано что именно там можно и нужно посмотреть).
STRING
Я вбила список генов в поисковую строку и сразу же получила таблицу 1, в которой напротив каждого гена обозначена его функция (удобно). Резюмируя, можно сказать, что все эти гены участвуютв метаболизме аминокислот (кажется только аспартата, глутамата, аспарагина и глутамина) Это не единственная их общая функция, но под эту категорию, кажется, подходят все гены. Также на изображении 1 представлен их, так называемый, граф взаимосвязанноти или граф белок-белковых взаимодействий.
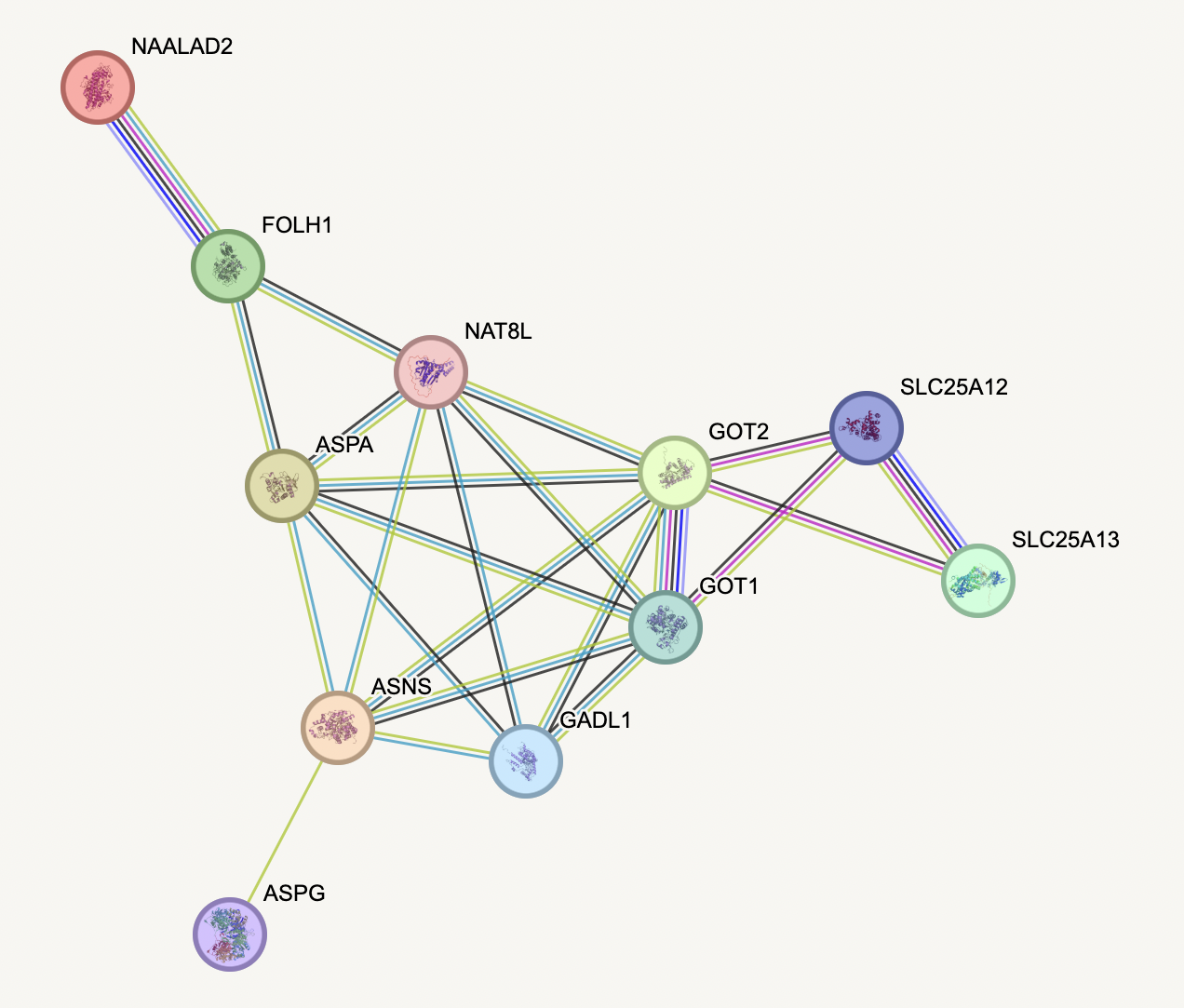
 Изображение белковой структуры в кружочках говорит нам о том, что для всех белков известна или хотя бы предсказана третичная структура. Ура. Так как граф достаточно понятный (имеет всего 10 вершин), можно рассмотртеь все его ребра, которые являются отображением взаимосвязи белков. Чтобы не быть голословной, ниже на изображении 2 представлена легенда для ребер графа.
Изображение белковой структуры в кружочках говорит нам о том, что для всех белков известна или хотя бы предсказана третичная структура. Ура. Так как граф достаточно понятный (имеет всего 10 вершин), можно рассмотртеь все его ребра, которые являются отображением взаимосвязи белков. Чтобы не быть голословной, ниже на изображении 2 представлена легенда для ребер графа.
 Дальше я решила немного поиграться с возможностями STRING и посмотреть, как будет выглядеть граф с применением кластеризации. А выглядеть он будет примерно так:
Дальше я решила немного поиграться с возможностями STRING и посмотреть, как будет выглядеть граф с применением кластеризации. А выглядеть он будет примерно так:
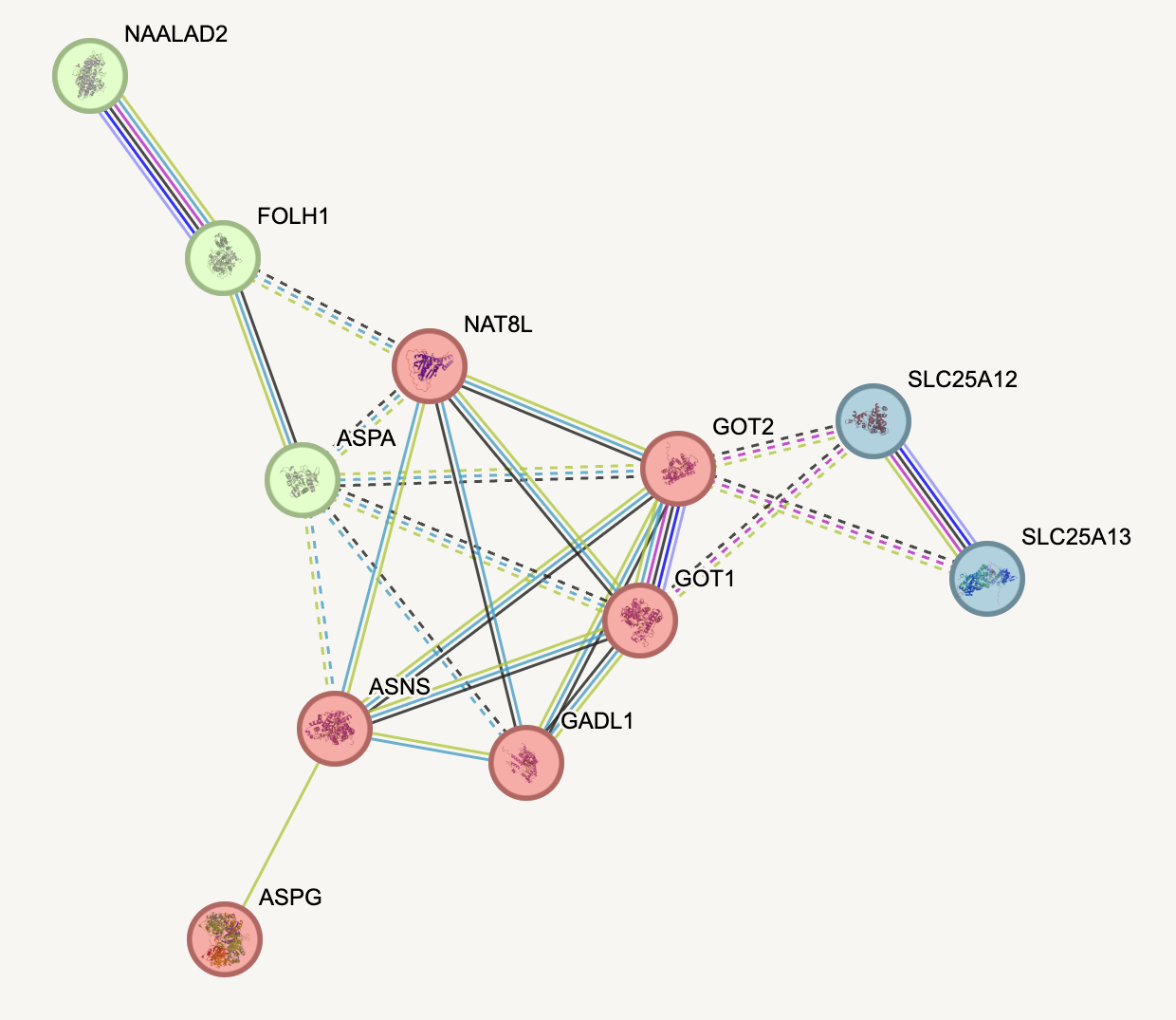 Белки красного кластера отвечают за метаболизм аспарагина и аспартата, белки желтого/зеленого кластера отвечают за... метаболизм аспарагина и аспартата (если честно я не очень поняла, почему они покрашены разными цветами. Возможно белки из разных кластеров хоть и участвуют в этом метаболизме, но выполняют глобально разные функции в этом механизме, либо же главное отличие в происхождении, строении, структуре и тд). И, наконец, белки синего кластера отвечают за несколько процессов сразу, но номером один в данном случае неожиданно стал... метаболизм аспартата и аспарагина. Также можно заметить, что между некоторыми белками из одного кластера наблюдается наибольшее число так назыываемых ассоциаций (см. Изображение 2). Наряду с этим, встречается и изолированная вершина графа, представленная белком ASPG, связанная с вершиной ASNS только найденной из источникв общей информацией (textmining).
Мое предположение насчет метаболизма аминокислот постепенно подтверждается, однако, кажется, я промахнулась с объектами этого биологического процесса. Чтобы окончательно убедиться в этом, посмотрим на изображение 4,5.
Белки красного кластера отвечают за метаболизм аспарагина и аспартата, белки желтого/зеленого кластера отвечают за... метаболизм аспарагина и аспартата (если честно я не очень поняла, почему они покрашены разными цветами. Возможно белки из разных кластеров хоть и участвуют в этом метаболизме, но выполняют глобально разные функции в этом механизме, либо же главное отличие в происхождении, строении, структуре и тд). И, наконец, белки синего кластера отвечают за несколько процессов сразу, но номером один в данном случае неожиданно стал... метаболизм аспартата и аспарагина. Также можно заметить, что между некоторыми белками из одного кластера наблюдается наибольшее число так назыываемых ассоциаций (см. Изображение 2). Наряду с этим, встречается и изолированная вершина графа, представленная белком ASPG, связанная с вершиной ASNS только найденной из источникв общей информацией (textmining).
Мое предположение насчет метаболизма аминокислот постепенно подтверждается, однако, кажется, я промахнулась с объектами этого биологического процесса. Чтобы окончательно убедиться в этом, посмотрим на изображение 4,5.
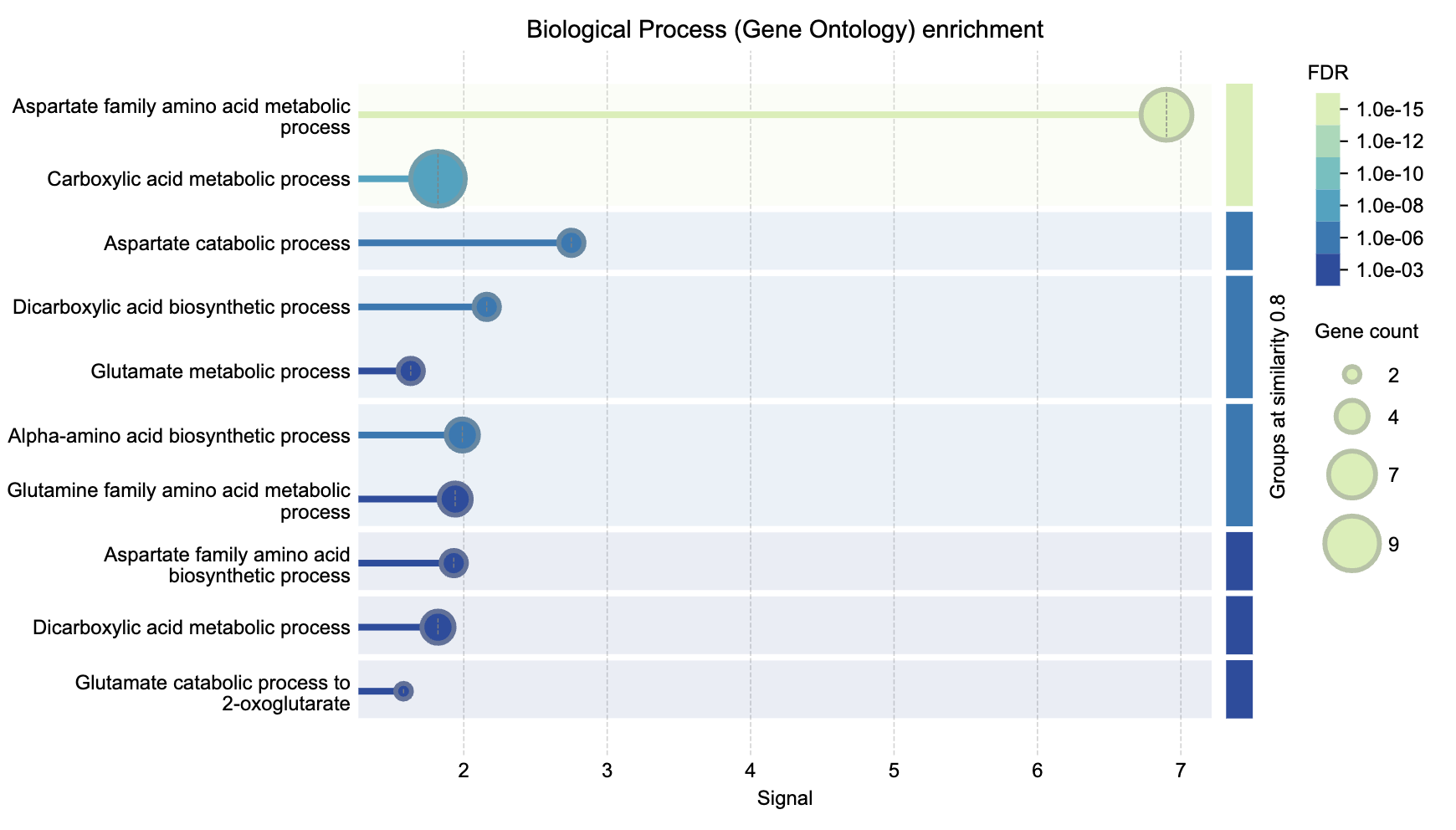
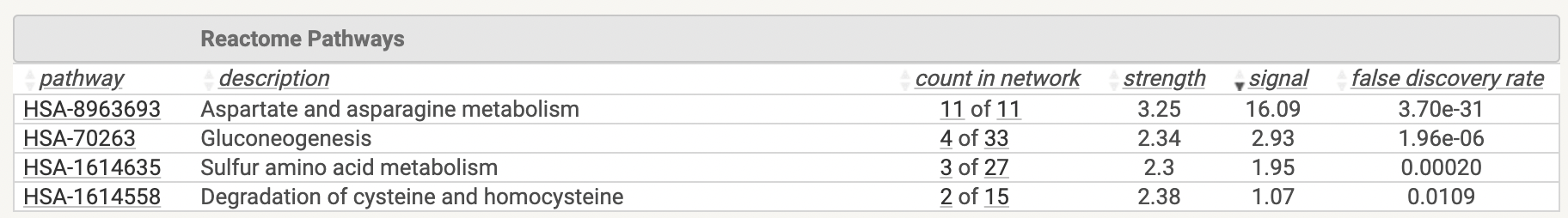 Из данных изображений видно, что все-таки 11 из 11 предложенных мне изначально генов (вообще их было 12, но почему-то рассматривается только 11. Почему так произошло - я могу только предполагать(нет, не могу)) участвуют в метаболизме аспартата и аспарагина.
Дальше мне просто стало интересно потыкать на другие разделы базы данных и мне, опять-таки, показалось интересным посмотреть, какие из представленных белков собираются в комплексы. Спойлер: белки практически одинаковыми названиями (см. изобрвжение 6).
Из данных изображений видно, что все-таки 11 из 11 предложенных мне изначально генов (вообще их было 12, но почему-то рассматривается только 11. Почему так произошло - я могу только предполагать(нет, не могу)) участвуют в метаболизме аспартата и аспарагина.
Дальше мне просто стало интересно потыкать на другие разделы базы данных и мне, опять-таки, показалось интересным посмотреть, какие из представленных белков собираются в комплексы. Спойлер: белки практически одинаковыми названиями (см. изобрвжение 6).
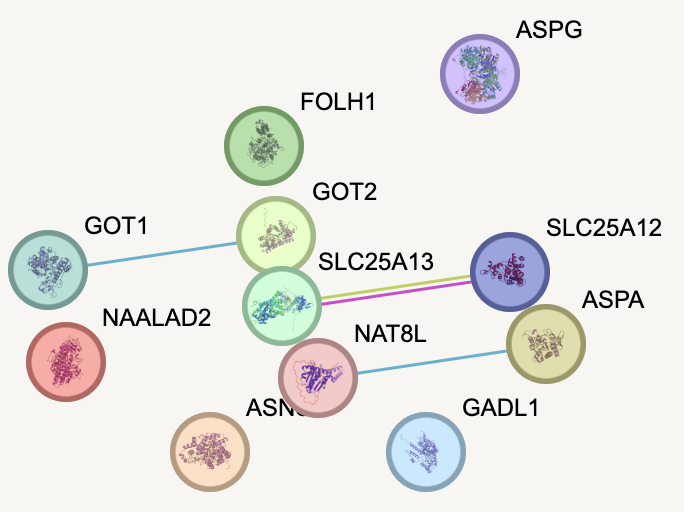

Изображение 1.Граф белок-белковых взаимодейтсвий. STRING

ну похоже же

Изображение 2.Цвета линий, из которых состоят ребра графа. Получился немного корявый перевод: под словосочетанием "совместное выражение" имеется в виду "совместная экспрессия", а "генная совместность" означает "совместное возникновение"

Изображение 3.Граф белковых кластеров

Изображение 4.GO-enrichment

Изображение 5.Статистика Reactome Pathways

Изображение 6.Белковые комплексы
The Human Protein Atlas
Для анализа генов в данной БД(база данных) была предоставлена возможность работать только с одним геном из предложенного списка. И кто я такая, чтоб этой возможностью не воспользоваться? Поэтому я решила анализировать ген ASNS (аспарагин синтетаза), потому что его функция показалась мне самой понятной. К тому же, он точно хорошо аннотирован, а структура и функции белка (ну я так считаю и надеюсь) хорошо изучены. В общей информации про ген есть пункт про цитоплазматическую экспрессию в нескольких различных типах тканей, включая поджелудочную железу, мозг и желудок. Можно было просто поверить на слово, но я решила посмотреть диаграммы экспрессии РНК и белка (изображения 7,8).
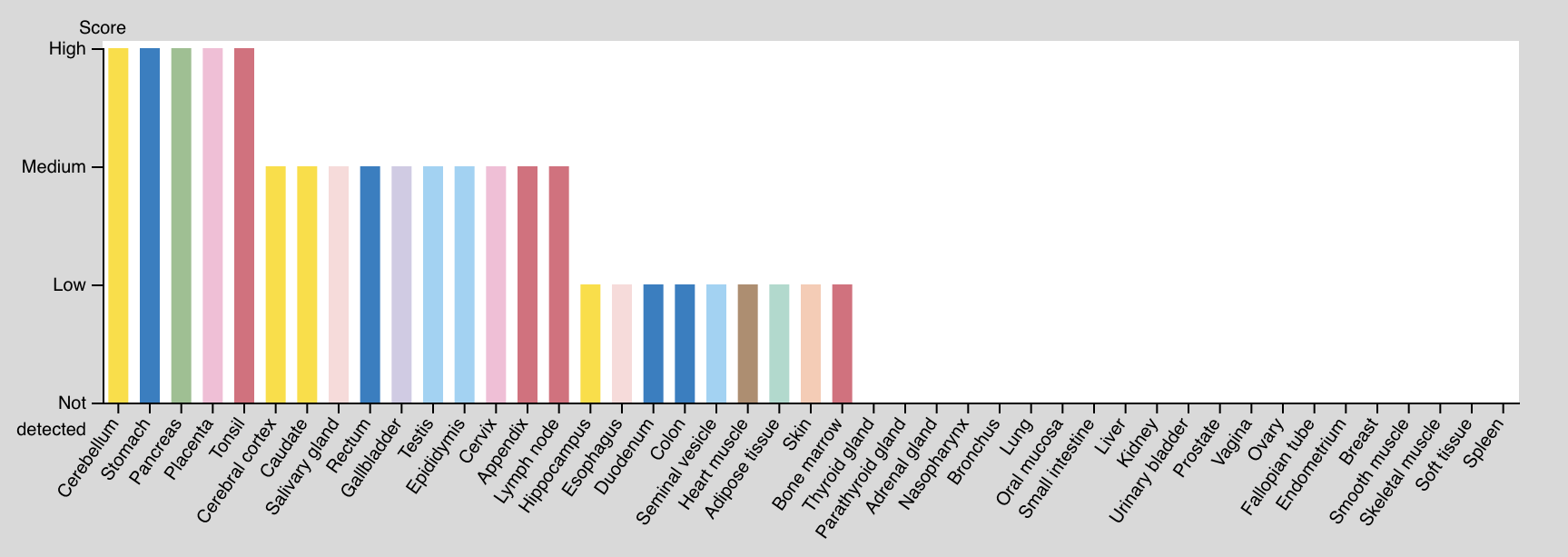
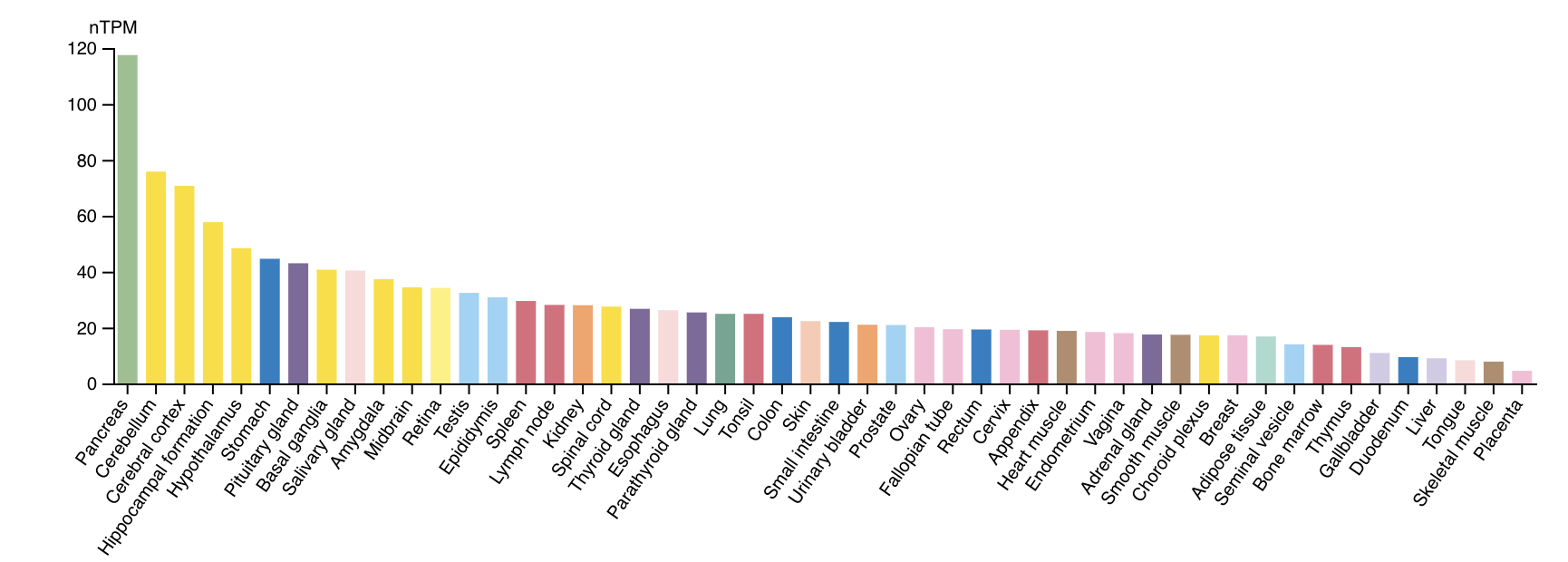 До просмотра данных диаграмм я думала, что уровень экспрессии РНК и белка в одних и тех же органах примерно одинаковый, но, видимо, я ошибалась. Сам белок экспрессируется только в части органов человека, в большинстве своем - в некоторых отделах мозга, в гормональных железах и в органах ЖКТ. РНК же эскпрессируется повсеместно, но в разном количестве. В целом, это все из максимально простого, интересного и поняятного об этом гене в данной БД.
До просмотра данных диаграмм я думала, что уровень экспрессии РНК и белка в одних и тех же органах примерно одинаковый, но, видимо, я ошибалась. Сам белок экспрессируется только в части органов человека, в большинстве своем - в некоторых отделах мозга, в гормональных железах и в органах ЖКТ. РНК же эскпрессируется повсеместно, но в разном количестве. В целом, это все из максимально простого, интересного и поняятного об этом гене в данной БД.

Изображение 7.Распределение уровня экспресии белка в организме челвоека

Изображение 8.Распределение уровня экспресии РНК в организме челвоека
Выше в анализе STRING были предприняты какие-то попытки анализа обогащения терминами, но я так и не поняла, достаточно ли этого, поэтому решила отдельно сделать данный анализ в БД Gene Ontology.
Хорошо, что я делаю этот анализ после всего вышеперечисленного, так как я уже точно знаю, в каком процессе участвуют данные гены, что между ними общего и так далее. Выдачу GO можно увидеть здесь. Пожалуй, главное, на чем здесь остается заострить внимание - коэффициент Fold Enrichment. Это значение >100 для 24 терминов(если я не сбилась при подсчете), то есть гены сильно связаны между собой некоторыми биологическими процессами, точнее - метаболизм аминокислот, а также значения p-value везде достаточно низки, что говорит о достоверности полученных результатов. Поэтому можно спокойно выдохнуть и в очередной раз убедиться, что мне попался не рандомный набор генов:)
Выводы
В результате проделанной работы с помощью БД String была получена основная информация о функциях генов из предложенного мне списка, упомянутого вначале, а так же преимущественно об их функциональной взаимосвязанности. Из этой информаци можно заключить, что исследуемые гены все участвуют в процессе метаболизма аминокислот (в основном - аспартата), а анализ обогащения терминами в БД GO лишь подтвердил это. Помимо этого, в БД Human Protein Atlas был рассмотрен отдельно ген ASNS (аспарагин синтетаза), а точнее была оценена экспрессия соответствующих ему РНК и белка в различных частях человеческого организма.


ну правда старалась...