Трансмембранные белки
Сравнение предсказаний трансмембранных (ТМ) участков в бета-листовом белке
Type: Transmembrane
Class: Beta-barrel transmembrane
Superfamily: Trimeric porins
Family: General Bacterial Porin
Species: Neisseria meningitidis
Localization: Bacterial Gram-negative outer membrane - внешняя мембрана грам-отрицательных бактерий
PDB Id: 7de8
UniProt Id: M4GGR4_NEIME
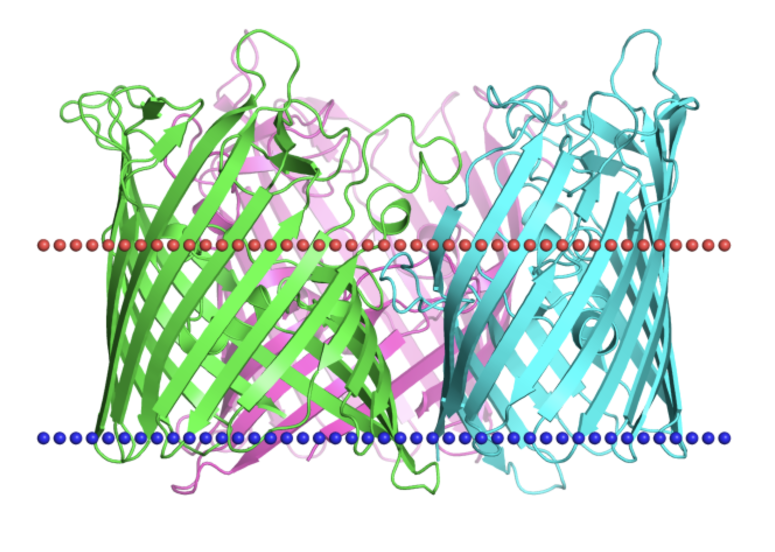
Изображение 1. белок PorB
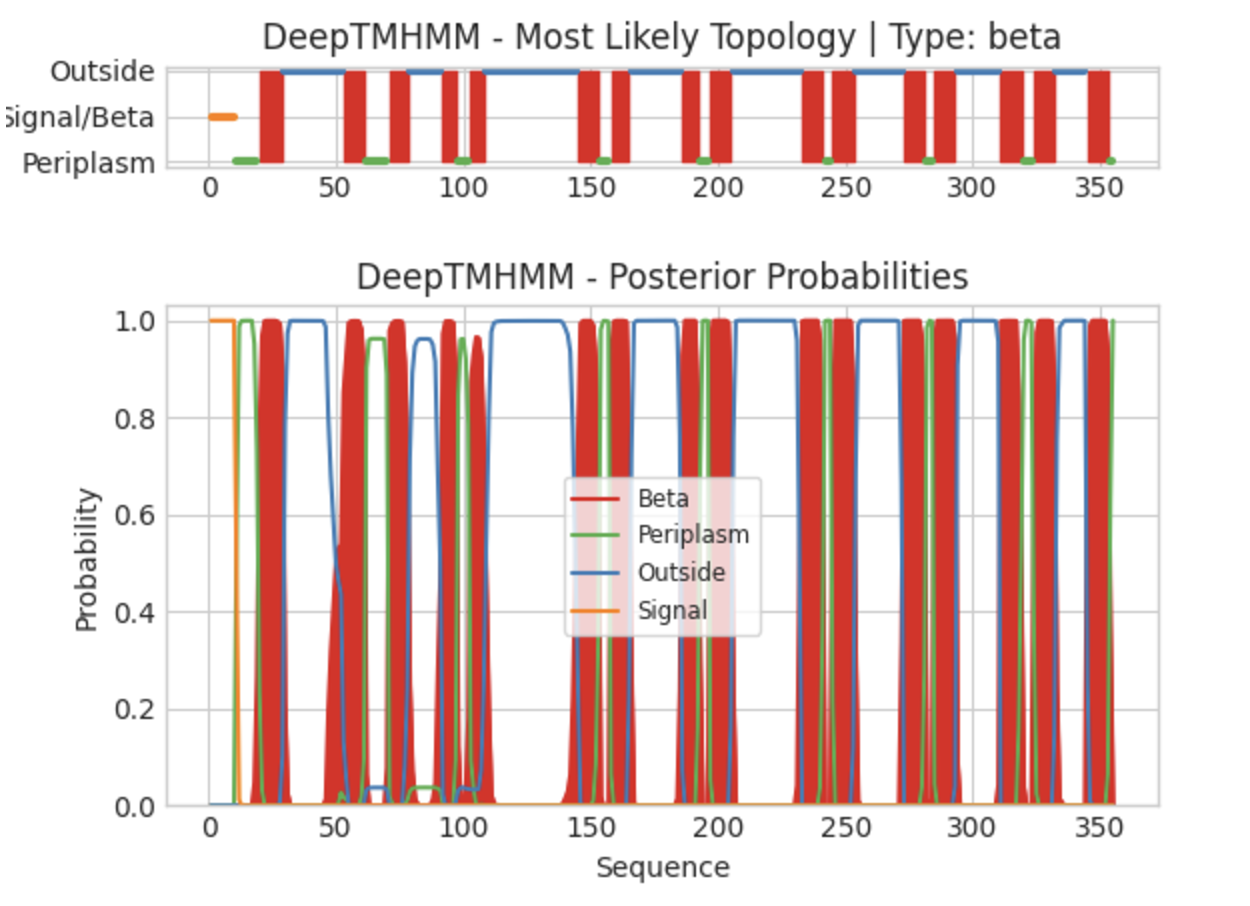
Изображение 2. Графическая выдача обработки последовательности белка PorB программой DeepTMHMM. На верхней части изображения представлена общая наиболее вероятная топология белка, а на нижней - уже топология с учетом вероятностей. Красным цветом изображены трансмембранные участки, зеленым - периплазматические, желтым - сигнальные, синим - участки наружней среды.

Изображение 3. Трансмембраные участки
Сравнение предсказаний трансмембранных участков в альфа-спиральном белке
Type: Transmembrane
Class: Alpha-helical polytopic
Superfamily: Disulfide bond oxidoreductase-B
Family: Disulfide bond oxidoreductase-B
Species: Escherichia coli
Localization: Bacterial Gram-negative inner membrane - внутренняя мембрана грам-отрицательных бактерий
PDB Id: 2leg
UniProt Id: DSBB_ECOLI
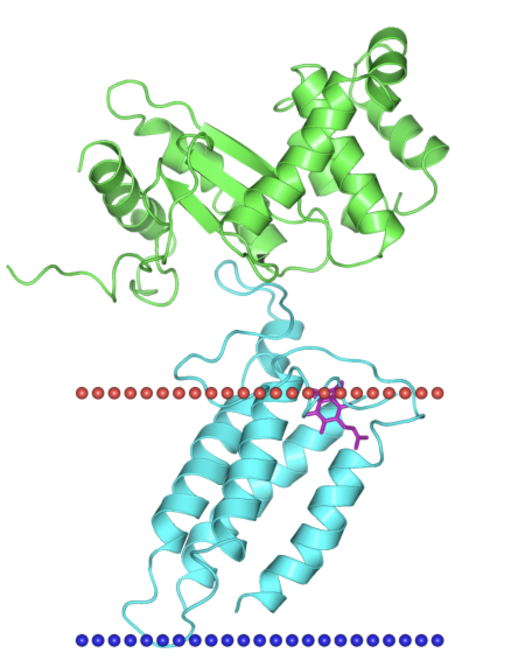
Изображение 4.Disulfide bond formation protein B (dsbB) - белок, участвующий в формировании дисульфидной связи в некоторых периплазматических белках, таких как PhoA или OmpA
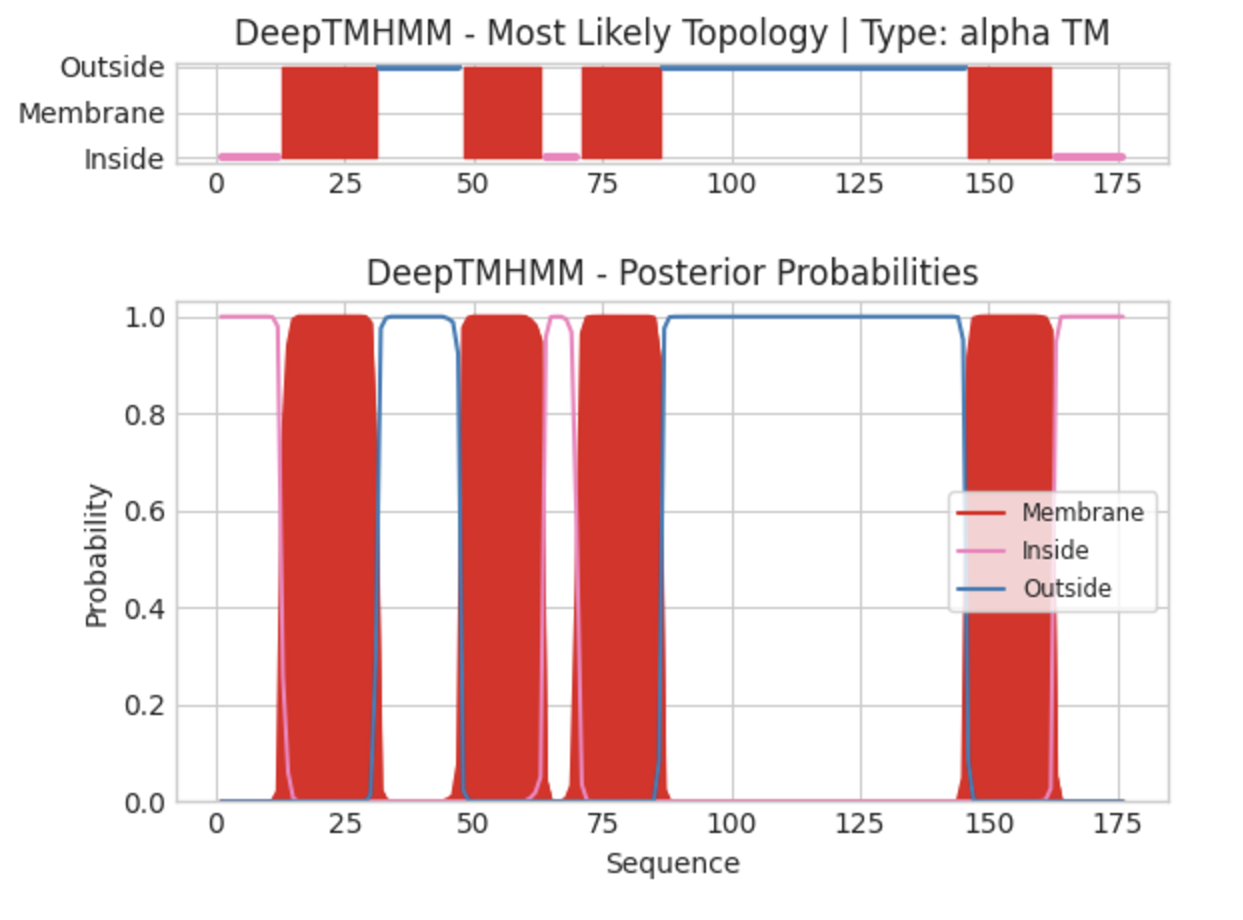
Изображение 5. Графическая выдача обработки последовательности белка dsbB программой DeepTMHMM. Красным цветом обозначены мембранные участки, розовым - учкастки внутри мембраны и синим - снаружи (по разные стороны мембраны).