Факторы транскрипции. Поиск de novo сигналов в ДНК
Подготовка данных
Группа положительного контроля - здесь для каждого CDS из генома было вырезано 32 нуклеотида перед старт-кодоном с учетом ориентации цепи. Fasta-файл с последовательностями.
Группа отрицательного контроля - здесь для каждого CDS из генома было вырезано 32 нуклеотида после старт-кодона с учетом ориентации цепи. Fasta-файл с последовательностями.
Тренировочная группа - здесь были отобраны из группы положительного контроля гены наиболее консервативных белков (гены, связанные с транскрипционными, трансляционными факторами, гены рибосомальных белков и тд), здесь ожидается найти нашу искомую последовательность или нечто схожее с ней (выявить паттерн и консенсус для бактерии Enterococcus lactis). Fasta-файл с последовательностями.
Поиск сигнала с помощью MEME
meme promotors.fasta -dna -nmotifs 2 -minw 5 -maxw 10
Команда выше была использована для поиска мотивов длины 5-10 нуклеотидов, так как у искомых сайтов консервативны участки -10 и -35 примерно такой длины. Были получены следующие результаты: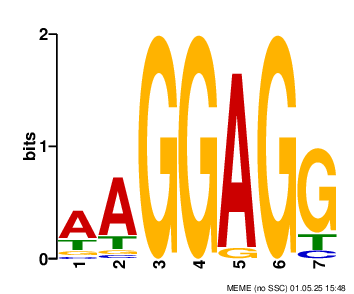
Изображение 1.Logo1, evalue=8.2e-045

Изображение 2.Logo2, evalue=2.9e-001

Изображение 3.Logo1-последовательность, комплементарная найденному мотиву

Изображение 4.Logo2-последовательность, комплементарная найденному мотиву
Поиск с помощью FIMO
fimo --oc fimo_pos -thresh 0.001 meme_out/meme.txt POSITIVE.fasta
fimo --oc fimo_neg -thresh 0.001 meme_out/meme.txt NEGATIVE.fasta
Cреди находок в положительном контроле обнаружилось 828 значимых находок, а в отрицательном - всего 44. Исходя из полученных данных можно предположить, что найденная ранее последовательность AAGGAGG является аналогом сайта посадки сигма-фактора РНК-полимеразы у бактерии Enterococcus lactis.