Геном бактерии Bartonella krasnovii кодирует 2 186 622 нуклеотида. Из таблицы 2 видим, что число адениловых и тиминовых оснований больше цитозиновых и гуаниновых. То есть в геноме больше двойных связей, чем тройных, более крепких. Основываясь на этом, можем выдвинуть гипотезу, что Bartonella krasnovii не является термофильной бактерией. Эта гипотеза согласуется с уже известными данными о том что Bartonella krasnovii является эндопаразитом животных и человека, температура внутренней среды которых редко превышает 40°C.
Краткий обзор генома бактерии Bartonella krasnovii
1 Аннотация
В данном обзоре будут рассмотрены базовые характеристики генома бактерии Bartonella krasnovii. Основное внимание будет уделено исследованию структуры и организации генома, то есть характеру расположения генов на плюс-цепи ДНК, их пересечениям, поиску гипотетических оперонов.
2 Введение
Род Bartonella включает в себя факультативные внутриклеточные грамотрицательные, гемотропные (то есть которые локализуются на поверхности эритроцитов), медленнорастущие, переносимые переносчиками бактерии. Дикие грызуны и их блохи являются пристанищем для большого разнообразия видов и штаммов рода Bartonella , включая несколько зоонозных (передающихся человеку от животных)[1].
3 Материалы и методы
3.1 Материалы
1 Аннотация
В данном обзоре будут рассмотрены базовые характеристики генома бактерии Bartonella krasnovii. Основное внимание будет уделено исследованию структуры и организации генома, то есть характеру расположения генов на плюс-цепи ДНК, их пересечениям, поиску гипотетических оперонов.
2 Введение
Род Bartonella включает в себя факультативные внутриклеточные грамотрицательные, гемотропные (то есть которые локализуются на поверхности эритроцитов), медленнорастущие, переносимые переносчиками бактерии. Дикие грызуны и их блохи являются пристанищем для большого разнообразия видов и штаммов рода Bartonella , включая несколько зоонозных (передающихся человеку от животных)[1].
3 Материалы и методы
3.1 Материалы
Данные по геному исследуемой бактерии были взяты с сайта Национального Центра Биотехнологической информации (NCBI), см S1 сопроводительных материалов.
Текст кодирующей последовательности генома:
GCF_003606345.3_ASM360634v3_genomic.fna GCF_003606345.3_ASM360634v3_cds_from_genomic.fna Таблица индивидуальных особенностей: GCF_003606345.3_ASM360634v3_feaures_table.txt
3.2 Методы
Таблицы индивидуальных особенностей были импортированы в Google sheets (см. S2 сопроводительных материалов). Мы составляли столбцы необходимых генов, белков и межгенных расстояний и с помощью определения карманов, необходимой для поставленной задачи длины, составляли диаграммы и гистограммы. Для составления значений для карманов использовались функции Google Sheets: =СЧЁТЕСЛИ, =СЧЁТЕСЛИМН, =МАКС, =МИН, =СРЗНАЧ.
4.4 Сравнение числа генов белков и генов различных РНК хромосомы и плазмиды pOE11-1 бактерии Bartonella krasnovii Был проведен анализ таблиц индивидуальных особенностей по столбцам features и class с помощью создания фильтров и контекстного поиска.
4.1 Нуклеотидный состав генома по нуклеотидам, 4.7 Нуклеотидный состав пересечений белок-кодирующих последовательностей Из таблицы индивидуальных особенностей были сделаны столбцы с пересечениями генов. С помощью языка программирования python Collab (см S3 сопроводительных материалов) мы проанализировали нуклеотидный состав пересечений и генома в целом.
4.8 Возможные опероны Вероятные опероны находились с помощью оценки расстояний между генами. Были удалены все расстояния между генами, превышающие 1 000 нуклеотидов, с сохранением порядковых номеров генов. На основе полученных расстояний между генами мы делали предположение о гипотетических оперонах и составлялись таблицы.
4 Результаты
4.1 Нуклеотидный состав генома по нуклеотидам
| Нуклеотид | Количество | % |
|---|---|---|
| A | 684943 | 31.32 |
| T | 668481 | 30.57 |
| G | 416643 | 19.05 |
| C | 416555 | 19.05 |
4.2 Распределение генов по плюс и минус цепям хромосомы и плазмиды
Геном Bartonella krasnovii представлен одной кольцевой хромосомой и одной плазмидой pOE11-1.
Из диаграммы (рис. 1)можно заметить, что на плюс-цепи хромосомы расположено больше генов, чем на минусовой, вероятно поэтому она и была выбрана основной при секвенировании генома.
У плазмиды pOE11-1 на рис. 2 мы наблюдаем совсем неровное распределение.
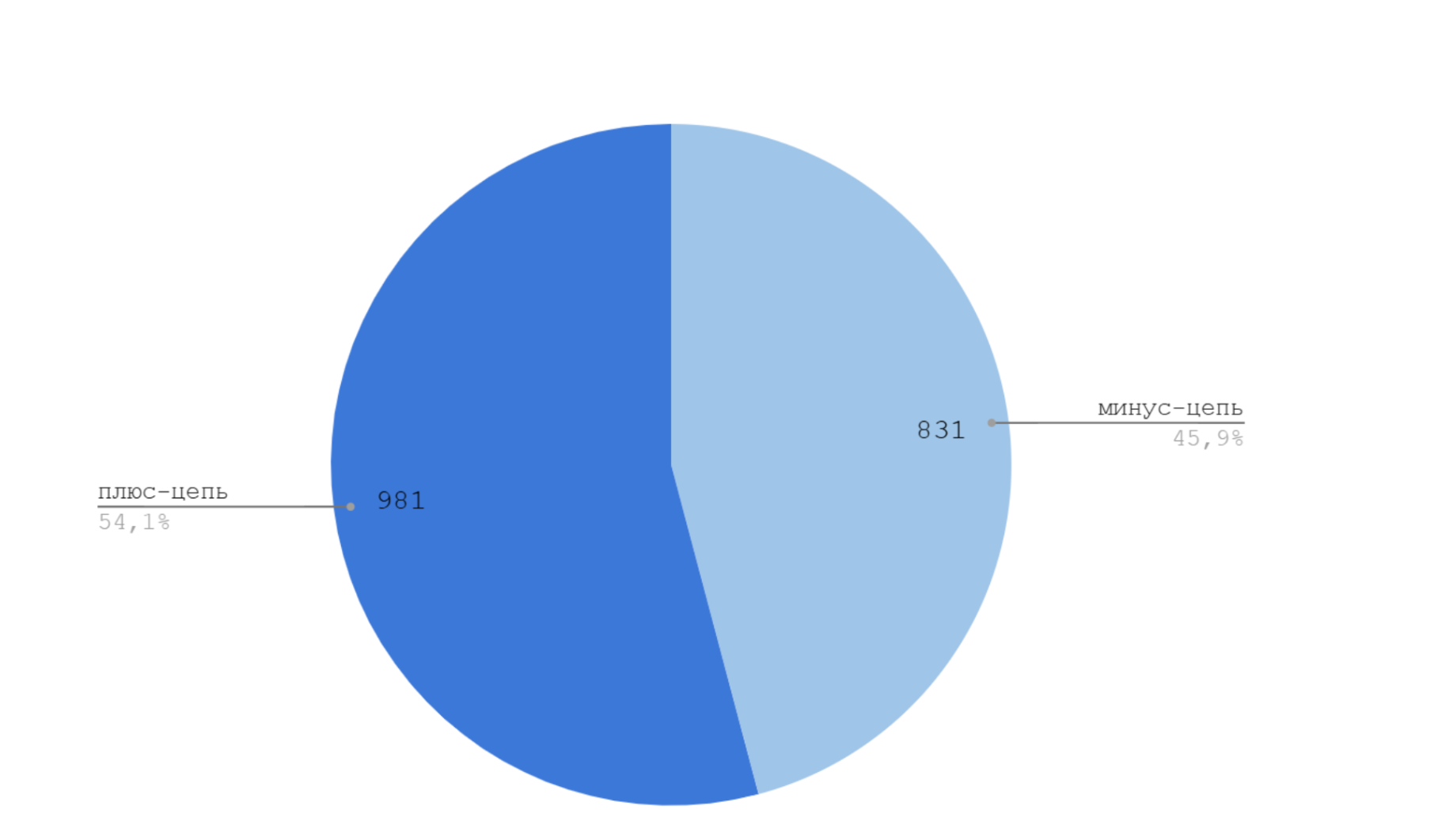
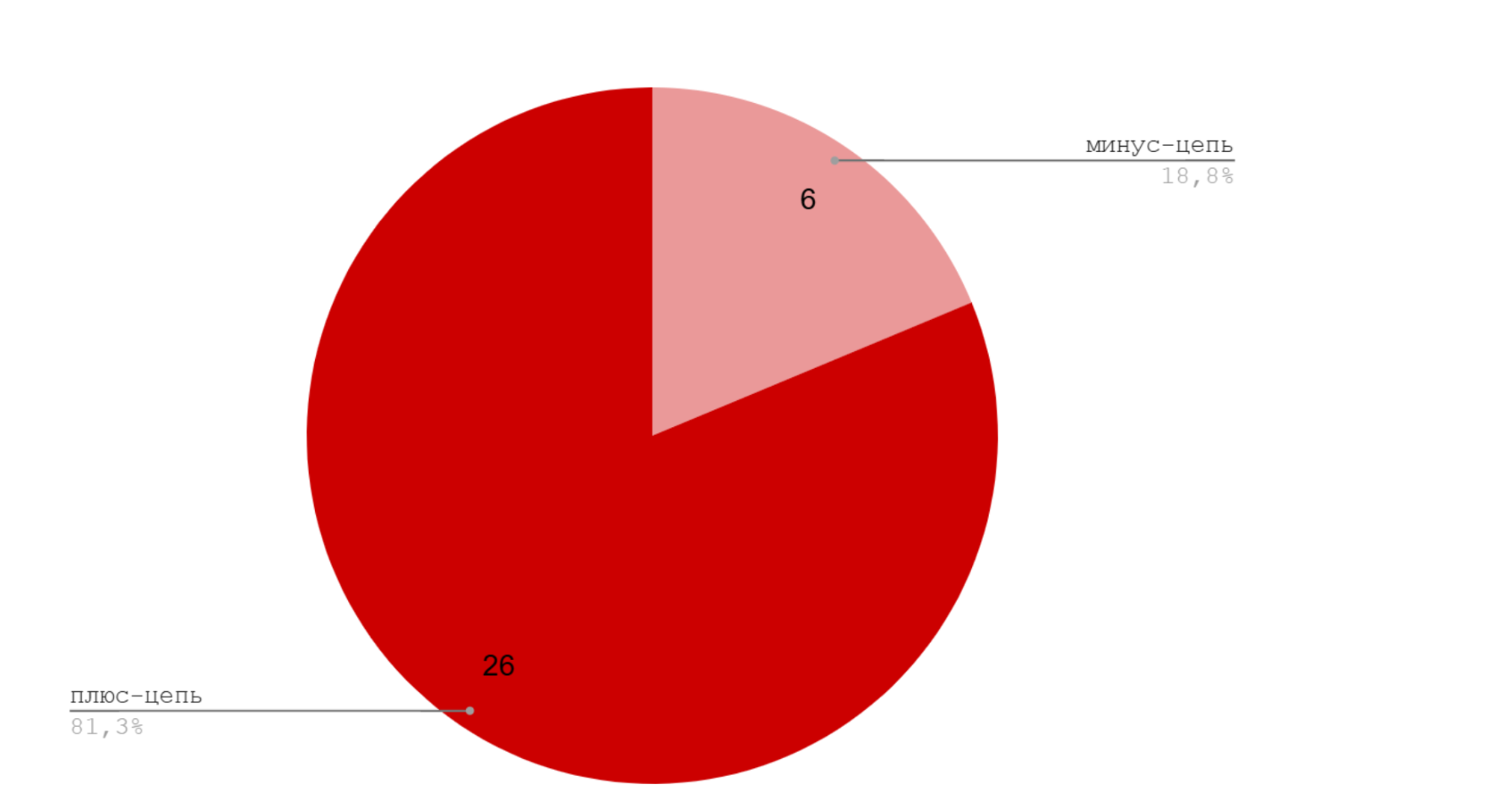
4.3 Длины белков, закодированных в геноме бактерии Bartonella krasnovii на (+) и (-) цепи
Было определено распределение количества белков в зависимости от их длины. Оно представлено в диаграмме (рис.3). Большинство белков длины от 40 до 480 аминокислот. Пик распределения — около 150 аминокислот в полипептиде.
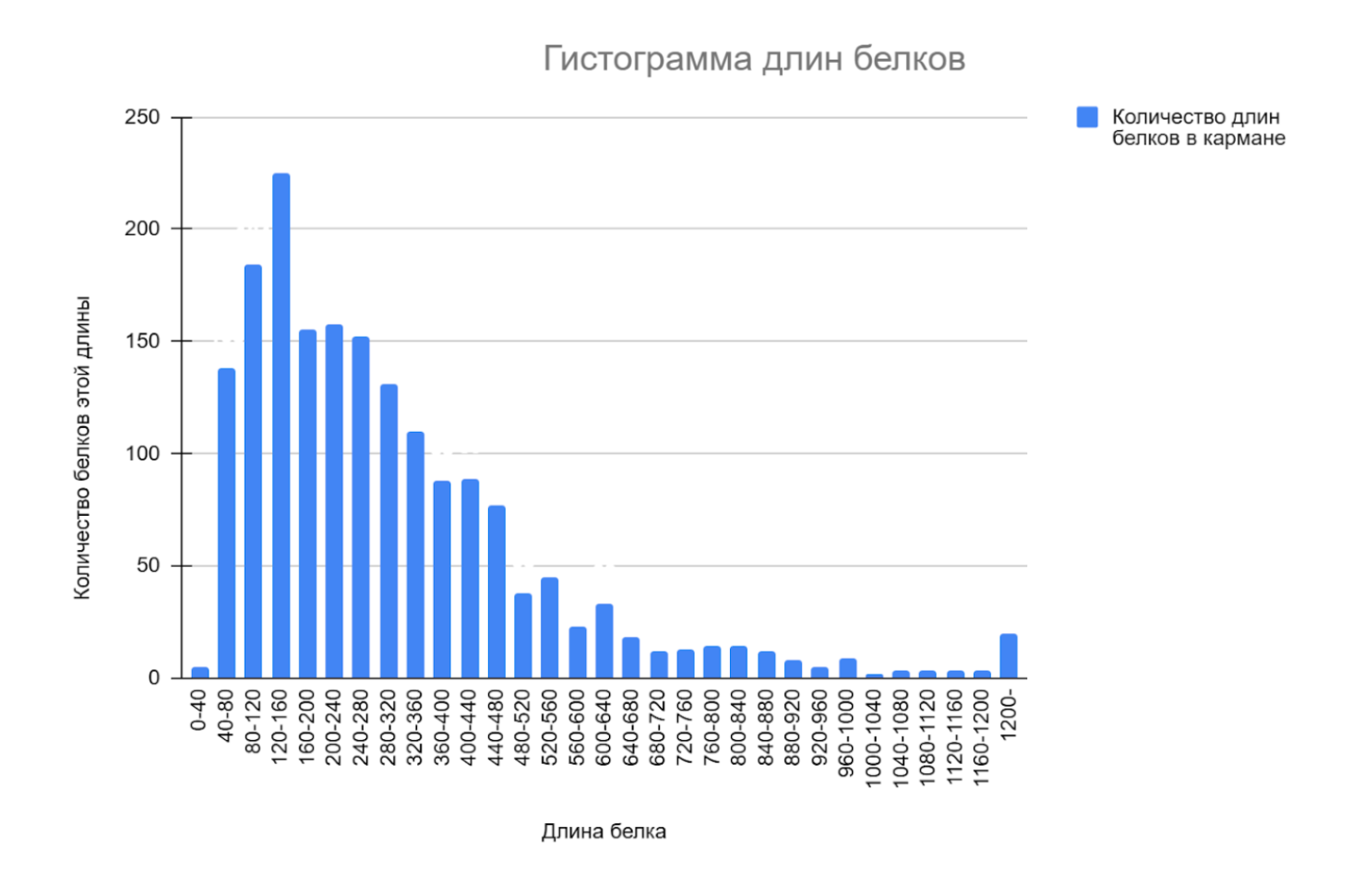
Самый короткий белок, не являющийся псевдогеном, — type II toxin-antitoxin system RelB/DinJ family antitoxin, длиной 36 аминокислот. Самый длинный белок — Vomp family autotransporter, длиной 4 237 аминокислот. Белки семейства Vomp — семейство белков наружной мембраны с различной экспрессией. Считается, что у рода Bartonella они способствуют адгезии. Бактерии часто изменяют экспрессию поверхностных адгезинов в зависимости от фазы или антигенной вариации, чтобы подорвать иммунный ответ хозяина и обеспечить адаптивное взаимодействие с различными структурами хозяина.
4.4 Сравнение числа генов белков и генов различных РНК хромосомы и плазмиды pOE11-1 бактерии Bartonella krasnovii
Как видно из таблицы 3, в плазмиде закодированы только белки. При том, что всего кодирующих последовательностей 1898, 98,37% содержит репликон основной хромосомы, а 1,63% содержит плазмида pOE11-1.
| Тип | Хромосома | Плазмида pOE11-1 |
|---|---|---|
| Белки | 1709 | 31 |
| Псевдогены | 81 | 0 |
| тРНК (tRNA) | 42 | 0 |
| рРНК (rRNA) | 6 | 0 |
| SRP RNA | 1 | 0 |
| Некодирующие РНК (ncRNA) | 1 | 0 |
| Транспортно-матричная РНК (tmRNA) | 1 | 0 |
| Рибонуклеаза P (RNase_P_RNA) | 1 | 0 |
Помимо обычных последовательностей, кодирующих белки, тРНК и рРНК, в геноме есть интересные виды РНК. Например, есть одна SRP RNA - The Signal Recognition Particle RNA ( частицы узнавания сигнала ). Только высвободившаяся из рибосомы последовательность синтезируемого белка распознаётся частицей SRP, которая затем связывается с малой субъединицей рибосомы, временно замедляет трансляцию на этой рибосоме и обеспечивает присоединение комплекса полипептид-рибосома к мембране шероховатого эндоплазматического ретикулума [3].
У B. krasnovii всего одна tmRNA (транспортно- матричная РНК).Она участвует в высвобождении рибосом, застрявших во время трансляции дефектных мРНК — транс-трансляция. В первую очередь она важна в стрессовых ситуациях, но у некоторых бактерий происходит и в нормальных условиях. [4] Также интерес вызывает Рибонуклеаза RNase_P_RNA. Она осуществляет эндонуклеолитическое расщепление РНК, удаляя лишние нуклеотиды на 5’- конце из предшественника тРНК. Она необходима для обработки тРНК, а также генерирует 5'-концы зрелых молекул тРНК [5].
Сопроводительные материалы
S1. Раздел на сайте NCBI о таксономической принадлежности бактерии Bartonella krasnovii. Taxonomy browser, Taxonomy ID: 2267275 https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=2267275
S2. Таблицы и построенные на их основе диаграммы и гистограммы: Гистограмма длин белков, гистограмма расстояний между белок-кодирующими последовательностями на + цепи, гистограмма количества нуклеотидов в пересечениях белок-кодирующих последовательностей на + цепи: Review materials
S3. Код в Colab с рассчётом количества нуклеотидов в геноме, нуклеотидного состава пересечений белок-кодирующих последовательностей: Review materials
Литература
[1]Ricardo Gutiérrez1ORCID icon, Tali Shalit, Barak Markus, Congli Yuan, Yaarit Nachum-Biala, Daniel Elad, and Shimon Harrus (25 February 2020). “ Bartonella kosoyi sp. nov. and Bartonella krasnovii sp. nov., two novel species closely related to the zoonotic Bartonella elizabethae, isolated from black rats and wild desert rodent-fleas Free” PMID: 32100689 DOI: 10.1099/ijsem.0.003952
[2] Peng Zhang, B. Chomel, +6 authors J. Koehler Published in Proceedings of the National Academy of Sciences of the United States of America 14 September 2004. “Semantic Scholar, A family of variably expressed outer-membrane proteins (Vomp) mediates adhesion and autoaggregation in Bartonella quintana”. DOI:10.1073/PNAS.0405284101,
[3] The Signal Recognition Particle (SRP) RNA Links Conformational Changes in the SRP to Protein Targeting Niels Bradshaw , Peter Walter , Received 2007 Mar 1; Revised 2007 Apr 25; Accepted 2007 May 3. PMCID: PMC1924838 PMID: 17507650
[4] Goodenbour JM, Pan T (29 October 2006). "Diversity of tRNA genes in eukaryotes". Nucleic Acids Research. 34 (21): 6137–6146. doi:10.1093/nar/gkl725. PMC 1693877. PMID 17088292.
[5] Marquez SM, Chen JL, Evans D, Pace NR (November 2006). "Structure and function of eukaryotic Ribonuclease P RNA". Molecular Cell. 24 (3): 445–56. doi:10.1016/j.molcel.2006.09.011. PMC 1716732. PMID 17081993.
[6] Anatoli Tchigvintsev,Dmitri Tchigvintsev,Robert Flick,Ana Popovic,Aiping Dong,Xiaohui Xu,Greg Brown,Wenyun Lu,Hong Wu,Hong Cui,Ludmila Dombrowski,Jeong Chan Joo,Natalia Beloglazova,Jinrong Min,Alexei Savchenko,Amy A. Caudy,Joshua D. Rabinowitz et al. (21 November 2013) “Biochemical and Structural Studies of Conserved Maf Proteins Revealed Nucleotide Pyrophosphatases with a Preference for Modified Nucleotides” PMID: 24210219 PMCID: PMC3899018 DOI: 10.1016/j.chembiol.2013.09.011