Сравнение программ выравнивания
Выбираем программы
Были выбраны следующие программы:
muscle -align PF05591.fa -output muscle.fasta
prank -d=PF05591.fa -o=prank.fasta
Программу prank мы берём как "плохую", а MSAProbs как "хорошую", поэтому сравним с ними полюбившуюся нам по прошлым практикумам программу muscle
Для сравнения была использована программа написанная Никой Bratz .
MUSCLE и MSAProbs
файл Jalview с выравниванием
Число последовательностей: 84
Длина выравнивания MUSCLE: 170
Длина выравнивания MSAProbs: 167
Процент совпадающих колонок в первом выравнивании: 87.65 %
Процент совпадающих колонок во втором выравнивании: 89.22 %
Число совпадающих блоков: 6
Совпадающие блоки при сравнении MUSCLE и MSAProbs:
| № | MUSCLE | MSAProbs | Длина |
|---|---|---|---|
| 1 | 1-15 | 1-15 | 15 |
| 2 | 18-39 | 18-39 | 22 |
| 3 | 45-51 | 45-51 | 7 |
| 4 | 55-90 | 55-90 | 36 |
| 5 | 100-162 | 97-159 | 63 |
| 6 | 164-166 | 161-163 | 3 |
Совпадающие колонки помимо блоков: (42,42)= (42,42) (53,53) = (53,53) (170,170) = (167,167)
Выравнивание MSAProbs короче, это было достигнуто за счёт меньшего количества гэпов. Также MSAProbs не допускает много колонк сгруппированных по 1-3, окруженные гэпами. Можно уверенно сказать, что MSAProbs выровнял лучше.
MUSCLE и PRANK
файл Jalview с выравниваниемКоличество последовательностей: 84
Длина выравнивания MUSCLE: 170
Длина выравнивания Prank: 190
Процент совпадающих колонок в первом выравнении: 77.65 %
Процент совпадающих колонок во втором выравнении: 69.47 %
Совпадающие блоки при сравнении MUSCLE и PRANK:
| № | MUSCLE | Prank | Длина |
|---|---|---|---|
| 1 | 1-14 | 1-14 | 14 |
| 2 | 17-36 | 17-36 | 20 |
| 3 | 50-61 | 54-65 | 12 |
| 4 | 64-84 | 68-88 | 21 |
| 5 | 102-162 | 119-179 | 61 |
| 6 | 164-165 | 181-182 | 2 |
PRANK справился хуже muscle. Участок 89-118 сделан очень плохо. PRANK сделал 10 лишних гэпов. Действительно плохая программа для множественного выравнивания.
Также можем заметить, что количество совпадающих блоков равно, но с MSAProbs их длина больше.
Выравнивание по совмещению структур
Для семейства установлены 3D структуры больших трубок из белковых комплексов, не видим больльшого смысла в том чтобы пытаться их совместить. Мы выбрали другое семейство - PF17887 Jak1 pleckstrin homology-like domain. Это домен который имеет отношение к янус-киназным рецепторам, которые распознают молекулы инсулина.
Сопоставлялись следующие белки: 5l04 - STRUCTURE OF INTERFERON LAMBDA 1 RECEPTOR WITH HUMAN KINASE JAK1 6e2p - Structure of human JAK2 FERM/SH2 in complex with Leptin Receptor 4po6 - Crystal structure of the human TYK2 FERM and SH2 domains with an IFNAR1 intracellular peptide
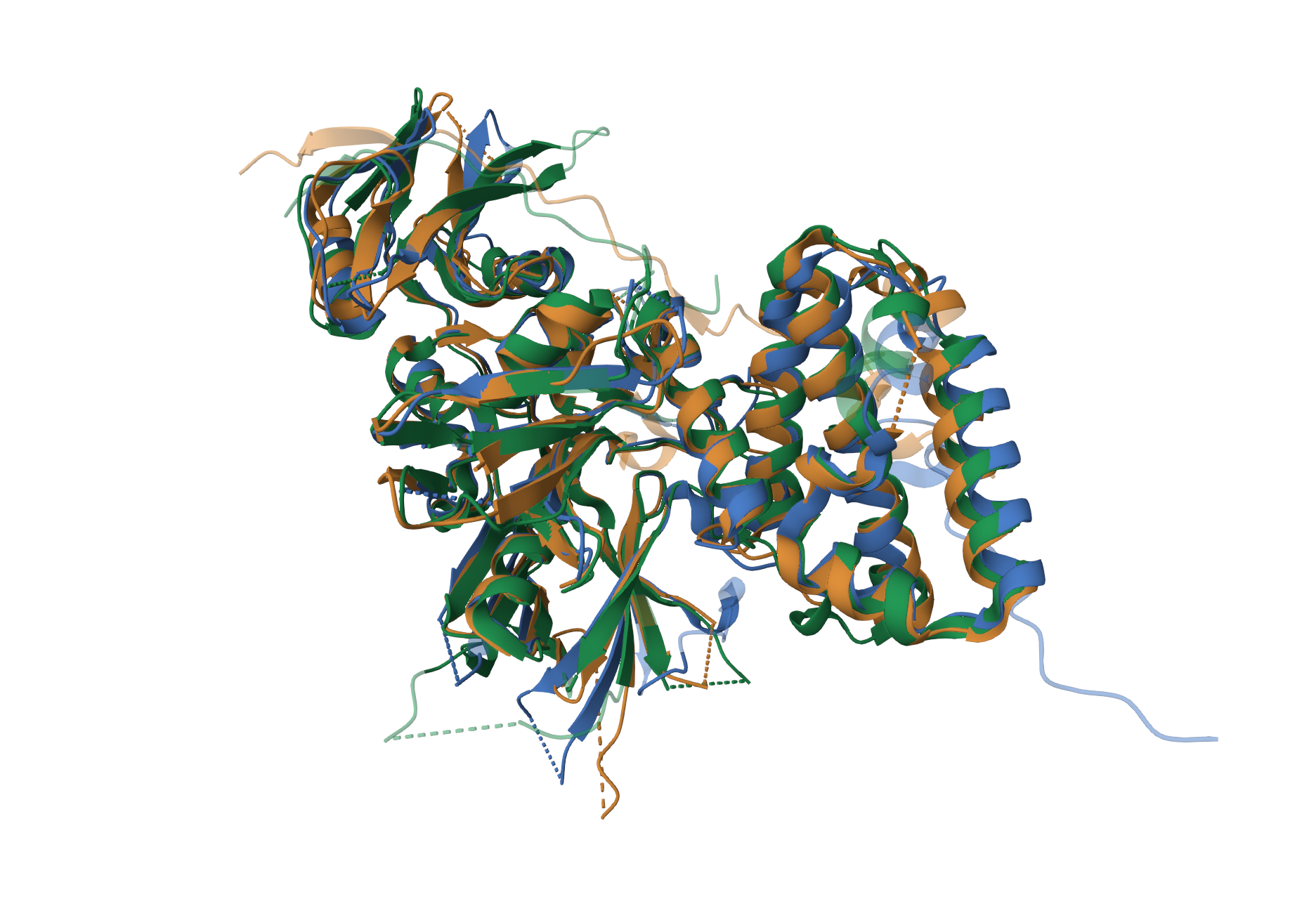
Число последовательностей: 3 Длина структурного выравнивания: 506 Длина выравнивания MUSCLE: 503 Процент совпадающих колонок в первом выравнении: 64.23 % Процент совпадающих колонок во втором выравнении: 64.61 % Число совпадающих блоков: 16
| Структурное выравнивание | MUSCLE | Длина |
|---|---|---|
| 3-8 | 3-8 | 6 |
| 110-133 | 109-132 | 24 |
| 142-144 | 141-143 | 3 |
| 146-170 | 145-169 | 25 |
| 174-175 | 173-174 | 2 |
| 177-214 | 176-213 | 38 |
| 217-218 | 216-217 | 2 |
| 224-250 | 223-249 | 27 |
| 267-270 | 266-269 | 4 |
| 276-288 | 274-286 | 13 |
| 295-300 | 292-297 | 6 |
| 304-320 | 301-317 | 17 |
| 324-397 | 321-394 | 74 |
| 399-400 | 396-397 | 2 |
| 403-422 | 400-419 | 20 |
| 449-506 | 446-503 | 58 |
Как можно понять из таблицы и файла Jalview, больше всего выравнивания отличаются на промежутке от 8 до 109 позиции. Здесь заметно, что у выравнивания muscle больше высоко идентичных колонок.
Описание MUSCLE
MUltiple Sequence Comparison by Log-Expectation - MUSCLE использует Итеративное рафинирование. Вкратце его можно поделить на 3 этапа: