Нуклеотидный BLAST
Ген, кодирующий δ-субъединицу АТФ-синтазы у сапсана
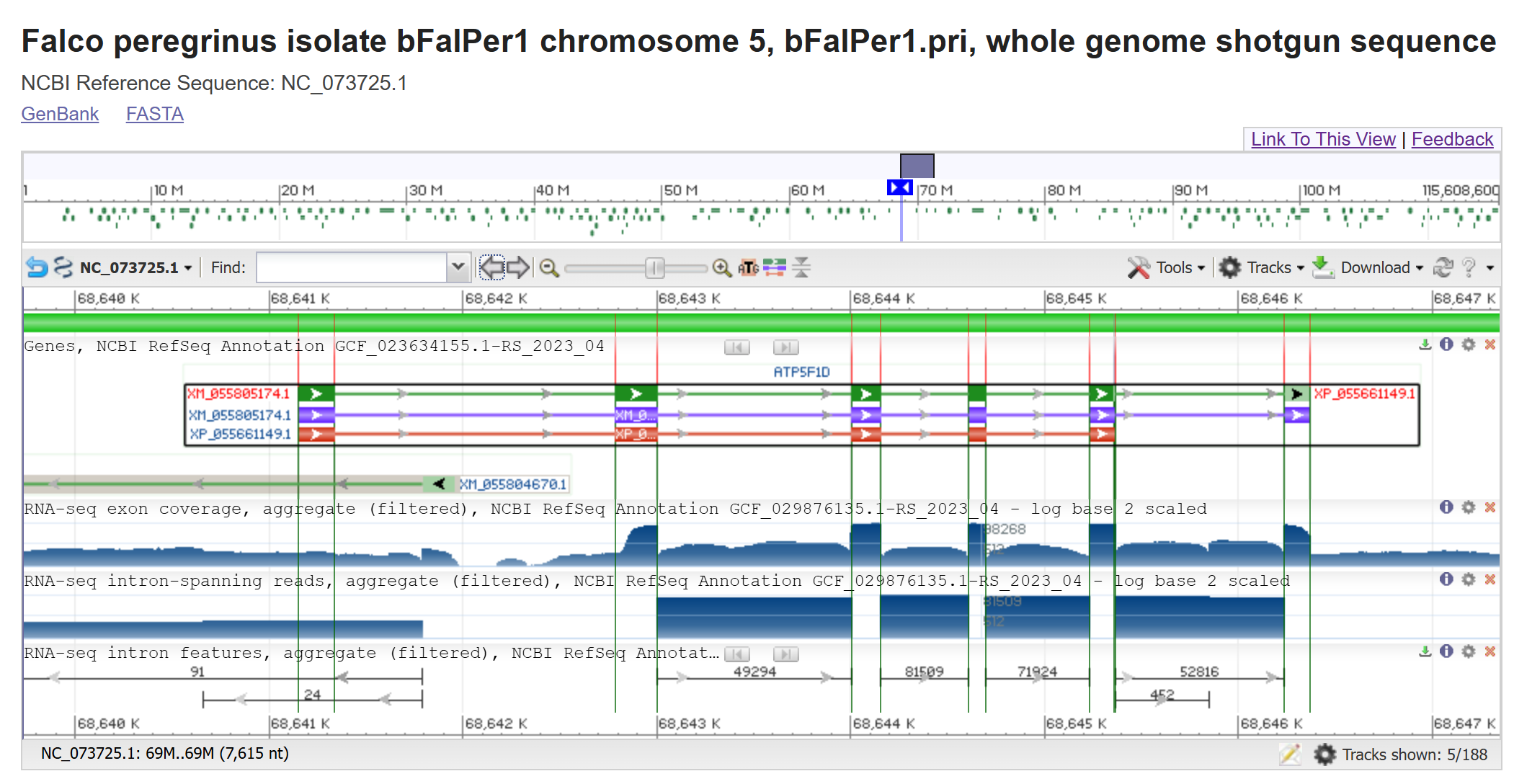
Идентификатор белка, ссылку на его последовательность: XP_055661149.1
Идентификатор нуклеотидной записи, к которой относится ген, кодирующий данный белок: LOCUS NC_073725
Координаты кодирующей белок части гена (Location): 68 641 151 .. 68 645 359
Ссылка на файл с последовательностью кодирующей белок части в FASTA-формате: Ген δ-субъединицы АТФ-синтазы
BLASTING
Будем сравнивать ген у отдалённых генетически летающих существ — нашего сапсана и пчёлок (Apoidea) по базе RefSeq Genome Database (refseq_genomes)
Так как megablast нужен для поиска почти идентичных последовательностей, есть сомнения в том что он предоставит хорошую выдачу. Воспользуемся сначала blastn. На вход он принимает нуклеотидную последовательность и сравнивает её просто с нуклеотидной базой данных. Априори, здесь должна быть небольшая выдача, т.к. мы сопоставляем только 2 последовательности без трансляции в возможный рамки считывания.
Всего две находки, удовлетворительное E-value только у первой.
Будем сравнивать с выдачей tblastx — он берёт на вход нуклеотидную последовательность, транслирует в шести рамках и сравнивает с нуклеотидной базой, последовательности в которой тоже транслируются в шести рамках. Поэтому выдача должна быть в разы больше, чем от blastn.
Выдача 100 хитов, в 50 раз больше чем от blastn, все находки с E-value ниже 0,01.
Из графического результата BLAST можем сделать вывод, что у гена есть два домена: средне консервативный и более консервативный домен ближе к концу последовательности.

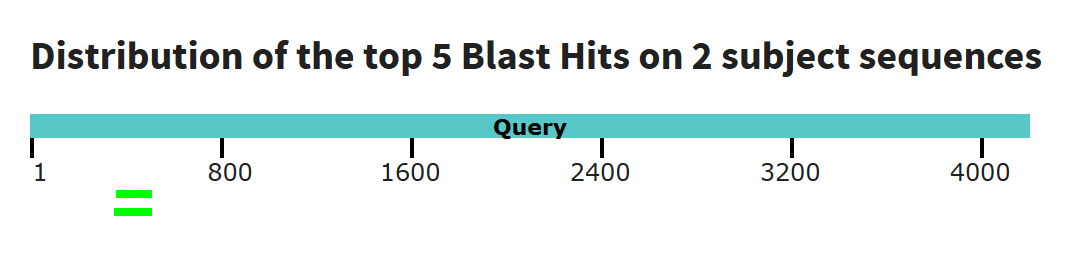

Гомологи рРНК в генетически далёком организме
Интересно попробовать поискать гомологию в консервативных учасках у совсем далёких оргинизмов. Аж из разных доменов. Наример: наша птичка сапсан и грам- бактерия Escherichia coli. Какие есть очень консервативные нуклеотидные последовательности? Конечно же рРНК! Вот их и попробуем выровнять - 16S и 23S рРНК прокариотика и последовательности нашего сапсана.
Мы скачали BLAST+ версии ncbi-blast-2.17.0+-win64. В консоли ввели команду
makeblastdb -in "C:\Users\mytia\Documents\ncbi_dataset\data\GCF_023634155.1\GCF_023634155.1_bFalPer1.pri_genomic.fna" -dbtype nucl -out falcon_db— получили индексированную базу под именем falcon_db
Гомологи 16S рРНК
Теперь надо найти гомологов рРНК у E. Coli. Будем делать поиск по очень далёким организмам, то есть megablast нам не подойдёт, на вход хотим давать нуклеотидную последовательность генома и искать по разным нуклеотидным последовательностям — нужен blastn:
blastn -task blastn -query "C:\Users\mytia\Documents\ncbi_dataset\data\GCF_023634155.1\16S.txt" -db falcon_db -out 16_blast.out -evalue 0.01 -outfmt 7— мы показали путь к файлу для которого надо искать по базе falcon_db, указали имя файла для вывода, пороговое E-value и тип вывода (табличка)
Blastn выдал 25 гомологичных учасков (hits), в 9 разных нуклеотидных последовательностях сапсана. % identity, alignment length, mismatches и участок гена рРНК от E.Coli который выравнивался (1494 — 1536) во всех находках одинаковые, отличается место на последовательности из нашей индексированной базы данных, к которой выровнялся участок. Это указывает на множественные копии или повторные элементы в геноме. Например на скэффолде NW_026599616.1 последовательность выровнялась 7 раз. И это при том, что у каждого выравнивания низкий е-value, что указывает на статистическую значимость.
Т.к. последовательность 16S рРНК выровнялась одним и тем же фрагментом к последовательности сапсана, то все 25 хитов можно считать гомологичными участками.
Проиллюстрируем один участок выравнивания на схеме:
CP014225.1:complement(926804-928359)|16S_rRNA|Escherichia NW_026599608.1 88.372 43 5 0 1494 1536 69205 69247 4.24e-05 56.3 CP014225.1:complement(926804-928359)|16S_rRNA|Escherichia NW_026599608.1 88.372 43 5 0 1494 1536 84501 84543 4.24e-05 56.3 CP014225.1:complement(926804-928359)|16S_rRNA|Escherichia NW_026599608.1 88.372 43 5 0 1494 1536 99829 99871 4.24e-05 56.3
Гомологи 23S рРНК
blastn -task blastn -query "C:\Users\mytia\Documents\ncbi_dataset\data\GCF_023634155.1\23S.txt" -db falcon_db -out 23_blast.out -evalue 0.01 -outfmt 7
- 111 попаданий всего, на 10 последовательностях (скэффолдах)
- Можно выделить ~25 гомологичных участков. Особенно много раз последовательность кишечной палочки выровнялась на участках [461299 - 496031], [2351651 - 2383878] и [1167435 - 1183321]. У последней находки слишком большой E-Value - 0.003, она скорее всего не является гомологом.
- Длина выравнивания: от 60 до 175 нуклеотидов
- Процент идентичности: от 67.28% до 81.75%
- E-value: от 2.82e-04 до 2.64e-17
- NW_026599616.1: 32 попадания — наибольшее кол-во hits
- NC_073722.1 , NC_073726.1 — выровнялись по одному разу
Видим, что попадания группируются в определенные регионы хромосом, наблюдаются множественные попадания в близких позициях с небольшими смещениями
Выводы
Среди 16S рРНК получилось заметно меньше находок. Это связано с тем, что 16S рРНК участвует в специфичных для прокариот процессах: 3′-конец содержит последовательность анти-Шайна-Дальгарно, с помощью которой 16S рРНК связывается с мРНК (которой нет у эукариот, у них elF4G узнаёт cap на мРНК); 3′-конец связывается с факторами, участвующими в инициации трансляции (S1 и S21). А 23S рРНК — это компонент большой (50S) субъединицы прокариотических рибосом, отвечающий за пептидилтрансферазную активность, то есть катализ образования пептидных связей при синтезе белка. Это менее специфичные для прокариот каталитические реакции, поэтому, вероятно, для 23S рРНК больше находок.
Карты локального сходства
Надо построить карты локального сходства по результатам поиска тремя программами: megablast, blastn, tblastx
Мы решили взять бактерию, с которой работали в первых двух семестрах — Bartonella krasnovii (NZ_CP031844.2) и бактерию того же рода Bartonella tribocorum (AM260525.1)
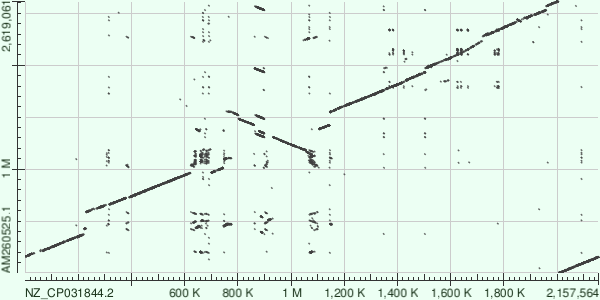
В центре явно видна инверсия. Blastn нашёл множество повторов на протяжении обоих геномов. Видно, что в последовательности генома второй бактерии AM260525.1 есть транслокации. Первый разрыв "главной диагонали" у AM260525.1 заполняют фрагменты из середины последовательности NZ_CP031844.2, которые затем повторяются и в середине последовательности AM260525.1. В правом нижнем углу виден участок, оказавшийся там в результате того, что кольцевые геномы секвенировались начиная с разных точек.
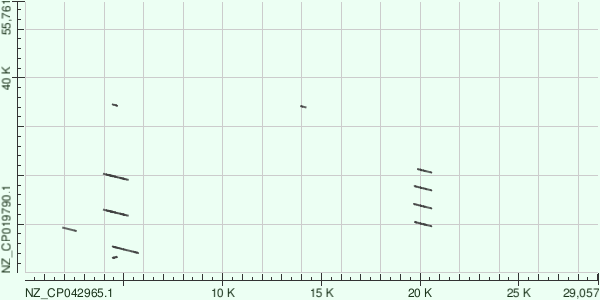
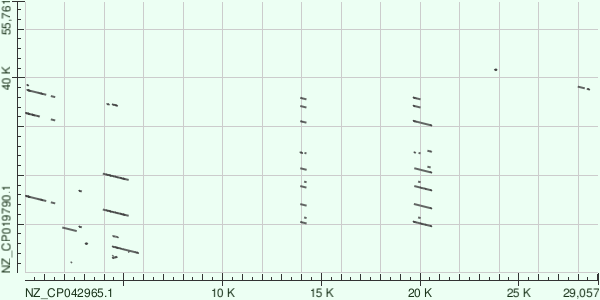
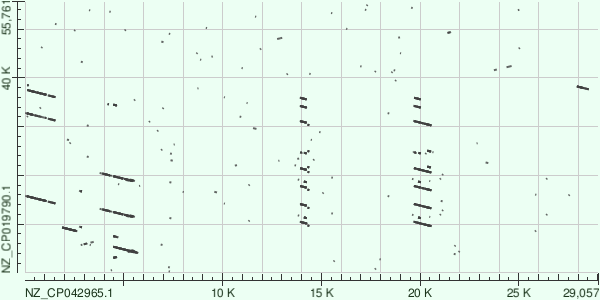
Сравнение плазмид
К сожалению, tblastx отказался выравнивать выбранные нами геномы, как и другие более менее длинные последовательности. Мы решили сравнить две плазмиды. Из нашей Bartonella krasnovii plasmid pOE11-1 (NZ_CP042965.1) и Bartonella schoenbuchensis R1 plasmid pVbh_BscR1 (NZ_CP019790.1)
Megablast нашёл полосы дублирующих фрагментов — 3 и 4 раза. Другие программы нашли ещё копии. Это наиболее интересные глобальные перестройки среди найденных последовательностей, которые смог выровнять tblastx.