Сборка генома de novo
Задание 1. Подготовка чтений программой trimmomatic
Таблица команд, используемых на данном этапе
| Input | Output | Command | What does it do |
| SRR4240389.fastq (weight=1387M), adapters.fasta (weight=) | task1.fastq (weight=1386M) | java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 SRR4240389.fastq task1.fastq ILLUMINACLIP:adapters.fasta:2:7:7 | Удаляем возможные остатки адаптеров, предварительно подготовив файл adapters.fasta с помощью
команды cat *.fa >> adapters.fasta Ридов было: 12950609 Ридов стало: 12947289 (99.97%) Ридов убрано: 3320 (0.03%) |
| task1.fastq (weight=1386M) | task1.1.fastq (weight=1386M) | java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 task1.fastq task1.1.fastq MINLEN:30 | Удаляем чтения длины менее 30; Ридов было: 12947289 Ридов стало: 12940837 (99.95%) Ридов убрано: 6452 (0.05%) |
Задание 2. Подготовка k-меров программой velveth
Была использована следующая команда:$ velveth task2.fastq 29 -fastq task1.1.fastq -shorttask2.fastq - output-директория (!), содержащая в себе несколько файлов:
Log, Rodmaps и Sequences
Задание 3. Сборка генома программой velvetg
Была использована следующая команда:$ velvetg task2.fastqN50: 3208
Max contig length: 14200
Задание 4. Анализ полученных контигов
Таблица по трем самым длинным контигам| Contig ID | Length | E-value | % Identity | Gaps / % | Chains | Chr start | Chr end | Part chr length | Read start | Read end | Part read length |
| 13 | 14200 | 0.0 | 77 | 206/2 | +/+ | 467412 | 474667 | 7256 | 5926 | 13237 | 7312 |
| 29 | 12664 | 0.0 | 79 | 97/4 | +/+ | 68262 | 70621 | 2360 | 1 | 2381 | 2381 |
| 22 | 10955 | 0.0 | 74 | 204/4 | +/- | 253244 | 257546 | 4303 | 6993 | 2691 | 4303 |
Комментарии
На рисунке по оси х распологается query - CP009253.1 (референсная хромосома), по оси y - контиги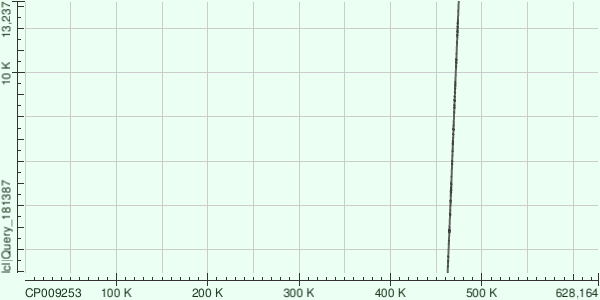
Участок контига 13 достаточно хорошо выравнивается на референсную хромосому;
Цепи соответствуют друг другу по направлению, что можно видеть из приведенного выше рисунка
Кроме того, на графике нет каких-либо разрывов, что говорит о целостности участка соответствия
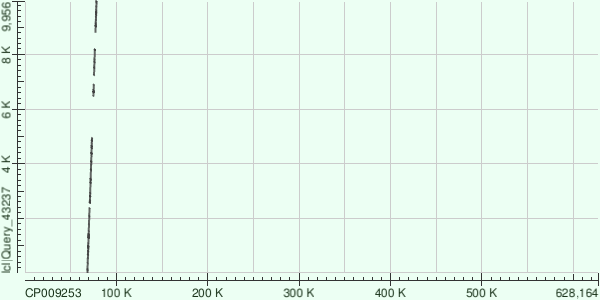
На карте мы видим несколько разделенных разрывами участков; действительно, выдача blast
дает нам несколько выравниваний с подряд идущими координатами (ниже представлены координаты референсной хромосомы):
68262 70621 (len = 2360)|+ 349 => 70970 73310 (len = 2341)|+ 1523 => 74833 75264 (len = 432) |+ 264 => 75528 76468 (len = 941) |+ 649 => 77117 78277 (len = 1161)Мы видим, что координаты участков, их длины соответствуют тому, что мы видим на карте сходства:
Сначала - два идущих подряд участка примерно одинаковой длины (2360 и 2341) с неболшим (349) расстоянием
между ними; после следует большой непокрытый участок (1523) с двумя небольшими учатками соответствия, разделенных
относительно небольшим промежутком
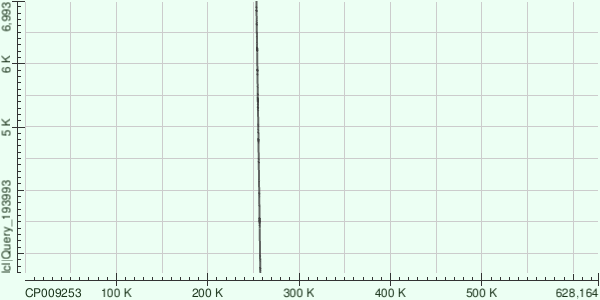
По рисунку, приведенному выше, мы видим, что цепи референса и нашего контига, противоположнонапрвленные
Это может быть связано с тем, что произошла инверсия (но мы не можем точно говорить об этом, так как в процессе
сборки цепь определяется случайным образом)
Так же, как и в первом случае, мы видим непрерывный участок соответствия контига 22 участку
бактериальной хромосомы
Среднее значение параметра % Identity равно 76.67 %
E-value = 0.0 во всех трех контигах
Так, мы можем сказать, что геном собран хорошо