Чтение последовательностей по Сэнгеру
Задание 1. Отчёт о проблемах при чтении хроматограмм
Последовательность в fasta-формате (прямая цепь)JalView-проект с выравниванием прямого и обратного прочтений
Ссылки на исходные файлы в .ab1 формате:
Прямая последовательность
Комплементарная обратная последовательность
Для анализа было представлено две последовательности:
1) WS2980_H3_F_A03_2013-06-11-22-09-18.ab1
2) WS2980_H3_R_A04_2013-06-11-22-09-18.ab1; R - "reverse"
Для последней была применена команда reverse complement (мы перешли к комплементарной цепочки).
Стоит заметить, что таким образом конец прочтения окажется в начале "перевернутой" хроматограммы.
В последствии будем пользоваться нумерацией для полученной комплементарной цепи
Для таких цепочек было произведено "выравнивание" вручную (методом рассмотрения двух окошек Chromas
с поиском соответсвующих участков подпоследовательности.
Укажем первоначальную нумерацию участков соответствия (до удаления нечитаемых концов):
Для WS2980_H3_F_A03_2013-06-11-22-09-18.ab1 участок, подходящий для выравнивания принадлежит
следующему "отрезку": 5' 19(Т) - 320(C) 3'
Для WS2980_H3_R_A04_2013-06-11-22-09-18.ab1 участок, подходящий для выравнивания принадлежит
следующему "отрезку": 50(Т) - 352(С)
Границы нечитаемых 5'- и 3'-участков:
WS2980_H3_F: 1 - 18 (с помощью опции Continuous edit удалим также нуклеотиды 359 - 361
как неопределенные но, по сути, есть только один пик, неразличимый с шумом, причем последующие
два пика, хорошо выраженные, обозначаются как неопределенные)
WS2980_H3_R: 322 - 352 (координаты определим по прямой последовательности - WS2980_H3_F)
Теперь, когда у нас изменилась нумерация последовательностей после удаления нечитаемых концов,
укажем новые номера участков соответствия:
1 - 302 (по прямой последовательности)
50 - 352 (по комплементарной обратной)
Как мы видим, длина участков соответствия различается на 1 (об этом будет написано позже)
Если говорить о качестве хроматограммы прямой последовательности, то стоит отметить, что до 29
нуклеотида (включительно), даже после применения опции Continuous edit присутствуют некоторые
неточности при выявлении пиков (на этом участке они сливаются, как, например, область нескольких
идущих подряд А: 23 - 26). В целом, в данной хроматограмме приходится ~ 2.14 неопределенных пиков
на каждые 50 нуклеотидов (высокий уровень шума). За исключением таких участков общий уровень шума
достаточно низкий. На участке 84 - 356 пики достаточно хорошо идентифицируемы, за исключением упомянутых
выше неоднозначных областях.
Таким образом, "на глаз" мы видим 4 - 5% неоднозначных пиков. Охарактеризуем качество хроматограммы как
достаточно высокое
Теперь рассмотрим диаграмму комплементарной обратной последовательности.
В этой последовательности на каждые 50 нуклеотидов приходится примерно 2 неоднозначных пика.
После 300 нуклеотида появляются проблемы в выделении индивидуальных пиков, соседние начинают
сливаться (к примеру, пересечение нескольких пиков 302 - 306 или область "попарных" слияний пиков - 342 - 347).
Достаточно высокий уровень наблюдается на отрезке 347 - 352, 3 - 6.
"На глаз" мы видим 5 - 6% неоднозначных пиков (учитывая участки пересечений). В целом уроень шума достаточно
невысокий.
Редактирование прямой последовательности
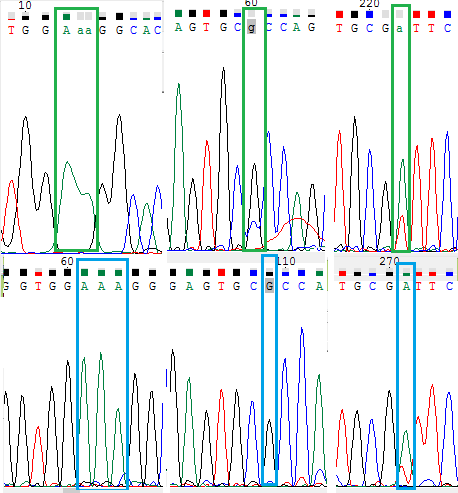
1) Добавлены А в позиции 13, 14 прямой последовательности (на основе сравнения с комплементарной обратной,
см. Рис ); произошло слияние нескольких пиков в в один, в результате чего 2 из 3 не были идентифицированы
2) G в позиции 60 (на основе сравнения со второй последовательностью)
3) A в 222 позиции (на основе сравнения со второй последовательностью)
Редактирование обратной комплементарной последовательности
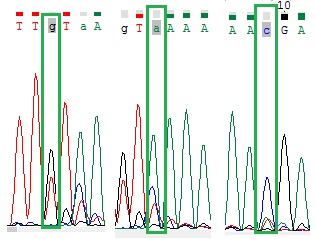
0.1) В позиции 3 наблюдается достаточно высокий черный пик (G), но этот нуклеотид обозначен как неопределенный;
вместо неопределенного добавлен G
0.2) В позицию 5 добавлен А (преобладающий по высоте зеленый пик)
0.3) В позицию 9 добавлен С (преобладающий по высоте синий пик)
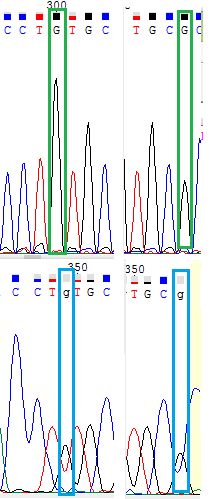
1) В позицию 349 добавлен G на основе сравнения с прямой последовательнотью
2) В позицию 353 добавлен G на основе сравнения с прямой последовательностью; если отпустить прямую, то ее
основание придется на локальное повышение черного пика над синим
Исправление пиков по одной последовательности
Исправление пиков по двум последовательностям
Выписка по выравниванию с помощью программы needle:
# Aligned_sequences: 2
# 1: 2980
# 2: 2980
# Matrix: EBLOSUM62
# Gap_penalty: 10.0
# Extend_penalty: 0.5
#
# Length: 408
# Identity: 302/408 (74.0%)
# Similarity: 302/408 (74.0%)
# Gaps: 104/408 (25.5%)
# Score: 1906.0
Предположительно, это может быть Psolus phantapus isolate D histone H3 (H3) gene, partial cds (с помощью
blast прямой последовательности); e-value = 4e-155 (97% Identities)
С помощью программы consambig из пакета EMBOSS была получена консенсусная последовательность:
>EMBOSS_001 ttntnaaangacggccagtatggctcgtaccaagcagacngcacgtaaatYYWMCSGKKG RANGGCACCGCGAAAACAACTGGCCACCAAGGCAGCCCGMAAGAGTGCNCCAGCTACCGG NGGAGTGAAGAAACCTCATCGTTACAGGCCCGGGACNGTCGCTCTCCGTGAGATCCGTCG CTACCAGAAGAGCACCGAGCTCCTGATCCGAAAANTGCCCTTCCAGCGTCTGGTCAGAGA AATCGCYCAGGACTTCAAGACCGAGCTGCGNTTCCAGAGNTCCGCCNTCATGGCNCTCCA GGAAGCNAGCGAAGCCTACCTCGTCGGTCTCTTCGAGGACACCAACCTNTGCNccatyca cgccaaacgtgtcacnatnatgccnaaggatatgtcatagctgtttcn
Задание 2. Пример нечитаемого фрагмента хроматограммы
Имя файла: WS2980_H3_F_A03_2013-06-11-22-09-18.ab1
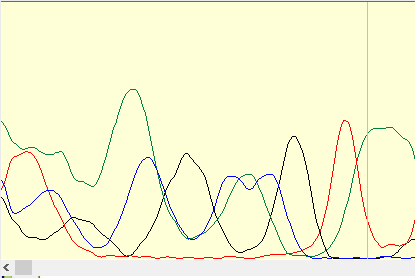
В качестве примера приведу участок хроматограммы прямой последовательности
(имя файла указано выше). Пики на данном участке широкие (возможно также, что произошло слияние нескольких
пиков в один), неоформленные, без возможности адекватной идентификации; есть возможность перепутать их с размытием краски.