Банки нуклеотидных последовательностей
Задание 1. Качество сборки генома эукариотического организма
Название: Пещерный крылан (Eonycteris spelaea)
Число сборок генома: 1
Основная информация по сборке Espe.v1:
| Общая длина (последовательность) | 1 966 861 576 |
| Число контигов сборки | 4 469 |
| N50 и L50 | 8 002 591 и 70 соответствено |
| Число аннотированных белков | 117 |
| Публикация с описанием проекта | PRJNA427241 |
| Последовательность контига PUFA01000002.1 | ссылка |

Краткая характеристика организма: пещерная нектарная летучая мышь; научное название вида
впервые опубликовано Добсоном в 1871 году.
Голова и тело имеют характерную длину 8.5 - 11 см, хвост 1.5 - 1.8 см
Встречается чаще в лесах, но может жить и на сельскохозяйственных угодьях
Задание 2. Описание семи ключей, используемых в таблицах особенностей
| Название | Описание | Пример |
| CDS | Кодирующая последовательность; последовательность нуклеотидов, которой соответствует последовательность аминокислот в белке ; содержит информацию о продукте гена, функции; названии, координатах гена и др.
/allele="text"
/artificial_location="[artificial_location_value]"
/citation=[number]
/codon_start=<1 or 2 or 3>
/db_xref="
|
Пример из раздела help
CDS 212..8668
/gene="NF1"
/note="putative"
/codon_start=1
/product="GAP-related protein"
/protein_id="AAA59924.1"
/translation="MAAHRPVEWVQAVVSRFDEQLPIKTGQQNTHTKVSTE..
|
| C_region | Константный участок легкой и тяжелой цепей иммуноглобулинов, а также альфа, бета и гамма цепей Т-клеточного рецептора; включает 1 и более экзонов в зависимости от каждой конкретной цепи Optional qualifiers /allele="text"
/citation=[number]
/db_xref="
|
Ссылка на запись C_region join(14002..14280,14849..14914,15573..15685) /standard_name="TCR delta chain constant region" |
| mobile_element | Участок генома, содержащий в себе мобильный элемент
Optional qualifiers /allele="text"
/citation=[number]
/db_xref="
|
Ссылка на запись mobile_element 1179..4322 /note="YCLWTy5-1; Ty5 element, LTR retrotransposon of the Copia (Pseudoviridae) group; this is the only near-full length Ty5 retrotransposon in S288C and it is non-functional" /mobile_element_type="retrotransposon:YCLWTy5-1" /db_xref="SGD:S000006831" |
| exon |
Участок генома, кодирующий сплайсирующийся участок mRNA, rRNA или tRNA;
может содержать 5' некодирующие области, белок кодирующие последовательности,
а также 3' некодирующие области
Optional qualifiers /allele="text"
/citation=[number]
/db_xref="
|
Ссылка на запись exon 97686..97955 /product="TCR alpha chain constant region" /note="putative" /citation=[1] /number=1 |
| rep_origin |
Начало репликации; стартовый участок дупликации нуклеиновой кислоты
Optional Qualifiers /allele="text"
/citation=[number]
/db_xref="
|
Ссылка на запись rep_origin 14575..14849 /note="ARS302; Inactive replication origin linked to the silent mating type locus HML; functions as a transcriptional silencer" /db_xref="SGD:S000028478" |
| J_segment |
Линкерный сегмент легкой и тяжелой цепей иммуноглобулинов, а также альфа, бета и гамма цепей Т-клеточного рецептора
Optional qualifiers /allele="text"
/citation=[number]
/db_xref="
|
Ссылка на запись J_segment 51609..51665 /standard_name="possible TCRAJ 40 gene segment" |
| misc_RNA | Какой-либо транскрипт или РНК-продукт, который не может быть определен другими РНК-ключами (prim_transcript, precursor_RNA, mRNA, 5'UTR, 3'UTR, exon, CDS, sig_peptide, transit_peptide, mat_peptide, intron, polyA_site, ncRNA, rRNA and tRNA)
Optional qualifiers /allele="text"
/citation=[number]
/db_xref="
|
Ссылка на страницу с записью misc_RNA complement(476684..476779) /product="purine riboswitch" |
Задание 3. Геномный проект Genome 10K
Цель: cеквенирование 10 000 геномов позвоночных, что создаст основу для разнообразных исследований

Сообщество ученых Genome 10K было основано Дэвидом Хаусслером, Оливером Райдером
и Стивеном О'Брайеном в апреле 2009 года на встрече в Калифорнийском университете (Санта-Крус)

В числе первых участников встречи были 55 ведущих ученых, представляющих крупные зоопарки, музеи,
исследовательские центры и университеты по всему миру. Genome 10K вырос, и по состоянию на 2017 год включает
проект генома позвоночных (Vertebrate Genomes Project, VGP)
Миссия G10K-VGP заключается в создании высококачественных геномов всех видов позвоночных животных, с минимумом ошибок
Cсылка на страницу: здесь

Oрганизация Сообщество ученых G10K (G10kCOS) представляет более 150 членов в более чем 50 учреждениях
Инфраструктура консорциума охватывает целый ряд задач: от сбора проб до секвенирования генома,
его сборки, аннотации, выравнивания, публикации общедоступных данных
G10KCOS представляет следующие таксономические группы:
- Млекопитающие
- Птицы
- Рыбы
- Рептилии
- Приматы
- Земноводные
- Беспозвоночные / Другие
Направления и виды деятельности в рамках проекта:
- Сборка геномов
- Выравнивание геномов, аннотация
- Филогенетический анализ
- Сравнительная геномика
- Сектор компьютерных вычислений (информатика)
- Сбор средств
- Интернет-деятельность
- Организация ежегодных совещаний
Планируемое число геномов: 10 000
Год завершения: 2015
Состояние на 2017 год: проект G10K успешно справился с поставленной задачей (10 000 геномов позвоночных);
далее было запланировано расширить проект до VGP (66 000 геномов)
Кстати, по состоянию на 13 сентября 2018 VGP "выпущено" 15 высококачественных референсных геномов

Последняя публикация по проекту (PubMed): ссылка
Задание 4. Таблица митохондриальных генов
Текст запроса при поиске:
tax_tree(32443) AND mol_type="genomic DNA" AND topology="CIRCULAR" AND organelle="mitochondrion"
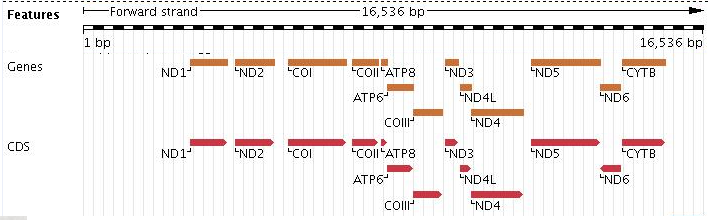
Sequence: KF703755.1
Общая длина последовательности: 16 536
Был произведен поиск по ENA на сайте EBI,
в результате чего было получено 24 находки в Update и 4 372 находки в Release
В качестве представителя таксона был выбран организм Hippocampus kelloggi (great seahorse), иначе говоря, -
Великий морской конь, также известный как Морской конь Келлог

Таблицу митохондриальных генов морского конька вы можете загрузить, кликнув по ссылке
На рисунке ниже представлено расположение соответствующих кодирующих участков в геноме; в рассматриваемой записи было представлено 13 белковых продуктов