Задание 1. База данных OPM
Выбранный белок (с альфа-спиралями в трансмембранном пространстве):
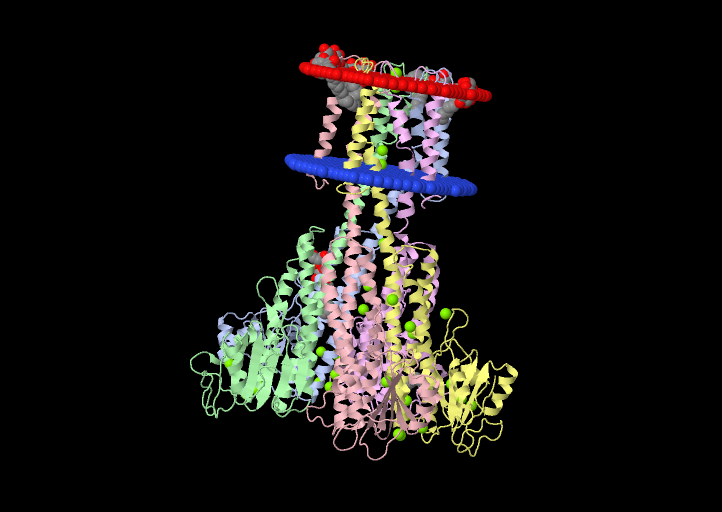
Magnesium transport protein CorA
PDB
Таблица 1. 4EV6
| Толщина гидрофобной части белка в мембране | 28.4 ±- 1.5 A |
| Координаты трансмембранных спиралей |
Subunits: 5 |
| Среднее количество остатков в одной трансмембранной спирали | 15.5 |
| Положение | Трансмембранный |
Выбранный белок (с бета-слоями в трансмембранном пространстве):
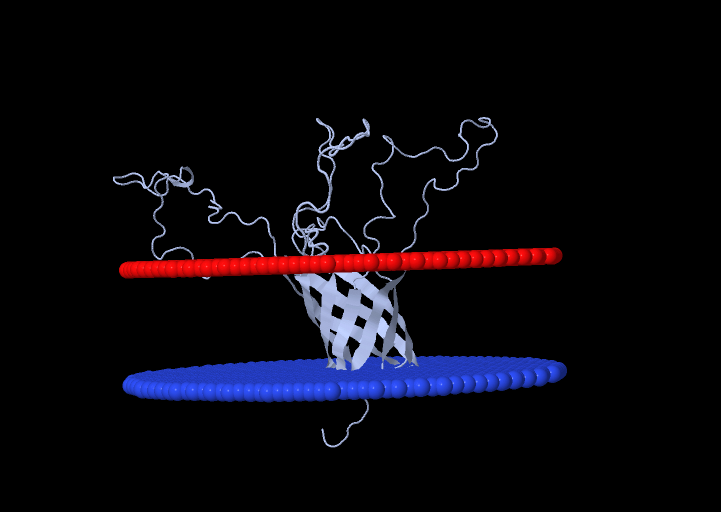
Opacity protein opA60, structure 2
PDB
Таблица 2. 2MLH
| Толщина гидрофобной части белка в мембране | 23.2 ± 2.7 A |
| Координаты трансмембранных спиралей |
1( 9- 16), 2( 53- 62), 3( 65- 73), 4( 116- 125) 5( 131- 142), 6( 191- 201), 7( 206-214), 8( 232- 236) |
| Среднее количество остатков в одной трансмембранной спирали | 6.25 |
| Положение | Трансмембранный бета-бочонок |
Задание 2. Анализ предсказания трансмембранных спиралей
Сервис TMHMM
Прежде всего, посмотрим на особенности предсказания трансмембранных спиралей,полученных на основе сервиса TMHMM
На вход диалоговое окно запрашивает последовательность в .fasta формате
В нашем FASTA-файле записана информация о 5 субъединицах нашего белка, в связи
с чем ожидается получения 5 вариантов предсказания (5 картинок, для каждой субъединицы)
Результаты приведены ниже
# 4EV6:A|PDBID|CHAIN|SEQUENCE Length: 339 # 4EV6:A|PDBID|CHAIN|SEQUENCE Number of predicted TMHs: 2 # 4EV6:A|PDBID|CHAIN|SEQUENCE Exp number of AAs in TMHs: 42.53828 # 4EV6:A|PDBID|CHAIN|SEQUENCE Exp number, first 60 AAs: 0.19337 # 4EV6:A|PDBID|CHAIN|SEQUENCE Total prob of N-in: 0.91337 4EV6:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 inside 1 280 4EV6:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 281 303 4EV6:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 outside 304 312 4EV6:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 TMhelix 313 333 4EV6:A|PDBID|CHAIN|SEQUENCE TMHMM2.0 inside 334 339
Первая строчка несет информацию об имени и длине последовательности, в нашем случае -
А-субъединицы изучаемой структуры 4EV6
Вторая строчка сообщает, по видиому, о количестве предсказанных
трансмембранных альфа-спиралей (TMH - TransMembrane Helices); их у нас две
Третья строчка говорит о предполагаемом количестве аминокислотных остатков, входящих
в состав альфа-спирали
Четвертая строчка - ожидаемое количество аминокислотных остатков,
входящих в состав трансмембранной альфа-спирали, из первых 60 аминокислот
В нашем случае это число небольшое, поэтому, вероятно, N-конец не является
лидерным пептидом или какой-либо сигнальной последовательностью
Пятая строчка содержит инфрмацию о полной вероятности того, что N-конец белка
расположен на внутренней стороне мембраны
Последующие несколько строк сообщают а номерах аминокислотных остатков, располагающихся
с наружной/внутренней стороны мембраны или непосредственно внутри
Посмотрим на предсказанные "границы" TMHs: [281 - 303] и [313 - 333];
Среднее число остатков, приходящихся на спираль: 21 (что больше числа, указанного в базе данных) +
каждая граничная точка сдвигается в среднем примерно
на 20 значений вправо (в сторону больших чисел)
Вероятно, это неточность в работе алгоритма предсказывания
Теперь рассмотрим графическое изображение
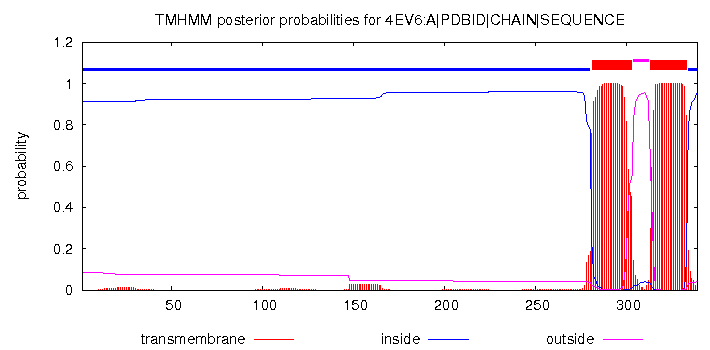
По оси у отложена вероятность того или иного положения участка белковой молекулы
По оси х расположены аминокислотные остатки
Красным цветом показана трансмембранная локализация
Розовымобозначена наружняя локализация
Синим - внутренняя
Над графиком показаны лучшие предсказания
Вероятно, график отличается (по предсказанной локализации) от структуры в базе данных
в связи с тем, что он строит предсказания, исходя из вероятности нахождения где-либо
для каждого отдельного аминокислотного остатка; референсная структура - наиболее выгодня в целом
(как я понимаю, без частностей, если речь не идет о каких-то участках связывания и т п
Сервис TMHMM предсказал все TMHs, для всех пяти доменом, с небольшими отличиями от референса
Сервис Phobius
Выдача выглядит следующим образом (как и в случае с TMHMM, на вход подаем последовательнось в .fasta - формате):ID 4EV6:A|PDBID|CHAIN|SEQUENCE FT TOPO_DOM 1 280 CYTOPLASMIC. FT TRANSMEM 281 301 FT TOPO_DOM 302 312 NON CYTOPLASMIC. FT TRANSMEM 313 333 FT TOPO_DOM 334 339 CYTOPLASMIC. //Сразу хочется заметить, что по содержательной части она менее подробная, если сравнивать с TMHMM
Первая строчка нечет информацию об имени (идентификаторе) последовательности, в нашем случае - А-субъединицы
Вторая строка содержит информацию о границах цитоплазматического участка белковой последовательности
Забавно, что "внемембранные" стороныназываются "цитоплазматическая" и "не цитоплазматическая"
Также в выдаче программы есть информация об интересующих нас "TRANSMEM" участках;
Границы: [281 - 301] и [313-339]
Нижние границы совпадают с выдачей рассматриваемого ранее сервиса, а одна верхняя - отличается, причем в + и - сторону
Среднее количество аминокислотных остатков на спираль: 20; это чуть больше похоже на референс, но отличие, как мне
кажется, малозначимое
Посмотрим на графическую выдачу Phobius
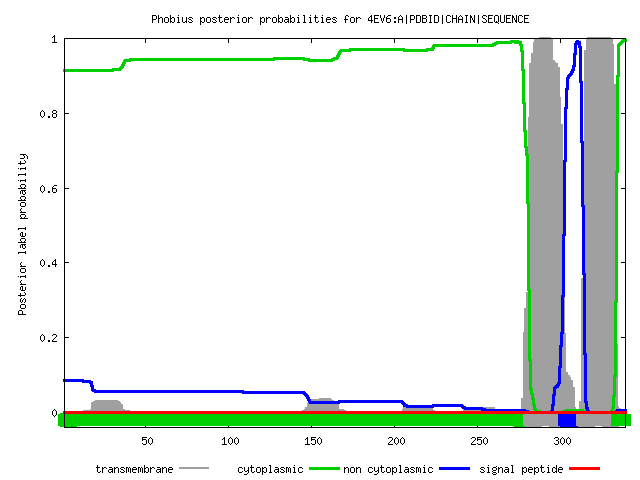
По оси у отложена постериорная вероятность положения участка белка
По оси х отложены аминокислотные остатки
Cерым обозначена трансмембранная локализация
Зеленым - цитоплазматическая
Синим - "не цитоплазматическая"
Красным - сигнальный пептид
Интересно, что в TMHMM он не был обозначен на графике, но информацию о наличии сигнального
пептида можно было получить из текстовой выдачи сервиса
Сервис Phobius предсказал все TMHs, для всех пяти доменом, с небольшими отличиями от референса
Задание 3. База данных TCDB
Белок 4EV6TCID: 1.A.35.3.1
Посмотрим, что означает запись TCID:
1.A: белки этой категории образуют каналы (поры)
и состоят преимущественно из альфа-спиралей, с возможными участками бета-слоев; конкретно наш белок относится к классу каналы альфа-типа
Трансмембранные белки-каналы, принадлежащие этому классу повсеместно обнаружены в мембране организмов всех типов - от бактерий до
эукариот. Обычно это белки катализируют перемещение движение веществ (в растворе) посредством энергонезависимого процесса транспорта
Как было сказано ранее, это белки состоят преимущественно из протяженных участков альфа-спиралей, тем не менее, бета-тяжи также могут
присутствовать в структуре и даже вносить вклад в образование канала
Канальные белки (наружной мембраны) поринового типа исключены из этого класса и вместо этого входят в состав класса 1.B
1.A.35 Семейство MIT (The CorA Metal Ion Transporter)
Семейство MIT, также названное CorA, - это большая и разнообразная группа, "размещенная" в
Грам+ и Грам- бактериях, сине-зеленых водорослях, археях, растенях, животных, дрожжах и др.
Большинство белков этой группы имеют длину около 300 - 400 аминокислотных остатков и предположительно содержат
2 или 3 трансмембранные альфа-спирали
Если посмотреть на наш белок, то можно увидеть, что в нем 10 трансмембранных спиралей, но, тем не менее, он принадлежит
этому cемейству. Думаю, дело в том, что в рассматриваемой структуре 5 субъединиц, каждая из которых содержит по две трансмембранных
cпирали - тогда отнесение белка к семейству MIT можно назвать верным
1.A.35.3.1
Cогласно информации с TCID, данная категория говорит о том, что изучаемый белок - транспортер двухвалентных
ионов металлов (Mg2+, Ca2+, Ni2+ и др.). Cryo-EM структура показывает пентамерный канал с асимметричной доменной структурой +
она предполагает связь цитоплазматического участка с трансмембранным доменом
GENE ONTOLOGY GO:0016021 C:integral to membrane GO:0005886 C:plasma membrane GO:0015087 F:cobalt ion transmembrane transporter activity GO:0015095 F:magnesium ion transmembrane transporter act... GO:0006824 P:cobalt ion transport GO:0015693 P:magnesium ion transport GO:0055085 P:transmembrane transport
Белок 2MLH
PDB STRUCTURES:
SORRY, there is no such ID