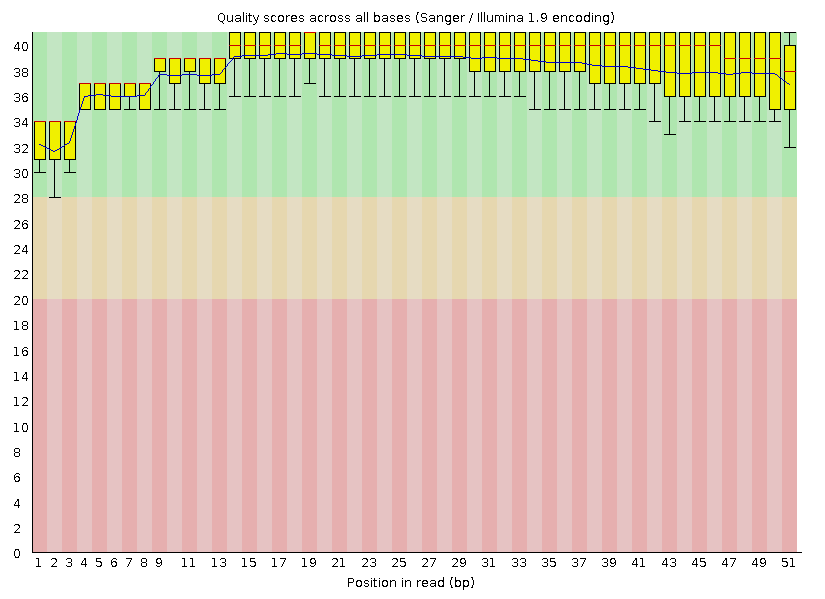
 Качество ридов. на картике заметро что качество ридов удовлетворительное (все спускается не ниже 28), значит
чистку можно не проводить
Качество ридов. на картике заметро что качество ридов удовлетворительное (все спускается не ниже 28), значит
чистку можно не проводить
fastqc chr9_1.fastq
команда выдает анализ качества чтений (2 файла: chr9_1_fastqc.html chr9_1_fastqc.zip)
hisat2-build -f chr9.fasta chr9
уже есть нужные файлы переложили в директорию /ht2/
hisat2 --no-softclip -x ../ht2/chr9 -U chr9.1.fastq -S kart_trans.sam
В случае с транскриптомом: мы смотрим только РНК, а там могут быть вырезаны разные участки: поэтому и риды будут ложиться с разрывами. Убираем --no-spliced-alignment
19976 (100.00%) were unpaired; of these:
114 (0.57%) aligned 0 times
19862 (99.43%) aligned exactly 1 time
0 (0.00%) aligned >1 times
99.43% overall alignment rate
Команда Hisat2 выдает общий процент покрытия ридами генома, количество ридов, которые легли ровно 1 раз, больше 1 раза, не легли вообще и количество тех, что легли.
отсюда видно что 19862 чтений легли на геном
samtools view -b kart_trans.sam -o kart_trans.bam
переаводит формат sam в bam
samtools sort -f kart_trans.bam out_trans.bam
сортируем по началу чтений, выходной файл bam (-f означает что задаем полностью имя выходного файла)
samtools index out_trans.bam
индексируем отсортированный bam (на выйходе формат файла out.bam.bai)
-f формат файла bam.sam
-s --stranded {yes,no,reverse} прямая или обратная цепь
-i --idattr (атрибут id по умолчанию suitable for Ensembl GTF files: gene_id)
-m --mode {union,intersection-strict,intersection-nonempty}режим для обработки ридов перекрывающихся больше 1 раза
htseq-count -f bam -s yes -i gene_id -m union out_trans.bam gencode.v19.chr_patch_hapl_scaff.annotation.gtf >> count
команда смотрит на то, какие гены и как покрываются ридами
Выдача програмы:
__no_feature 19847 __ambiguous 0 __too_low_aQual 0 __not_aligned 114 __alignment_not_unique 0
Не все чтения легли в греницы генов. (всего 15 чтений положено на ген). Некоторые чтения вообще не выровнялись, а большинство чтений (19847) не ложаться на гены. возможно они распределены по интронам. Единственный покрытый ген оказался: ENSG00000119335.12 который был покрыт 15 раз. У данного гена название: SET nuclear proto-oncogene. ген ответственный за синтез мультизадачного белка, который принимает участие в апоптозе, транскрипции, сборке нуклеосом и сопровождении (chaperoning) гистонов.