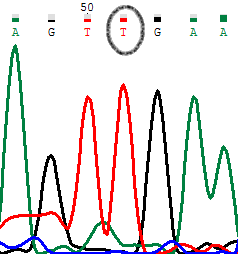
На обратной диаграмме видно, что сигнал от С и Т сравним. Но во первых, прямое секвенирование дает четкое представление, что сигнал от С - шум, тк сигнала от С вообще на прямой диаграмме не зафиксировано. Во вторых, если посмотреть на обратную диаграмму уровень шума стал сильно повышаться, а сигналы от соседних нуклеотидов тоже “забиваются” шумом сравнимой силы. Плюс ко всему сигнал от С сместился относительно “нормальных” сигналов- значит это шум

PIC1. Прямое секвенирование.
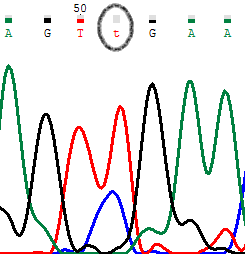
PIC2. Обратное секвенирование.
на прямой последовательности мы видим сравнимые сигналы от G и А (пик G выше). Отсутствие шума на соседних нуклеотидах дает нам основание полагать, что это полиморфизм (R). На обратной последовательности мы видим четкое доминирование сигнала от G над А.(заметим что здесь п рисутствует такой же сигнал от А) Если смотреть на обратное отдельно, то можно было бы подумать, что сигнал от А-это шум, но если обратить внимание что у соседних нуклеотидов шума совсем нет, то делаем выбор в сторону того что это полиморфизм.
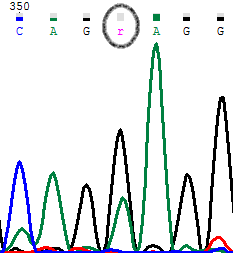
PIC1. Прямое секвенирование.
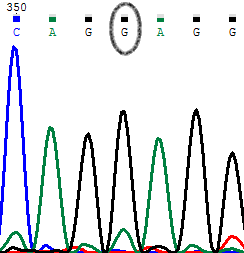
PIC2. Обратное секвенирование.
прямая диаграмма показывает сравнимые сигналы от А G(А выше). при условии того что соседние нуклеотиды имеют примерно такой же уровень шума что и 99, но определяются однозначно. Обратная диаграмма показывает, что на этом месте однозначно стоит А, а сигнал от последующего нуклеотида (G) просто перекрывается с сигналом от А. Таким образом, делаем вывод, что в прямой последовательности стоит А. А G это шум.
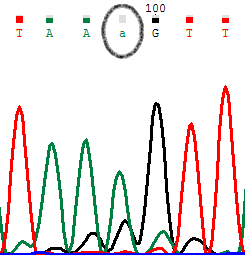
PIC1. Прямое секвенирование.
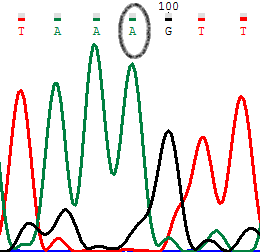
PIC2. Обратное секвенирование.
Здесь мы видим шум А с сигналом от Т. их пики сравнимы на прямой диаграмме но не сравнимы на обратной. Надо отметить, что уровень шума на обеих диаграммах-достаточно высок. Что может склонять нас к тому что это проблемный нуклеотид. С другой стороны и на прямом и на обратном секвенировании на этом месте зафиксированы сигналы от одних и тех же нуклеотидов (А и Т)- что может склонить нас к тому что это полиморфизм, но видно что на обратной диаграмме, уровень сигнала от А это явно шум. Поэтому делаем выбор в сторону проблемного нуклеотида и исправляем прямую последовательность.
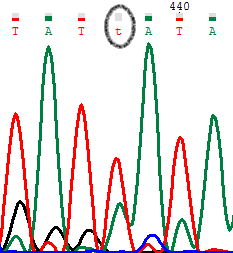
PIC1. Прямое секвенирование.
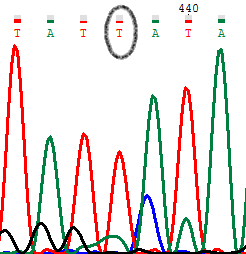
PIC2. Обратное секвенирование.
Шум(сигнал от С) сдвинут сильно от сигнала от Т вправо. Из того что расстояние между пиками должно быть одинаковым, делаем вывод что это шум и исправляем N->t. Согласно тому, что показывает прямое секвенирование.
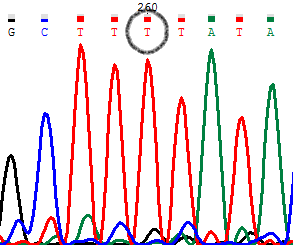
PIC1. Прямое секвенирование.
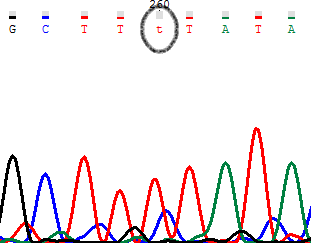
PIC2. Обратное секвенирование.
Прямое: начало 0, конец 28. Обратное: начало 33, конец 15.
1:10
на прямом: шум не поднимается больше чем на ? от сигнала. Такие шумы встречаются примерно 3 раза на первую треть последовательности, в середине 1 нуклеотид с шумом более чем на ? от интенсивности сигнала встречается примено на 60 нуклеотидов, под конец такие шумы учащаются до 1:40
на обратном: поднятие уровня шума больше чем на ? от сигнала прослеживается довольно часто, если смотреть от начала, то можно заметить что встречаемость такого шума в начале и в конце последовательности частая, а в середине редкая.
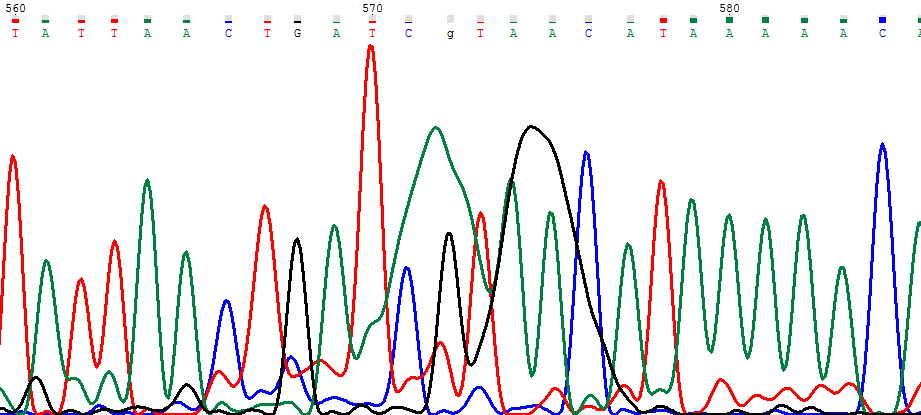
PIC3. Обратное секвенирование.
На 123-137 мы видим высокие пики сигналов от шума. Это явно мусор, тк он испускает все 4 возможных сигнала на протяжении целых 14 нуклеотидов. На прямой последовательности нуклеотиды определяются однозначно, а сигналы от других возможных вариантов испускаются на уровне шума. Это все значит что исправляем обратную последовательность на данные из прямой.
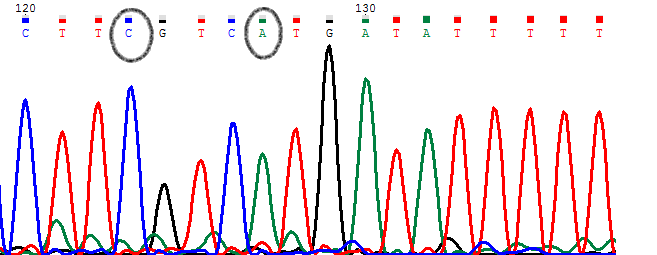
PIC1. Прямое секвенирование.
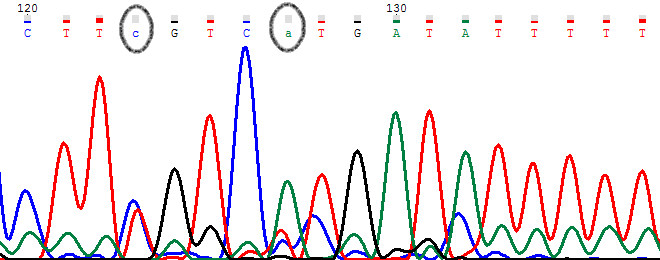
PIC2. Обратное секвенирование.