prot.fasta там лежат протеомы выбранных бактерий #предварительно из prot.fasta нужно сделать файл с "базой данных" makeblastdb -in proteoms.fasta -dbtype prot blastp -db proteoms.fasta -query CLPX_ECOLI.fasta -evalue 0.001 -out blastp -outfmt 6 #в файле blastp теперь таблица о ближайших гомологах, к сожалению на каком-то этапе возникли трудности, потому что в prot.fasta несколько раз сложены одни и те же последовательности, пришлось питоном оставлять уникальные (unic_fasta.py) #Стряпаем id.txt с id белков в форме prot.fasta:XXXX_YYYY из выданной таблицы seqret list::id.txt >> clpx_bac2.fasta #вырезаем последовательности #Строим выравнивание muscle -in clpx_bac2.fasta -out clpx_bac2_muscle.fasta #Удаляем ненужную часть названия cat clpx_bac2_muscle.fasta | cut -d' ' -f1 >> clpx_bac_tree.fasta #Потом в программе MEGA надо отформатировать в .meg, затем построить выравнивание, затем построить филогению (Data Philogenetic Analises), затем строится дерево "UPGMA tree".
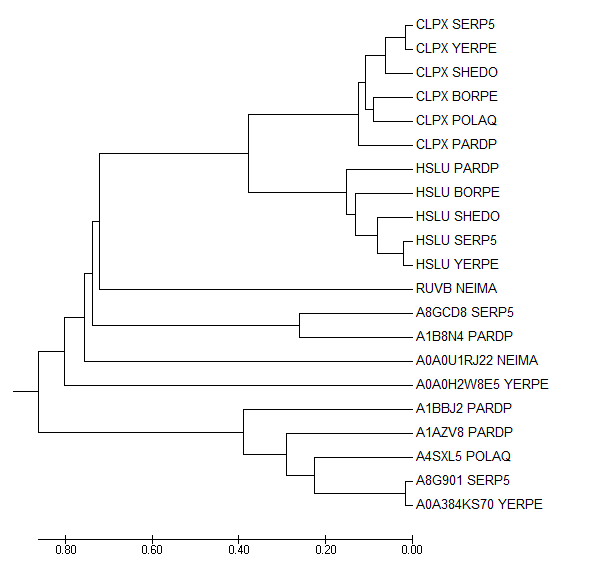
Несколько пар паралогов:(CLPX_PARDP HSLU_PARDP),(CLPX_SERP5 HSLU_SERP5) Три группы попарно ортологичных белков:(CLPX_SERP5 CLPX_YEPRE)(HSLU_PARDP HSLU_YEPRE),(A8G901_SERP5 A0A384KS70_YEPRE) Примеры отражённых на дереве эволюционных событий: 1) дупликация гена (CLPX HSLU _SEPR5),(CLPX HSLU _SHEDO) 2) разделение путей эволюции белков в результате видообразования (A8GCD8_SERP5 A1B8N4_PARDP), (A8G901_SERP5 A0A384KS70_YEPRE).
E-mail: Задавайте вопросы по электронной почте