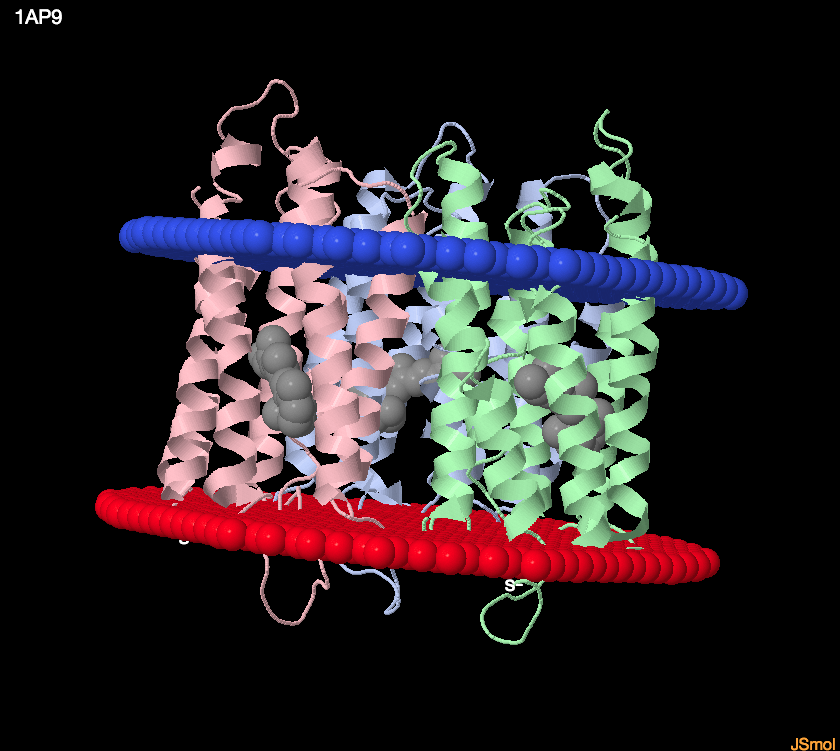
 Изображено множество альфа-спиралей пронизывающих мембрану. Красная сторона- наружняя сторона мембраны.
Изображено множество альфа-спиралей пронизывающих мембрану. Красная сторона- наружняя сторона мембраны.| Название | Bacteriorhodopsin, from cubic phase | ||||||
| ID | 1ap9 | ||||||
| Hydrophobic Thickness or Depth | 30.6 ± 1.2 ? | ||||||
| subunits (3) | |||||||
| A - Tilt: 7 - TM segments: | 1( 9- 29) | 2( 44- 62) | 3( 80- 97) | 4( 108- 126) | 5( 134- 153) | 6( 174- 191) | 7( 201- 222) |
| C - Tilt: 7 - TM segments: | 1( 9- 29) | 2( 44- 62) | 3( 80- 97) | 4( 108- 126) | 5( 134- 153) | 6( 174- 191) | 7( 201- 222) |
| B - Tilt: 7 - TM segments: | 1( 9- 29) | 2( 44- 62) | 3( 80- 97) | 4( 108- 126) | 5( 134- 153) | 6( 174- 191) | 7( 201- 222) |
| Среднее количество остатков в одной трансмембранной спирали (или одном ?-тяже) белка. | 18,6 | ||||||
| Место нахождения | внешняя мембрана Archaebacterial |
 Изображено множество альфа-спиралей пронизывающих мембрану. Красная сторона- наружняя сторона мембраны.
Изображено множество альфа-спиралей пронизывающих мембрану. Красная сторона- наружняя сторона мембраны.
| Название | Major outer membrane protein TtoA | |||||||
| ID | 3dzm | |||||||
| Толщину гидрофобной части белка в мембране: | 28.5 ± 2.6 ? | |||||||
| Координаты трансмембранных спиралей ( | ||||||||
| A - Tilt: 2 - TM segments: | 1(4-12) | 2(16-25) | 3(33-42) | 4(74-83) | 5(93-104) | 6(128-139) | 7(144-155) | 8(195-206) |
| Среднее количество остатков в одной трансмембранной спирали (или одном ?-тяже) белка. | 9,875 | |||||||
| В какой мембране находится белок | Bacterial Gram-negative outer membrane |
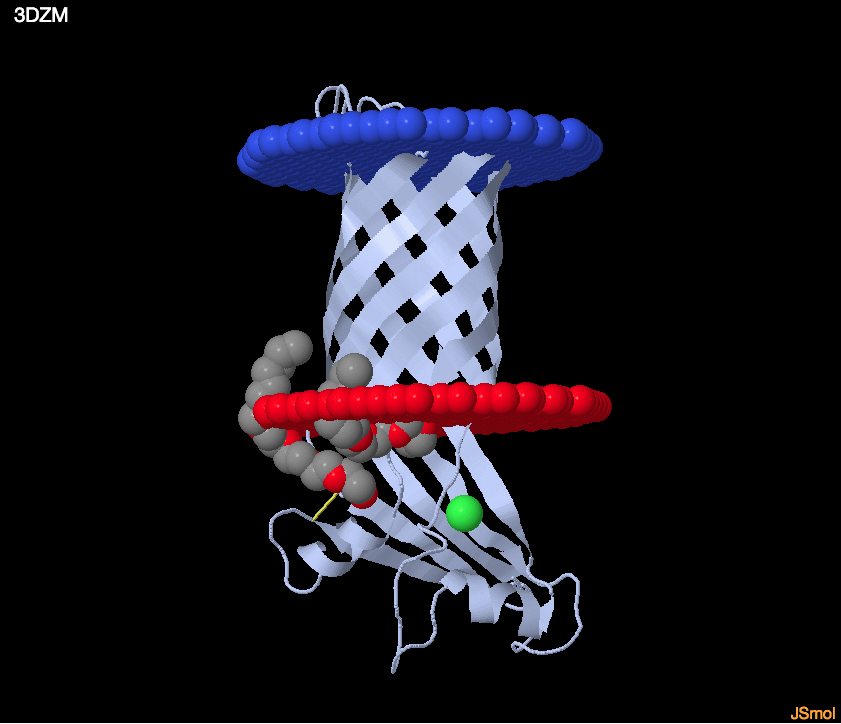 Изображен трансмембранный бета лист образующий пору (бочка). Внешняя среда находится снизу.
Изображен трансмембранный бета лист образующий пору (бочка). Внешняя среда находится снизу.
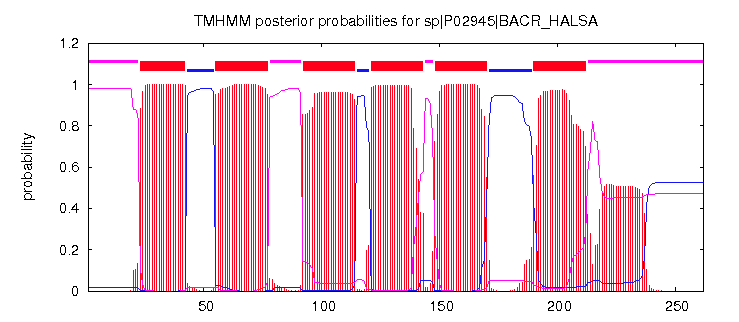
Таблица выведенная TMHMM
Трансмембранные последовательности на графике отмечены красным, цепочки оказавшиеся на n стороне помечены синим, а p сторона розовым. В самом верху результирующая цепочка (ввиде ленты). Данный сервис указал количество трансмембранных фрагментов 6. На самом деле OPM их 7. Таким образом есть незамеченная спираль и Координаты спиралей сдвинуты.
| # sp|P02945|BACR_HALSA | Length: 262 | |||
| # sp|P02945|BACR_HALSA | Number of predicted TMHs: 6 | |||
| # sp|P02945|BACR_HALSA | Exp number of AAs in TMHs: 140.4032 | |||
| # sp|P02945|BACR_HALSA | Exp number, first 60 AAs: 26.1196 | |||
| # sp|P02945|BACR_HALSA | Total prob of N-in: 0.01887 | |||
| # sp|P02945|BACR_HALSA | POSSIBLE N-term signal sequence | |||
| sp|P02945|BACR_HALSA | TMHMM2.0 outside | 1 | 22 | |
| sp|P02945|BACR_HALSA | TMHMM2.0 TMhelix | 23 | 42 | |
| sp|P02945|BACR_HALSA | TMHMM2.0 inside | 43 | 54 | |
| sp|P02945|BACR_HALSA | TMHMM2.0 TMhelix | 55 | 77 | |
| sp|P02945|BACR_HALSA | TMHMM2.0 outside | 78 | 91 | |
| sp|P02945|BACR_HALSA | TMHMM2.0 TMhelix | 92 | 114 | |
| sp|P02945|BACR_HALSA | TMHMM2.0 inside | 115 | 120 | |
| sp|P02945|BACR_HALSA | TMHMM2.0 TMhelix | 121 | 143 | |
| sp|P02945|BACR_HALSA | TMHMM2.0 outside | 144 | 147 | |
| sp|P02945|BACR_HALSA | TMHMM2.0 TMhelix | 148 | 170 | |
| sp|P02945|BACR_HALSA | TMHMM2.0 inside | 171 | 189 | |
| sp|P02945|BACR_HALSA | TMHMM2.0 TMhelix | 190 | 212 | |
| sp|P02945|BACR_HALSA | TMHMM2.0 outside | 213 | 262 |
Трансмембранные последовательности на графике отмечены серым; цитоплазменные (n сторона) отмечены зеленым; внешние (p) сторона отмечены синим
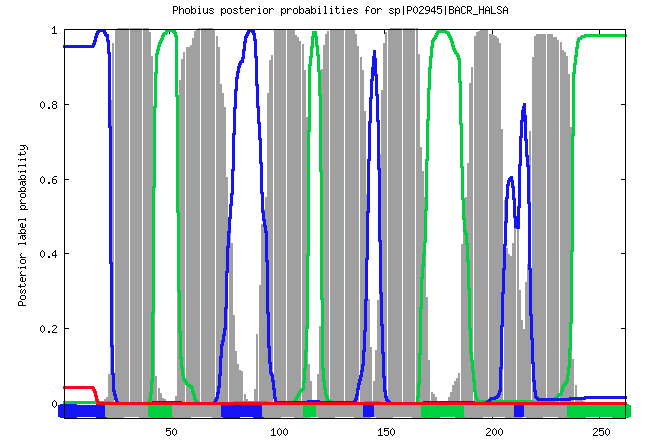
Данный алгоритм распознает еще одну нужную спираль! Но при этом координаты все равно определяются неправильно, так же как в TMHMM.
Таблица выведенная Phobius
| ID | sp|P02945|BACR_HALSA | |
| FT | TOPO_DOM 1 22 | NON CYTOPLASMIC. |
| FT | TRANSMEM 23 42 | |
| FT | TOPO_DOM 43 53 | CYTOPLASMIC. |
| FT | TRANSMEM 54 76 | |
| FT | TOPO_DOM 77 95 | NON CYTOPLASMIC. |
| FT | TRANSMEM 96 114 | |
| FT | TOPO_DOM 115 120 | CYTOPLASMIC. |
| FT | TRANSMEM 121 142 | |
| FT | TOPO_DOM 143 147 | NON CYTOPLASMIC. |
| FT | TRANSMEM 148 169 | |
| FT | TOPO_DOM 170 189 | CYTOPLASMIC. |
| FT | TRANSMEM 190 212 | |
| FT | TOPO_DOM 213 217 | NON CYTOPLASMIC. |
| FT | TRANSMEM 218 237 | |
| FT | TOPO_DOM 238 262 | CYTOPLASMIC. |
Альфа
TC:3.E.1.1.1 (Bacteriorhodopsin 1ap9) (Значение 3: Primary Active Transporters, 3.E: Light absorption-driven transporters, 3.E.1: The Ion-translocating Microbial Rhodopsin (MR) Family)
Бета
TC:1.B.6.8.1 (Porin, Hypothetical conserved protein 3dzm) (1:Channels/Pores, 1.B: ?-Barrel Porins, 1.B.6: The OmpA-OmpF Porin (OOP) Family)
Bacteriorhodopsin. Proton efflux occurs via a transient linear water-molecule chain in a hydrophobic section of the Brho channel between Asp96 and Asp85 (Freier et al., 2011). It can be converted to a chloride uptake pump by a single amino acid substitution at position 85. However, halorhodopsin (3.E.1.2.1), which pumps chloride ions (Cl-) into the cell, apparently does not use hydrogen-bonded water molecules for Cl- transport (Muroda et al. 2012). Nango et al. 2016 used time-resolved serial femtosecond crystallography and an x-ray free electron laser to visualize conformational changes in bRho from nanoseconds to milliseconds following photoactivation. An initially twisted retinal chromophore displaces a conserved tryptophan residue of transmembrane helix F on the cytoplasmic side of the protein while dislodging a key water molecule on the extracellular side. The resulting cascade of structural changes throughout the protein shows how motions are choreographed as bRho transports protons uphill against a transmembrane concentration gradient. Nango et al. 2016 have created a 3-d movie of structural changes in the protein showing that an initially twisted retinal chromophore displaces a conserved tryptophan residue of transmembrane helix F on the cytoplasmic side of the protein while dislodging a key water molecule on the extracellular side. Brho has light-independent lipid scramblase activity (Verch?re et al. 2017). This activity occurs at a rate >10,000 per trimer per second, comparable to that of other scramblases including bovine rhodopsin and fungal TMEM16 proteins. BR scrambles fluorescent analogues of common phospholipids but does not transport a glycosylated diphosphate isoprenoid lipid. In silico analyses suggested that membrane-exposed polar residues in transmembrane helices 1 and 2 of BR may provide the molecular basis for lipid translocation by coordinating the polar head-groups of transiting phospholipids. Consistent with this possibility, extensive coarse-grained molecular dynamics simulations of a BR trimer in a phospholipid membrane revealed water penetration along transmembrane helix 1 with the cooperation of a polar residue (Y147 in transmembrane helix 5) in the adjacent protomer. These findings suggest that the lipid translocation pathway may lie at or near the interface of the protomers of the BR trimer (Verch?re et al. 2017). Retinal isomerization has been observed in the using a femtosecond x-ray laser (Nogly et al. 2018).
Porin of 224 aas and 8 beta strands, TtoA (Estrada Mallarino et al. 2015). The crystal structure is known (3DZM) (Nesper et al. 2008). The 2.8 ? structure reveals a transmembrane 8 stranded ?-barrel, an extracellular cation-binding region and an external 5-? stranded sheet (Brosig et al. 2009).
E-mail: Задавайте вопросы по электронной почте