|
Практикум№1(2 блок) A-, В- и Z- формы ДНК. Структура РНКС помощью программы fiber пакета 3DNA были постройнны A-, B- и Z-формы дуплекса ДНК. Последовательность одной из нитей представляет собой 5 раз повторенную последовательность "gatc".
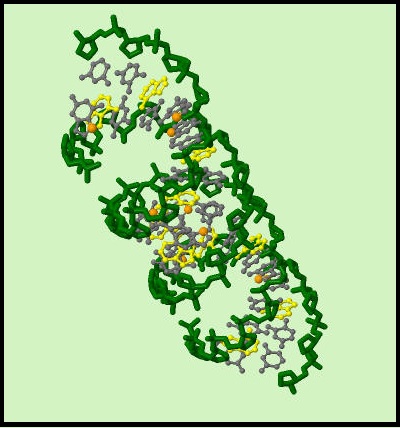
Рис1. Трехмерная структура A-формы ДНК. С помощью программы Jmol было получено рис1. Мы выделили цветом или способом отображения •сахарофосфатный остов ДНК-зеленый цвет •все нуклеотиды-серый цвет •все аденины-желтый цвет •атом N7 во всех гуанинах - оранжевый цвет. Для выполнения следующего задания мне нужно было скачать с сайта PDB структуры с идентификаторами 1il2 и 1odh, включающие комплекс балка и транспортной РНК и ДНК-белковый комплекс соответственно. Мы проверили заданные структуры ДНК и РНК на наличие разрывов, их не обнаружили.
Сравнение структур 3-х форм ДНК с помощью средств JMolС помощью программы Jmol сравниваются параметры структур А-, В- и Z- форм ДНК. Результаты представлены в таблицы1. Таблица1. Сравнивнение параметров структур А-, В- и Z- форм ДНК
Далее ну)\(но было сравнить торсионные углы в структурах А- и В-форм. Таблица2. Торсионные углы А-, В- форм ДНК
Я выберала заданное нам азотистое основание в любом месте структуры и определила, какие атомы основания явно обращены в сторону большой бороздки, а какие - в сторону малой. (Рисунок ни)\(е)
С поощью программ find_pair и analyze мы определили торсионные углы в заданной структуре ДНК. Самое "деформированное" основание, на первый взгляд, второй аденин.
Strand I
base alpha beta gamma delta epsilon zeta chi
1 C --- --- -60.1 147.1 162.3 -85.8 -106.3
2 G 158.4 -158.8 169.0 139.5 -155.5 -144.4 -122.7
3 A -40.4 165.2 37.1 147.1 -169.0 -110.8 -105.0
4 T 35.9 179.0 -61.3 151.8 -171.0 -127.1 -96.7
5 G 29.4 167.9 -46.6 155.1 165.4 -90.4 -103.1
6 C -50.7 -164.5 36.4 136.5 -156.4 -106.8 -110.5
7 G -60.0 163.7 40.5 148.1 -112.2 166.2 -96.3
8 G -59.5 142.0 37.5 135.6 -168.9 -87.9 -128.8
9 G -68.7 179.9 38.0 132.4 171.7 -117.0 -113.9
10 T -34.6 -171.4 35.0 141.1 -168.0 -108.4 -117.1
11 G -54.8 175.0 38.6 136.7 -177.4 -124.5 -112.3
12 C 43.1 174.1 -51.2 151.9 167.1 -82.1 -109.1
13 A 170.0 147.3 172.8 149.0 --- --- -144.1
Strand II
base alpha beta gamma delta epsilon zeta chi
1 G -61.4 151.9 42.0 143.1 --- --- -99.6
2 C -34.4 156.8 31.1 147.0 -132.6 -162.6 -96.8
3 T -39.5 -179.1 43.3 148.0 -162.1 -117.3 -107.2
4 A -56.6 -167.3 37.9 139.0 173.7 -118.1 -112.2
5 C -58.5 -162.0 40.0 139.7 175.4 -92.6 -112.5
6 G -26.7 150.9 44.9 140.2 175.2 -105.8 -117.1
7 C -85.0 -142.0 45.0 142.4 -179.7 -137.0 -106.2
8 C -40.9 156.6 28.1 133.0 164.0 -81.1 -120.1
9 C -38.5 168.2 29.6 132.9 -148.8 -153.5 -105.3
10 A -47.6 165.4 39.6 137.0 -169.1 -105.0 -112.9
11 C -66.1 -162.8 38.2 137.2 -165.8 -124.4 -106.6
12 G 24.0 -172.7 -66.2 150.3 165.8 -74.4 -104.0
13 T --- --- 172.6 150.1 -154.2 -126.6 -98.5
Ср.зн. -25.99 30.19 26.57 141.93 -39.92 -100.23 -109.84
Для вторичной структуры заданной тРНК характерны следующие особенности: 1. Антикодоновый стебель: [938-944] - [932-926] 2. Акцепторный стебель: [901-907] - [972-966] 3. Т-стебель: [949-953] - [965-961] 4. D-стебель: [910-913] - [925-922] Ни)\(е представленны данные о взаимодействиях в РНК. Видны неканонические взаимодействия между нуклеотидами (обозначены звездочками). Цветом выделены отдельные цепи RMSD of the bases (----- for WC bp, + for isolated bp, x for helix change)
Strand I Strand II Helix
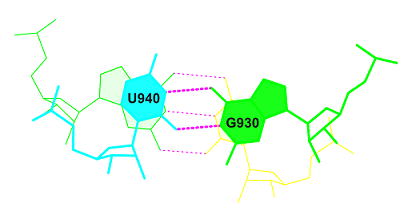
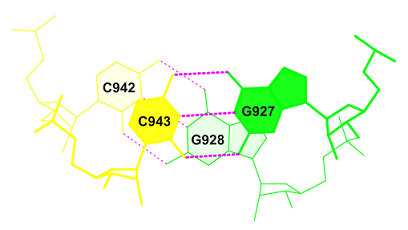
Из этой схемы видно, что нуклеотиды выделенные красным цветом образуют стебли, нуклеотиды водородные связи в которых способствуют поддержанию третичной структуры тРНК - должны связывать отдаленные участки тРНК - U1954 и А1958,Р1955 и G1917, A1914 и A1921, U1916 и U1959,G1918 и С1956. А неканонические пары - U1905 и G1968, P1955 и G1917,С1938 P1932 ,U1940 G1930 , A1944 G1926, P1913 G1922,U1908 A1946 , A1914 A1921, U1916 U1959. Bозможные стекинг-взаимодействия Площади перекрывания
16 GU/GC 7.38( 3.96) 0.00( 0.00) 0.00( 0.00) 7.11( 4.09) 14.49( 8.06)
19 CC/GG 0.05( 0.00) 0.00( 0.00) 0.55( 0.00) 1.95( 0.55) 2.55( 0.55)
Далее с помощью программы Jmol мы получили изобра)\(ения перекрываний. См. ни)\(е.
|