Для этого белка по данным Рfam найдено 3043 последовательностей домена, относящиеся к 805 видам.
Существует всего 62 архетиктур этого белка.
Пектат-лиаза (ЕС 4.2.2.2) является ферментом, принимающим участие в гниения растительной ткани.
Пектата лиазы катализирует расщепление пектата до олигосахаридов.
Далее было скаченно полное выравнивание всех последовательностей домена с сайта PFAM. Проект в Jalview
Следующим нашим шагом была получена таблица, содержащая информацию о доменной структуре каждой последовательности
и ее таксономии. Данные были получины с помощью предоставленных скриптов. Результат
,
разделенное на две группы, по архитектурам. Данное выравнивание уже обрезано для построение дерева
Далее на основе этого выравнивания было получено деверо с помощью программы Mega(был использован метод Test Maxcium Liklihood).
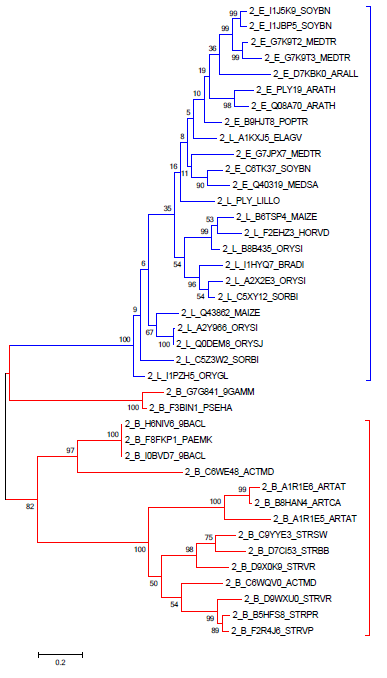
Рис1. Изображение дерева. Программа Mega. Красным обозначен домен 1 - Pec_lyase_C, CBM_1; Синим - 2 - Pec_lyase_N, Pec_lyase_C
Скобочная форма
((((((((((((((2_E_I1J5K9_SOYBN,2_E_I1JBP5_SOYBN),(2_E_G7K9T2_MEDTR,2_E_G7K9T3_MEDTR)),2_E_D7KBK0_ARALL),(2_E_PLY19_ARATH,2_E_Q08A70_ARATH)),2_E_B9HJT8_POPTR),2_L_A1KXJ5_ELAGV),(2_E_G7JPX7_MEDTR,(2_E_C6TK37_SOYBN,2_E_Q40319_MEDSA))),2_L_PLY_LILLO),(((2_L_B6TSP4_MAIZE,2_L_F2EHZ3_HORVD),2_L_B8B435_ORYSI),(2_L_I1HYQ7_BRADI,(2_L_A2X2E3_ORYSI,2_L_C5XY12_SORBI)))),(2_L_Q43862_MAIZE,(2_L_A2Y966_ORYSI,2_L_Q0DEM8_ORYSJ))),2_L_C5Z3W2_SORBI),2_L_I1PZH5_ORYGL),(2_B_G7G841_9GAMM,2_B_F3BIN1_PSEHA)),((((2_B_H6NIV6_9BACL,2_B_F8FKP1_PAEMK),2_B_I0BVD7_9BACL),2_B_C6WE48_ACTMD),(((2_B_A1R1E6_ARTAT,2_B_B8HAN4_ARTCA),2_B_A1R1E5_ARTAT),(((2_B_C9YYE3_STRSW,2_B_D7CI53_STRBB),2_B_D9X0K9_STRVR),(2_B_C6WQV0_ACTMD:,(2_B_D9WXU0_STRVR,(2_B_B5HFS8_STRPR,2_B_F2R4J6_STRVP)))))))
Дерево отчетливо делится на 2 множества - красным выделено множество прокариот,
синим - второе множество, состоящее из эукариот. Если рассматривать множество эукариот, то на дереве не видно четкого разделения между группами Eudicotyledons, Liliopsida .
Часть 3: профиль
Было выбрано подсемейство Bacteria (так как выделялась хорошая клада).
Для построения профиля использовался пакет HMMER. С помощью программы hmm2build, hmm2calibrate был построен профиль.
По этому профилю был проведен поиск по всем белкам, включающим домен PF00544
с помощью программы hmm2search. Результат
После построили Roc-кривую и таблицу (см. ниже).
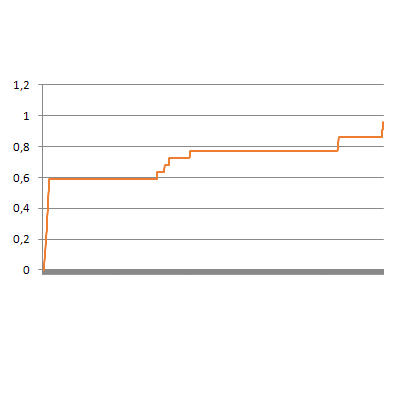
Рис.2. ROC-кривая для выбранного подсемейства. Вычисления(лист Line)
Таблица 1. Данные по профилю для E-value 1.1e-109
| Принадлежит семейству | Не принадлежит семейству | Сумма |
| Выше порога | 14 | 4 | 28 |
| Ниже порога | 9 | 932 | 941 |
| Сумма | 23 | 936 | |