Ферменты и метаболические пути. База данных KEGG.Работа с KEGG PATHWAYВ этом задании мы будем учиться работать с базой данных KEGG. Для этого я выбрала биохимический путь, а именно синтез жирных кислот (R00061). Ссылка на этот синтез Из списка организмов, доступных для рассмотрения в БД KEGG, было выбрано три организма: бактерия - Erwinia billingiae(Erwinia, Gammaproteobacteria, Bacteria), археи - Methanotorris igneus(Methanotorris, Euryarchaeota, Archaea) и эукариот - Branchiostoma floridae ( Lancelets, Vertebrates, Animals). На примере организма ланцетника, я посмотрела какие ферменты в его огранизме(они обозначены зеленым). Такое количество ферментов в данном случае говорит о том, что они не обрзуют цепь последовательных реакций. Таким образом данный организм не способен осуществить такой биохимический путь. Работа с KEGG REACTIONДля выбранного метаболического пути была найдена реакция R01280, в которой принимает участие один из сложных органических кофакторов. На рис1. картинку метаболического пути с данной реакцией, покрашенной в голубой цвет. Рис1. Картинка метаболического пути с данной реакцией, покрашенной в голубой цвет. Изображение реакции с изображениями структурных формул веществ представено ниже. В данной реакции из гексадеканоата получается гексадеканоил-CoA, также в реакции учавствует CoA и АТФ.
Рис2. Изображение реакции с изображениями структурных формул веществ. Работа с KEGG ORTHOLOGYПоскольку для данного метаболического пути не получилось найти реакцию с ортологическими белками, подходящую по всем параметрам, то был взят другой метабалический путь (Деградация жирных кислот) В этом пути была найдена реакция R04741, которую катализирует двое ортологических рядов белков: K07515, K10527. Рис3. Картинка метаболического пути с данной реакцией, покрашенной в зеленый цвет. Далее мы получили файл с последовательностями для каждого ортологического ряда из БД Uniprot. Далее было проведено выравнивание всех последовательностей из всех обнаруженных ортологических рядов с помощью программы Muscle. После чего было получено множественное выравнивание данных последовательностей. Проект программы Jalview. В целом все белки были хороше выравнены, но я удалила несколько, так как их последовательно сильно отличались от других. Это может быть связано с тем, что данные белки давно отделились. Далее было построено филогенетическое дерево для выбранных белков.(Рис.4.) 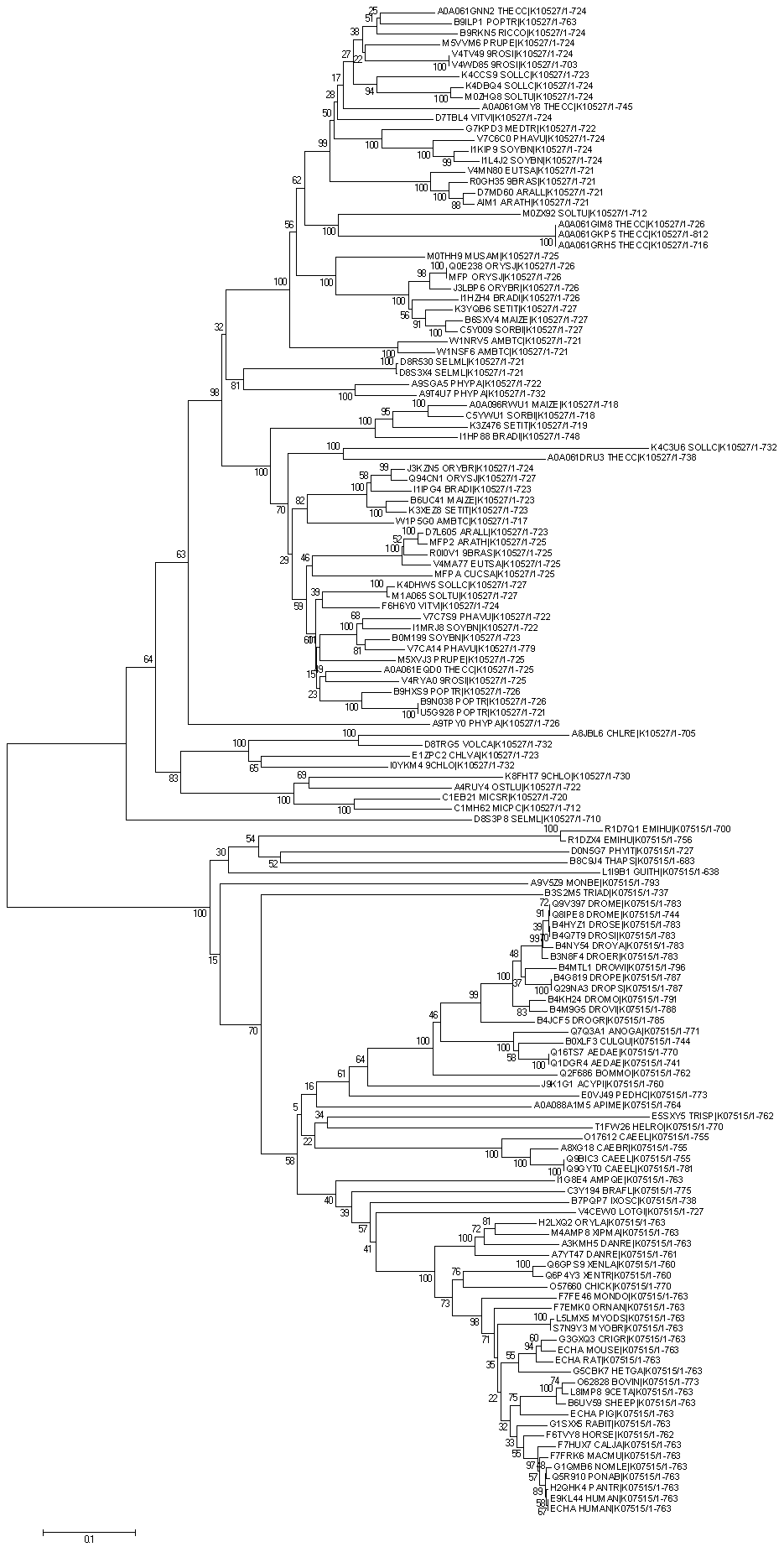 Риc.4 Филогенетическое дерево ортологов Из выравнивания можно сделать вывод, что данные белки являются гомологами, так как по окашиванию данного выравнивания можно предположить, что что встречаются участки(блоки) совпадающих последовательностей, но также происходит небольшое разделение выравнивания на две группы. Этот вывод подтверждается нашим выравниванием. |