Упр.1

Упр.2. Спиральные параметры разных форм ДНК
| A-форма | B-форма | *Z-форма | |
Тип спирали (правая или левая) |
Правая |
Правая |
Левая |
Шаг спирали (A) |
28.03 |
33.75 |
43.5 |
Число оснований на виток |
11 |
10 |
12 |
Ширина большой бороздки |
16,81[T31B.P-C12A.P] |
17.21 [T11A.P-C27B.P] |
9.86 [C16B.P-G9A.P] |
Ширина малой бороздки |
7,98[T31B.P-C4A.P] |
11.69[T.11A.P-A34B.P] |
15.17[C16B.P-G5A.P] |
Упр.3. Сравнение торсионных углов в структурах А- и В-форм.
Торсионные углы для тимина.| alpha | beta | gamma | delta | epsilon | zeta | chi | |
| А-ДНК | 64.11 | 174.79 | 41.72 | 79.08 | -147.87 | -108.23 | 85.22 |
| В-ДНК | 85.87 | 136.38 | 31.10 | 143.42 | -140.77 | -160.52 | -34.35 |
Некоторые значения углов не соответствуют таблице в презентации. Возможно это связано с тем, что я меряла из для тимина или же это погрешность анализа.
Исследование структуры тРНК
1.Краткое описание структуры в файле 1G59_old.pdb
В файле приведены координаты атомов следующих молекул:Для исследования была выбрана цепь B, представляющая глутамил-тРНК со следующей последовательностью:
[1] 5'-GGCCССAUCGUCUAGCGGUUAGGACGCGGCCCUCUCAAGGCCGAAACGGGGGUUCGAUCCCCCUGGGGUCACCA-3' [76],
где 1 и 76 - номера первого и последнего нуклеотида.В последовательности на 3'-конце представлен триплет CCA, к которому присоединяется аминокислота. Приведены координаты его атомов.
2.Исследование вторичной структуры
С помощью программы find_pair пакета 3DNA были определены возможные водородные связи между азотистыми основаниями (1G59_old.out). В соответствии с полученными данными:
| Рис.1. Вторичная структура Глутамил-тРНК из Thermus Thermophilus | Скрипт для получения изображения |
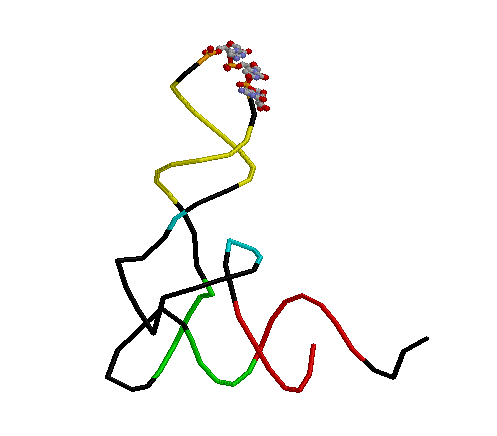
|
background white restrict RNA wireframe off color black backbone 100 select (501-507, 566-572) and RNA color red select (549-553, 561-565) and RNA color green select (510-512, 523-525) and RNA color cyan select (538-544, 526-532) and RNA color yellow select (534, 535, 536) and RNA backbone off wireframe 100 cpk 150 color cpk restrict *:B Даный скрипт служит для получения в RasMol изображения остова исследуемой тРНК, где акцепторный стебель выделен красным, Т-стебель - зеленым, D-стебель - голубым, антикодоновый - желтым. В шарнирной модели представлен антикодон (СUC)к глутамату. |

Замечания:
3.Исследование третичной структуры
A)1. Программа analyze показала предполагаемые стекинг-взаимодействия между парами оснований, связанных водородными связями. Всего 28 возможных стекинг-взаимодействий (28 пар оснований). Из них было выбрано возможное взаимодействие между G7-G49 и C66-U65 нуклеотидами:.

Из изображения видно, что нуклеотиды слабо перекрываются. Числовые данные это подтверждают: площадь перекрывания равна 3.57 квадратных ангстрем. Данное значение относительно невелико (в сравнении с другими возможными стекинг-взаимодействиями). Следовательно, вероятность реального существования этого стекинг-взаимодействия является низкой. Рассмотрим то же возможное стекинг-взаимодействие в RasMol:
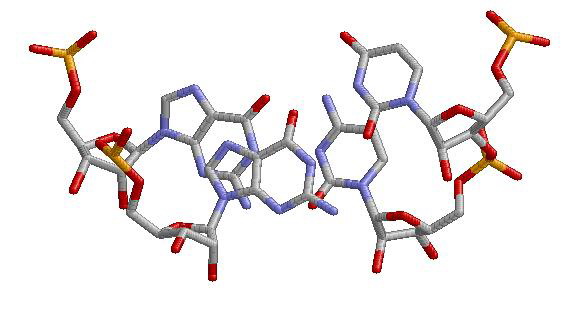
По этому изображению, становится ясно, что перекрываются только два нуклеотида и площадь их перекрывания очень мала.
Дополнительные водородные связи между основаниями D- и Т-петель.
Исходя из представленной выше информации относительно аминокислотных остатков, образующих D- и Т-стебли, разумно утверждать, что
Пользуясь выдачей программы analyze, можно утверждать, что существует два таких взаимодействия: 55U-17G (неканоническое), 18G-56C (каноническое).
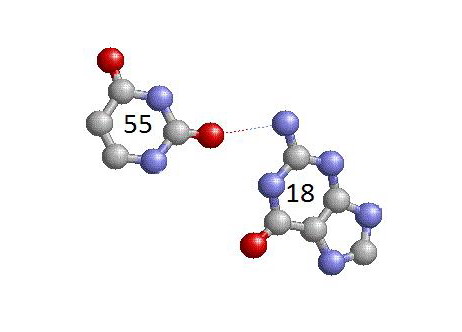
Участок структуры |
Позиции в структуре (по результатам find_pair) |
Результаты предсказания |
Результаты предсказания по алгоритму Зукера |
Акцепторный стебель |
|
|
предсказано 7 пар |
D-стебель |
5' 10-12 3' | Нет предсказаний |
Предсказано 4 пары, включающие 3 верных |
T-стебель |
5' 49-53 3' |
Нет предсказаний |
Предсказаны все 5 пар |
Антикодоновый стебель |
5' 38 - 44 3' |
Предсказано 4 из 7 реальных |
Предсказано 9 пар, включающих в себя 5 верных |
Общее число канонических пар нуклеотидов |
22 |
11 |
20 |
Предсказание EINVERTED
Minimum score threshold=20, так как при больших значениях ничего не было найдено, а при меньших результаты не менялись. Для обнаружения антикодонового стебля пришлось опустить gap penalty=o.Интересно, что программа автоматически заменяет урацил на тимин в последовательности. Заметно также, что предсказание взаимодействий ведётся в предположении, что цепь может перегибаться только в одном месте, ближе к середине. Следовательно, хорошо отыскивается акцепторный стебель и чуть хуже антикодоновый, тогда как отыскать D- или Т-стебель, наверное, невозможно. Это и подтверждается результатами.