Дан список бактерий:
| Название | Мнемоника |
| Bacillus anthracis | BACAN |
| Bacillus subtilis | BACSU |
| Clostridium botulinum | CLOB1 |
| Clostridium tetani | CLOTE |
| Enterococcus faecalis | ENTFA |
| Finegoldia magna | FINM2 |
| Geobacillus kaustophilus | GEOKA |
| Lactobacillus acidophilus | LACAC |
| Lactobacillus delbrueckii | LACDA |
| Lactococcus lactis | LACLM |
| Listeria monocytogenes | LISMO |
| Staphylococcus aureus | STAA1 |
| Staphylococcus epidermidis | STAES |
| Streptococcus pyogenes | STRP1 |
| Streptococcus pneumoniae | STRPN |
И дано их филогенетическое дерево:
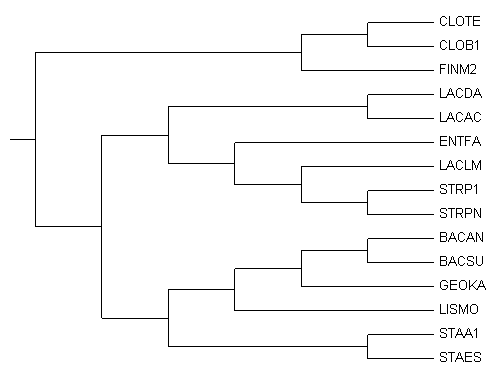
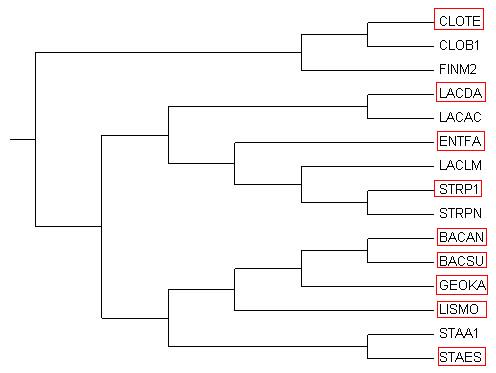
Я выбрала 9 произвольных видов:
CLOTE, LACDA, ENTFA, STRP1, BACAN, BACSU, GEOKA, LISMO, STAES
| Название | Мнемоника |
| Bacillus anthracis | BACAN |
| Bacillus subtilis | BACSU |
| Clostridium tetani | CLOTE |
| Enterococcus faecalis | ENTFA |
| Geobacillus kaustophilus | GEOKA |
| Lactobacillus delbrueckii | LACDA |
| Listeria monocytogenes | LISMO |
| Staphylococcus epidermidis | STAES |
| Streptococcus pyogenes | STRP1 |
Вот эти виды на общем дереве:
Скобочная формула дерева:
(CLOTE, ((LACDA, (ENTFA, STRP1)), (STAES, (LISMO, (GEOKA, (BACAN, BACSU))))));
Изображение дерева:
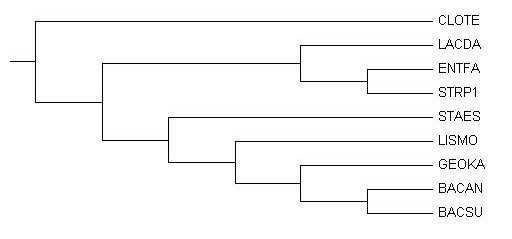
Ветви дерева:
Дерево содержит 6 нетривиальных ветвей:
1)(BACAN, BACSU) против (CLOTE, LACDA, ENTFA, STRP1, STAES, LISMO, GEOKA)
2)(GEOKA, BACAN, BACSU) против (CLOTE, LACDA, ENTFA, STRP1, STAES, LISMO)
3)(LISMO, GEOKA, BACAN, BACSU) против (CLOTE, LACDA, ENTFA, STRP1, STAES)
4)(STAES, LISMO, GEOKA, BACAN, BACSU) против (CLOTE, LACDA, ENTFA, STRP1)
5)(CLOTE, STAES, LISMO, GEOKA, BACAN, BACSU) против (LACDA, ENTFA, STRP1)
6)(CLOTE, LACDA, STAES, LISMO, GEOKA, BACAN, BACSU) против (ENTFA, STRP1)
Таксономия отобранных бактерий:
| Название | Мнемоника | Таксономия |
| Bacillus anthracis | BACAN | Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus cereus group |
| Bacillus subtilis | BACSU | Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus subtilis group |
| Clostridium tetani | CLOTE | Bacteria; Firmicutes; Clostridia; Clostridiales; Clostridiaceae; Clostridium |
| Enterococcus faecalis | ENTFA | Bacteria; Firmicutes; Bacilli; Lactobacillales; Enterococcaceae; Enterococcus |
| Geobacillus kaustophilus | GEOKA | Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Geobacillus |
| Lactobacillus delbrueckii | LACDA | Bacteria; Firmicutes; Bacilli; Lactobacillales; Lactobacillaceae; Lactobacillus |
| Listeria monocytogenes | LISMO | Bacteria; Firmicutes; Bacilli; Bacillales; Listeriaceae; Listeria |
| Staphylococcus epidermidis | STAES | Bacteria; Firmicutes; Bacilli; Bacillales; Staphylococcaceae; Staphylococcus |
| Streptococcus pyogenes | STRP1 | Bacteria; Firmicutes; Bacilli; Lactobacillales; Streptococcaceae; Streptococcus |
Вот ветви, выделяющие таксоны:
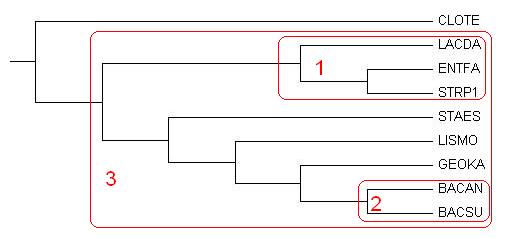
| Ветвь | Таксон | Таксономия | |
| 1 | (LACDA, ENTFA, STRP1) против
(CLOTE, STAES, LISMO, GEOKA, BACAN, BACSU) | Lactobacillales | Bacteria; Firmicutes; Bacilli; Lactobacillales |
| 2 | (BACAN, BACSU)
против (CLOTE, LACDA, ENTFA, STRP1, STAES, LISMO, GEOKA) | Bacillus | Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Bacillus |
| 3 | (LACDA, STAES, LISMO, GEOKA, BACAN, BACSU, ENTFA, STRP1)
против
(CLOTE) | Bacilli | Bacteria; Firmicutes; Bacilli |
Реконструкция филогенетического дерева
Для реконструкции филогенетического дерева был выбран S2(мнемоника RS2)
Из базы данных Swiss-Prot были получены последовательности и выравнены программой muscle.
Выравнивание экспортировано в GeneDoc и подкрашены консервативные позиции внутри таксона:

Диагностические позиции:
| Позиция | Таксон | Аминокислота |
| 28 | Bacilli | K |
| Clostridia | A | |
| 49 | Lactobacillales | M, L(алифатические аминокислоты) |
| 65 | Bacilli | A, G (алифатические аминокислоты) |
| Clostridia | K | |
| 90 | Clostridia | H |
| 108 | Bacilli | Q, S (полярные незаряженые) |
| Clostridia | K | |
| 113 | Bacilli | R, H (полярные с положительным зарядом) |
| Clostridia | T | |
| 115 | Clostridia | E |
| 121 | Bacillus | Q |
| 139 | Bacilli | K |
| Clostridia | L | |
| 141 | Bacillus | L |
| 147 | Bacilli | F |
| Clostridia | N | |
| 153 | Bacilli | D |
| Clostridia | N | |
| 154 | Bacilli | M |
| Clostridia | L | |
| 155 | Lactobacillales | P |
| Clostridia | N | |
| 156 | Clostridia | A |
| 157 | Lactobacillales | R |
| 158 | Lactobacillales | I |
| 159 | Bacilli | P |
| Clostridia | V | |
| 161 | Lactobacillales | V |
| 179 | Bacilli | K |
| Clostridia | I | |
| 197 | Bacilli | I |
| Clostridia | V | |
| 230 | Lactobacillales | D |
| 253 | Lactobacillales | V |
Реконструкция филогенетического дерева программой fprotpars
Для запуска программы fprotpars была использована команда:
fprotpars -sequence RS2_aligned.fasta -outfile RS2.fprotpars
Программа нашла два наиболее "бережливых" дерева:
+--------------------STRP1
+--2
! ! +-----------------ENTFA
! +--3
! ! +--------------STAES
! +--4
! ! +-----------LISMO
! +--5
1 ! +--------BACSU
! +--8
! ! +-----GEOKA
! +--6
! ! +--BACAN
! +--7
! +--CLOTE
!
+-----------------------LACDA
((STRP1,(ENTFA,(STAES,(LISMO,(BACSU,(GEOKA,(BACAN,CLOTE))))))),
LACDA)
+--ENTFA
+-----------------3
! +--STRP1
+--2
! ! +--------------STAES
! +-----4
! ! +-----------LISMO
! +--5
! ! +--------BACSU
1 +--8
! ! +-----GEOKA
! +--6
! ! +--BACAN
! +--7
! +--CLOTE
!
+-----------------------LACDA
(((ENTFA,STRP1),(STAES,(LISMO,(BACSU,(GEOKA,(BACAN,CLOTE)))))),
LACDA)
Оба из представленных деревьев кардинально отличаются от правильного, единственная совпадающая ветвь
(STAES, LISMO, BACSU, GEOKA, BACAN,CLOTE) против (LACDA, ENTFA, STRP1), все остальные противоречат ветвям правильного дерева.
Реконструкция филогенетического дерева программой fprotpars
Программа была запущена командой:
fprotdist -sequence RS12.muscle.fasta -outfile RS12.fprotdist
Выдача программы: RS2.fprotdist
LACDA CLOTE STRP1 ENTFA STAES LISMO GEOKA BACAN BACSU
LACDA 0.000000 0.486860 0.435955 0.391741 0.536234 0.512328 0.446016 0.419908 0.450846
CLOTE 0.486860 0.000000 0.459729 0.422162 0.401865 0.360443 0.340334 0.352468 0.384315
STRP1 0.435955 0.459729 0.000000 0.278163 0.561312 0.400944 0.375102 0.370008 0.467618
ENTFA 0.391741 0.422162 0.278163 0.000000 0.443135 0.318550 0.337937 0.295251 0.332919
STAES 0.536234 0.401865 0.561312 0.443135 0.000000 0.353706 0.222982 0.255914 0.282383
LISMO 0.512328 0.360443 0.400944 0.318550 0.353706 0.000000 0.220487 0.184069 0.246379
GEOKA 0.446016 0.340334 0.375102 0.337937 0.222982 0.220487 0.000000 0.169656 0.122655
BACAN 0.419908 0.352468 0.370008 0.295251 0.255914 0.184069 0.169656 0.000000 0.129099
BACSU 0.450846 0.384315 0.467618 0.332919 0.282383 0.246379 0.122655 0.129099 0.000000
Согласно утверждению ультраметричности: "из трех расстояний между тремя объектами два всегда равны между собой и не меньше третьего".
Оценим, насколько расстояния отклоняются от ультраметричности:
-
Рассмотрим СLOTE, STRP1 и BACSU - все из далеких групп
d(CLOTE, STRP1) = 0.459729
d(STRP1, BACSU) = 0.467618
d(CLOTE, BACSU) = 0.384315
Наиболее близкие расстояния (0.459729 и 0.467618) больше третьего (0.302540).
Эта тройка не удовлетворяет аксиоме ультраметричности, так как не имеет равных значений. -
Рассмотрим BACAN, BACSU и GEOCA - первые две - наиболее близкие
d(BACAN, BACSU) = 0.129099
d(BACSU, GEOCA) = 0.122655
d(BACAN, GEOCA) = 0.169656
Наиболее близкие расстояния (0.129099 и 0.122655) меньше третьего (0.169656).
Эта тройка не удовлетворяет аксиоме ультраметричности.
Согласно принципу аддитивности: если есть 4 последовательности: A, B, C, D, -
то из трех сумм d(A,B) + d(C,D); d(A,C) + d(B,D); d(A,D) + d(B,C)
две равны между собой и больше третьей.
Оценим, насколько расстояния отклоняются от аддитивности:
-
Рассмотрим CLOTE, LACDA, GEOKA и BACSU
d(CLOTE, LACDA) + d(GEOKA, BACSU) = 0.486860 + 0.122655 = 0.609515
d(CLOTE, GEOKA) + d(LACDA, BACSU) = 0.340334 + 0.450846 = 0.791180
d(CLOTE, BACSU) + d(LACDA, GEOKA) = 0.384315 + 0.446016 = 0.830331
Эти последовательности не удовлетворяют аддитивности.
Реконструкция филогенетического дерева программой fneighbor, с использованием алгоритмов UPGMA и Neighbor-Joining
С помощью программы fneighbor по матрице расстояний были постороены два дерева ( по алгоритмам UPGMA и Neighbor-Joining).
Соответствующие команды для запуска:
Neighbor-Joining:
fneighbor -datafile RS12.fprotdist -outfile RS2.Neighbor-Joining.fneighbor
-outtreefile RS2.Neighbor-Joining.fneighbor.tree
UPGMA:
fneighbor -datafile RS2.fprotdist -outfile RS12.UPGMA.fneighbor
-outtreefile RS2.UPGMA.fneighbor.tree -treetype u
Neighbor-Joining:
+---------STRP1
+-1
! +-----ENTFA
!
! +-----------CLOTE
! !
2--3 +------LISMO
! ! !
! +--4 +--BACAN
! ! !
! +-6 +---------STAES
! ! +-5
! +-7 +--GEOKA
! !
! +----BACSU
!
+--------------LACDA
((STRP1:0.17687,ENTFA:0.10130):0.02952,(CLOTE:0.20006,(LISMO:0.12462,
(BACAN:0.05332,((STAES:0.17107,GEOKA:0.05191):0.01712,BACSU:0.07391):0.01636):0.02178):0.04167):0.05692,LACDA:0.24525);
UPGMA:
+-------------LACDA
!
! +----------CLOTE
! !
--8 +-6 +--------STAES
! ! ! !
! ! +-5 +------LISMO
! ! ! !
! ! +-3 +---GEOKA
+-7 ! +-1
! +-2 +---BACSU
! !
! +----BACAN
!
! +--------STRP1
+--4
+--------ENTFA
(LACDA:0.22999,((CLOTE:0.18394,(STAES:0.13937,(LISMO:0.10849,
((GEOKA:0.06133,BACSU:0.06133):0.01336,BACAN:0.07469):0.03380):0.03088):0.04457):0.01542,
(STRP1:0.13908,ENTFA:0.13908):0.06028):0.03063);
UPGMA отличается от Neighbor-Joining только двумя ветвями, в случае UPGMA это
(GEOKA, BACSU) против (LACDA, CLOTE, STAES, LISMO, BACAN, STRP1, ENTFA) и
(GEOKA, BACSU, BACAN) против (LACDA, CLOTE, STEES, LISMO, STRP1, ENTFA),
а в случае Neighbor-Joining это
(GEOKA, STAES) против (LACDA, CLOTE, BACSU, LISMO, BACAN, STRP1, ENTFA) и
(GEOKA, STAES, BACSU) против (LACDA, CLOTE, LISMO, BACAN, STRP1, ENTFA)
И Neighbor-Joining, и UPGMA имеют с fprotpars только 1 общую ветвь:
(LACDA, STRP1, ENTFA) против (GEOKA, STAES, BACSU, CLOTE, LISMO, BACAN)
Neighbor-Joining отличается от правильного дерева двумя ветвями:
(GEOKA, STAES) против (LACDA, CLOTE, BACSU, LISMO, BACAN, STRP1, ENTFA) и
(GEOKA, STAES, BACSU) против (LACDA, CLOTE, LISMO, BACAN, STRP1, ENTFA),
у правильного дерева
(BACAN, BACSU) против (CLOTE, LACDA, ENTFA, STRP1, STAES, LISMO, GEOKA) и
(GEOKA, BACAN, BACSU) против (CLOTE, LACDA, ENTFA, STRP1, STAES, LISMO) соответственно.
UPGMA отличается от правильного дерева только на 1 ветвь:
(GEOKA, BACSU) против (LACDA, CLOTE, STAES, LISMO, BACAN, STRP1, ENTFA),
у правильного дерева здесь
(BACAN, BACSU) против (CLOTE, LACDA, ENTFA, STRP1, STAES, LISMO, GEOKA)
Таким образом, наиболее близко к правильному дереву получается построение с помощью UPGMA, а наиболее далеко fprotpars.