Укоренение в среднюю точку
Дерево, построенное методом Neighbor-Joining:
+---------STRP1
+-1
! +-----ENTFA
!
! +-----------CLOTE
! !
2--3 +------LISMO
! ! !
! +--4 +--BACAN
! ! !
! +-6 +---------STAES
! ! +-5
! +-7 +--GEOKA
! !
! +----BACSU
!
+--------------LACDA
((STRP1:0.17687,ENTFA:0.10130):0.02952,(CLOTE:0.20006,(LISMO:0.12462,
(BACAN:0.05332,((STAES:0.17107,GEOKA:0.05191):0.01712,BACSU:0.07391):0.01636):0.02178):0.04167):0.05692,LACDA:0.24525);
Скобочная формула была копирована в файл с именем intree. Был запущен файл retree.exe. Программа выдала следующее сообщение:
Tree Rearrangement, version 3.69 Settings for this run: U Initial tree (arbitrary, user, specify)? User tree from tree file N Format to write out trees (PHYLIP, Nexus, XML)? PHYLIP 0 Graphics type (IBM PC, ANSI, none)? IBM PC W Width of terminal screen, of plotting area? 80, 80 L Number of lines on screen? 24 Are these settings correct? (type Y or the letter for one to change)
Сначала нажала кнопку Y.
Программа изобразила некое укоренение:
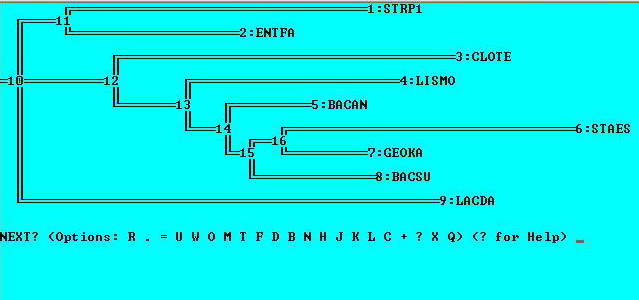
Нажала М, и программа укоренила дерево:
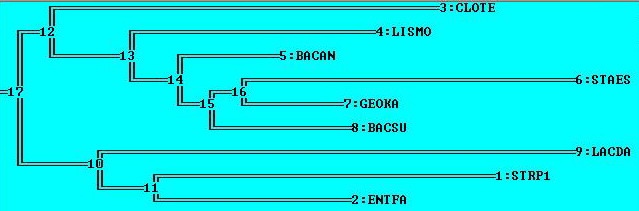
Укоренение произошло в ветвь (CLOTE, LISMO, BACAN, STAES, GEOCA, BACSU) против (LACDA, STRP1, ENTFA)
Но данное укоренение не соответствует правильному дереву, а дерево UPGMA укоренять не нужно, оно уже укорененное. Деревья, построенные методом максимальной
бережливости не предполагают существование молекулярных часов и не выдает длины ветвей, поэтому
их и нельзя укоренить.
Использование внешней группы
В качестве внешней группы использовался белок того же семейства (RS2) из сенной палочки. В файл формата fasta
с последовательностями выбранных бактерий была добавлена пследовательность данного белка из Bacillus subtilis.
Полученный файл был выравнен программой muscle.

На первый взгляд последовательность белка S2 сенной палочки от остальных существенно не отличается.
Но если посмотреть внимательнее, можно заметить, что белок то и дело отличается от остальных по конкретным точечным позициям.
По полученному выравниванию был запущен fprotpars.
Protein parsimony algorithm, version 3.69
One most parsimonious tree found:
+-----------------------RS2_CLOTE
!
+--2 +--------------------RS2_GEOKA
! ! !
! +--7 +-----------------RS2_BACAN
! ! !
! +--8 +--------------RS2_BACSU
! ! !
! +--9 +-----------RS2_LISMO
! ! !
1 +--6 +--------RS2_STAES
! ! !
! +--5 +--RS2_ENTFA
! ! +--4
! +--3 +--RS2_STRP1
! !
! +-----RS2_LACDA
!
+--------------------------RS2_ECOLI
Уберем внешнюю группу ECOLI и получим укорененное дерево:
(CLOTE,(GEOKA,(BACAN,(BACSU,(LISMO,(STAES,((ENTFA,STRP1),LACDA)))))))
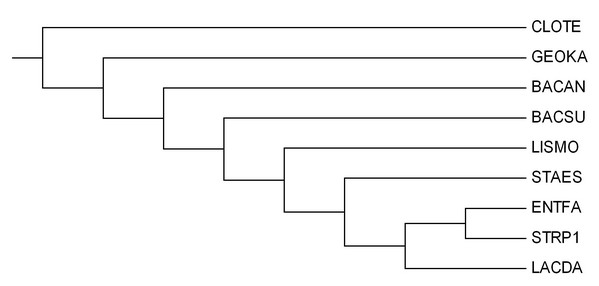
Оно имеет 2 общие ветви с правильным:
(CLOTE, LACDA, STAES, LISMO, GEOKA, BACAN, BACSU) против (ENTFA, STRP1)
(CLOTE, STAES, LISMO, GEOKA, BACAN, BACSU) против (LACDA, ENTFA, STRP1)
Бутстрэп
Сперва подала выравнивание последовательностей на вход fseqboot.
Получила файл с бутстрэп-репликами.
Данный файл подала на вход fprotpars.
Получила изображения и
скобочные формулы.
Файл с изображениями деревьев был подан на вход программе fconsense
для создания единого дерева по принципу "расширенного большинства" (extended majority rule tree).
Выдача программы:
скобочная формула и
дерево по принципу "расширенного большинства".
+---------------------------------------------------------------STRP1
|
| +-------CLOTE
| +--84.0-|
| +--100.0-| +-------BACAN
| | |
| +--97.5-| +---------------GEOKA
| | |
| +--86.2-| +-----------------------BACSU
| | |
| +--93.0-| +-------------------------------LISMO
| | |
| +--51.3-| +---------------------------------------STAES
| | |
+-------| +-----------------------------------------------ENTFA
|
+-------------------------------------------------------LACDA
remember: this is an unrooted tree!
Несмотря на большую поддержку всех ветвей с правильным деревом совпала только 1 ветвь:
(CLOTE, STAES, LISMO, GEOKA, BACAN, BACSU) против (LACDA, ENTFA, STRP1).
Зато дерево полностью совпало с деревом, предсложенным fprotpars в прошлой практической работе.
Информация о ветвях:
Species in order: 1. STRP1 2. ENTFA 3. STAES 4. LISMO 5. BACSU 6. GEOKA 7. BACAN 8. CLOTE 9. LACDA Sets included in the consensus tree Set (species in order) How many times out of 100.00 .....***. 100.00 ....****. 97.50 ..******. 93.00 ...*****. 86.17 ......**. 84.00 .*******. 51.33 Sets NOT included in consensus tree: Set (species in order) How many times out of 100.00 ..******* 48.17 .....*.*. 16.00 ..*.****. 13.83 ..*.....* 4.50 ...*.***. 2.50 .*.*****. 1.50 .**...... 1.00 ..*.***** 0.50
Среди невключенных ветвей правильных тоже нет.
Таким образом, ни один из методов не смог дать нам правильного дерева. Отрицательный результат - тоже результат. Возможно, подобное можно объяснить, во-первых, небольшой длиной выбранного белка (в выравнивании трудно выявить большие отличия), а, во-вторых, горизонтальным переносом генов.