Поиск и визуализация гомологов белка
Задача - найти гомологи белка CLPX_ECOLI в протеомах бактерий, которые были выбраны ранее. Для этого я скопировала протеомы в рабочую папку, объединила их в один файл, проиндексировала его и провела поиск программой blast.
cat * > all_proteoms.fasta
makeblastdb -in all_proteoms.fasta -dbtype prot
blastp -task blastp -query CLPX_ECOLI.fasta -db all_proteoms.fasta -out blastp_out.txt -outfmt 6 -evalue 0.001
Из файлов с протеомами был получен файл с последовательностями белков.
При построении дерева использовались параметры:
- Алгоритм выравнивания - MAFFT
- Алгоритм построения дерева - FastME
- "Gamma distributed rates across sites" — No
- "Starting tree" — BIONJ
- "No refinement"
- Число бутстреп реплик - 100
Дерево было укоренено в среднюю точку.
Примеры ортологов: A1AZV8 PARDP - Q12Q18 SHEDO, HSLU SHEDO - HSLU ECOLI, CPLX PARDP - CPLX ROSDO
Примеры паралогов: A1AZV8 PARDP - HSLU PARDP, HSLU HAEIN - CPLX HAEIN, FTSH ECOLI - CPLX ECOLI
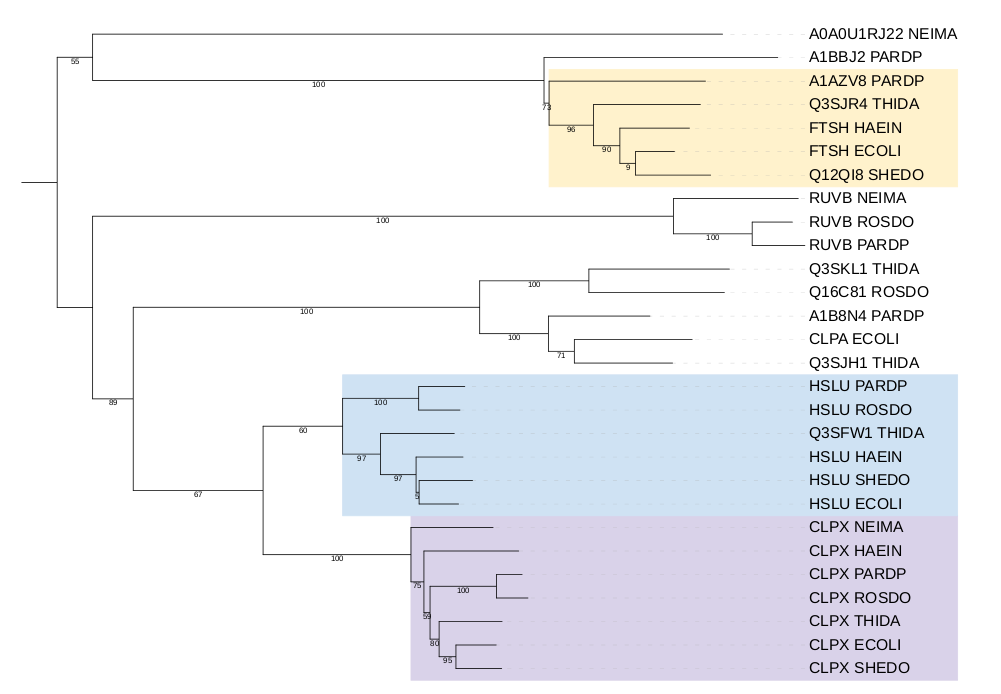
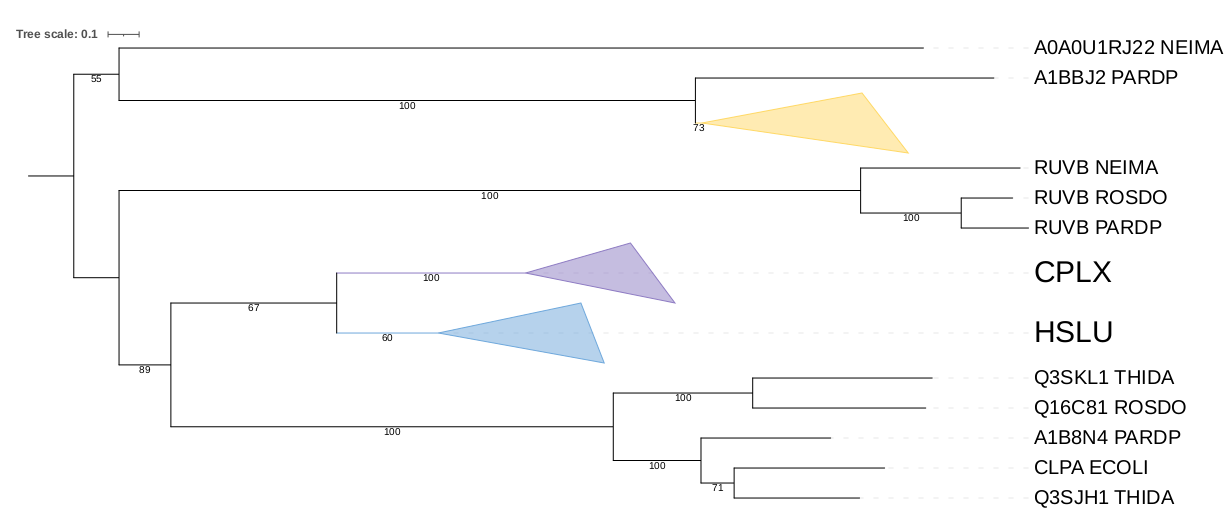
В группу CPLX вошли 7 белков, HSLU - 6, а в группу с белками с разными названиями - 5. Все деревья внутри них близки к филогении, но нигде не выделилась клада THIDA-HAEIN.