Занятие 4
Построение дерева по нуклеотидным последовательностям
Видовое дерево
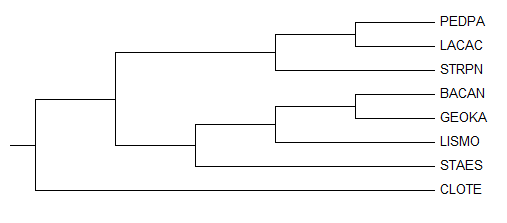
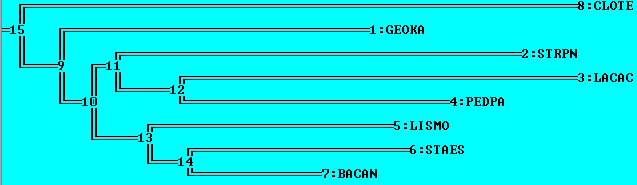
Дерево, полученное по 16S rRNA последовательностям методом Neighbor-Joining
Выравнивание
(CLOTE:0.11782,(GEOKA:0.06561,((STRPN:0.08509,(LACAC:0.08417,PEDPA:0.05754):0.01284):0.00537,
(LISMO:0.05201,(STAES:0.04659,BACAN:0.02756):0.00933):0.01133):0.00783):0.0076);
Дерево было укоренено в среднюю точку в правильную ветвь {CLOTE} против {PEDPA, LACAC, STRPN, LISMO, GEOKA, BACAN, STAES}.
В этом дереве имеется верная ветвь {PEDPA, LACAC}:{остальные листья}, которая отсутствовала во всех прошлых деревьях (см. практикум 3).
Т.о. 16S рнк показали верное отношение этих видов к другим, в отличие от белка EF-Ts.
Однако, в остальном дерево получилось хуже, чем при реконструкции методом N-J по белкам (4 из 5 ветвей) - только 2 нетривиальных ветви из правильного дерева присутствуют. Укоренение прошло верно.
Построение и анализ дерева, содержащего паралоги
Дерево гомологов CLPX_BACSU в данных организмах, полученное методом Neighbor-Joining
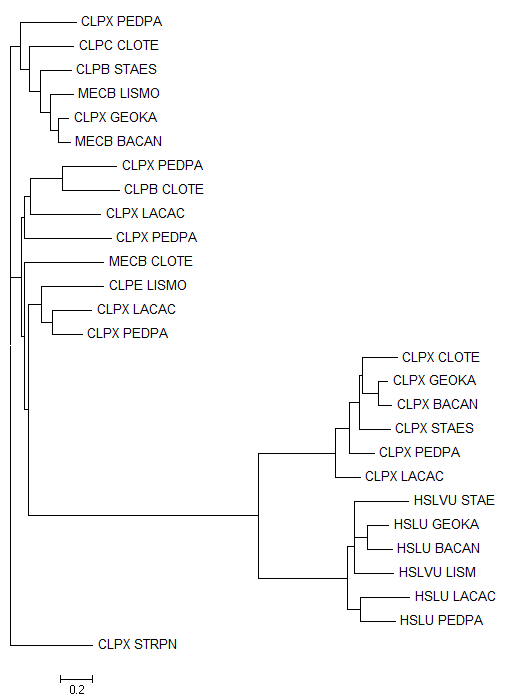
В ходе наименования белков выяснилось, что не всем белкам можно однозначно определить их функцию (CLPX, CLPB, CLPC или др.), ввиду их аннотации.
Однако, HSLU (она же CLPY) субъединица оказалось выделена четко на дереве.
Ортологи:
HSLU_GEOKA (YP_147067.1) - HSLVU_LISMO (EFR84790.1)
CLPX_CLOTE (NP_782910.1) - CLPX_LACAC (YP_193739.1)
Паралоги: CLPX_BACAN (NP_846917.1) - MECB_BACAN (NP_842649.1)
CLPX_LACAC (YP_193739.1) - CLPX_LACAC (YP_193548.1)
CLPX_CLOTE (NP_782910.1) - CLPB_CLOTE (NP_781219.1)