| Главная страница | Обучение | Обо мне | Ссылки | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
BLAST | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Задание 1. В программе BLAST был проведен поиск гомологов белка YP_005259411 (антранилат-фосфорибозилтрансфераза археи Pyrobaculum oguniense). Ссылка на поисковый запрос в БД Swissprot Ссылка на поисковый запрос в БД RefSeq В банке RefSeq было найдено 12066 последовательностей (при выставленном максимуме 20000, т.е. это все). По данному банку не удалось выяснить, сколько последовательностей принадлежит различным таксонам, поэтому я посмотрела также находки по БД Swissprot. В этой базе данных было найдено 557 последовательностей, из них 80 относятся к таксону Archaea, 470 - Eubacteria. Для Eucariotae выяснить не удалось, но, предположительно, 7 последовательностей. В Таблице 1 представлены данные по нескольким находкам в базе данных RefSeq, представленным BLAST-ом. Третье выравнивание (следующее за лучшим) прописано в таблице для того, чтобы показать минимальное E-value. Таблица 1. Данные о находках в БД RefSeq
Для дальнейшей работы использовались результаты поиска BLAST по базе данных SwissProt. Так, в этой БД было найдено 557 последовательностей. Данные, аналогичные полученным по RefSeq, представлены в Таблице 2. Таблица 2. Данные о находках в БД SwissProt
При выборе гомологов белка использовался следующий критерий: query cover > 65% и E-value < 0.001. Отхождение от параметров, поставленных в задании, обусловлено тем, что у первой же последовательности с query cover ниже 70% тем не менее очень низкое E-value.
Поэтому я отделила гомологов несколько иначе (см. Рис. 1): я выбрала место, где E-valuе у следующей находки вырастает в 2 раза (предварительно отсортировав по Query cover). Там же видно, что выше идут антранилат-фосфорибозилтрансферазы бактерий и архей (последняя находка над линией - белок хлоропласта), а ниже линии, проведенной мной, начинаются белки эукариот, к тому же имеющие другое название.
Получилось 493 гомолога. Задание 2. Ссылка на поисковый запрос по таксону в БД SwissProt Был проведен поиск по таксону Archaea. Результаты получились иными, нежели в поиске по всем таксонам: среди архей было найдено 106 последовательностей, тогда как среди всех таксонов нашлось лишь 80. Впрочем, примерно 25 последовательностей имеют E-value больше 0.001, а значит, гомологами их назвать нельзя. Данные о выравниваниях с одной и той же последовательностью из двух разных запросов в Таблице 3. Score не изменился (так как использовалась одна и та же матрица и одни и те же 2 последовательности). Изменилось E-value: оно немного уменьшилось вследствие сужения поля поиска (т.е. размеров банка). Таблица 3. Сравнение выравниваний при разных поисковых запросах
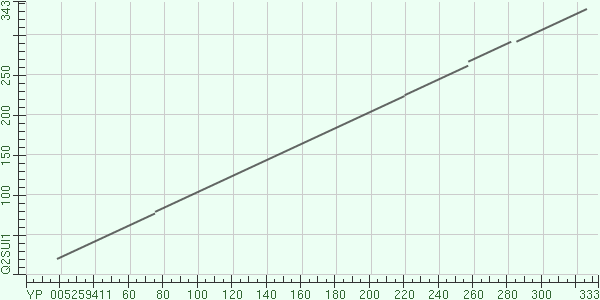
Задание 3. Карта локального сходства двух последовательностей: исходной (YP_005259411) и выбранной из списка найденных (идентификатор в Swissprot Q2SUI1 представлена на Рис.2.
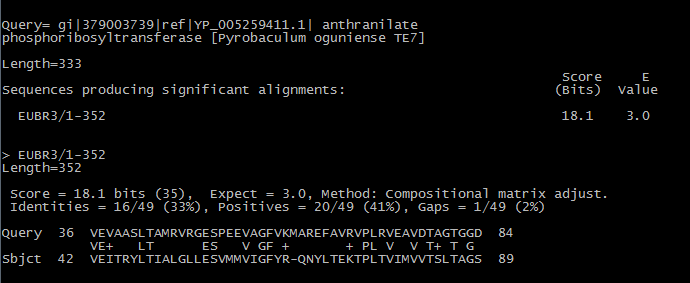
По этой карте видно, что выравнивание покрывает практически всю заданную последовательность (92% query cover). Прерывистость линии обозначает гэповые позиции в парном выравнивании. Гэп около 75 позиции (примерно) маленький, а около 260-ой и 285-ой позиций гэповый участок немного больше (разрыв линии больше). Задание 4. База данных database была создана на основе множественного выравнивания align_09.fasta. в результате поиска в ней гомологов моего белка YP_005259411 получена одна находка: участок выравнивания с последовательностью EUBR3 (см. Рис.3).
участок довольно маленький (49 позиций, то есть примерно 15% общей длины заданной последовательности).
Его характеристики представлены в Таблице 4. Таблица 4. Данные о находке в БД align_09.fasta
Приведенные в Таблице 4 параметры ясно дают понять, что выбранная из базы данных последовательностей не является гомологом моего белка: слишком высокий E-value (3.0), низкий bit score - всего 18.1, для сравнения - у худшей находки в БД SwissProt почти в два раза больше. Количество идентичных и сходных колонок, конечно, велико, но лишь относительно длины выровненного участка. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| © Alexandra Boyko, 2014. Faculty of Bioengineering and Bioinformatics, MSU. | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||