| Главная страница | Обучение | Обо мне | Ссылки | ||||||||||||||||||||||||||||||||||||||||||||||||||||||
PSI-BLAST. Множественное выравнивание. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
Практикум представлен в виде Jal-view проекта. Задание 1. В Таблице 1 представлена информация об итерациях PSI-BLAST, проведенных для составления семейства гомологов белка антранилат-фосфорибозилтрансферазы археи Pyrobaculum oguniense. В результате поиска со стандартными параметрами порога E-value в первой итерации после длинного списка антранилат-фосфорибозилтрансфераз шли белки, вряд ли попадающие в искомое семейство (AMP-phosphorylase, thymidine phosphorylase и др.), поэтому был выставлен порог 1е-20, разделяющий эти белки и антранилат-фосфорибозилтрансферазы. Таблица 1. Данные об итерациях PSI-BLAST
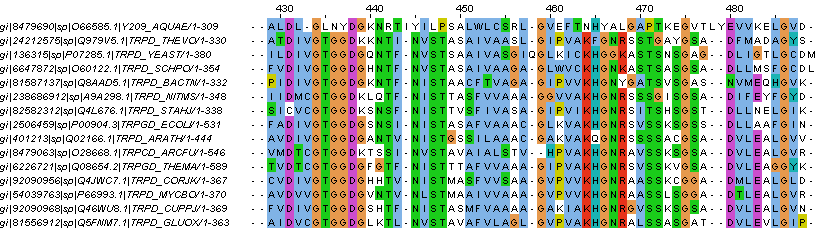
Во второй итерации к предыдущим находкам добавилась одна: неохарактеризованный белок c идентификатором O66585 (его параметры см. в Таблице 1). Он был включен в дальнейшие итерации, так как у него достаточно велик query cover (92%) и E-vаluе в принципе достаточно мал, чтобы быть достоверным. В общем-то, результаты стабилизировались уже на третьей итерации. Задание 3. Было построено множественное выравнивание семейства с помощью программы muscle по запросу "muscle -in family.fasta -out align_family.fasta". Результат представлен в Jal-view проекте, вкладка align_family_muscle. Задание 4. Seed семейства антранилат-фосфорибозилтрансфераз было выбрано с помощью функции Remove Redundancy с порогом 76%, так как только при таком пороге удалось выделить подходящее число последовательностей. Участок seed'а показан на Рис.1 (без выравнивания). В seed вошло 15 белков.
Выравнивание seed'a программой muscle представлено в Jal-view проекте во вкладке align_seed_muscle. Задание 5. Второе выравнивание seed'a было построено с помощью программы mafft на сервере kodomo (запрос mafft seed.fasta > seed_mafft.fasta). Оно представлено в Jal-view проекте во вкладке align_seed_mafft. Задание 6. Выравнивание двух предыдущих выравниваний относительно друг друга было построено с помощью программы muscle: "muscle -profile -in1 seed_mafft.fasta -in2 seed_muscle.fasta -out both.fasta". В jar-проекте вкладка align_seed_muscle&mafft. В строке Identical Columns "+" отмечены стоблцы, совпадающие в обоих выравниваниях. Можно отметить, что в этом выравнивании, по-видимому, программа muscle стремиться сопоставить аминокислоты блоками, то есть так, что получается крупный "блок", внутри которого два выравнивания совпадают (участок самого крупного "блока" показан на рис. 2). Такие "блоки" перемежаются с фрагментами, в которых произошло смещение одного выравнивания на одну позицию.
Этот сдвиг возник в результате того, что программа вставила в нижнее выравнивание (сделанное программой muscle), гэпы в первую позицию. Меня это удивило, потому что в начале выравнивания очевидно, что если бы не было этого гэпа, выравнивания бы сошлись. Однако при избавлении от этой колонки гэпов теряется такая сильная схожесть в "блоках".
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| © Alexandra Boyko, 2014. Faculty of Bioengineering and Bioinformatics, MSU. | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||